用PCR扩增和克隆鸡传染性贫血病毒全基因组DNA
作者:陈为民 崔治中 段玉友 刘岳龙
单位:扬州大学畜牧兽医学院动医系,扬州225009
关键词:鸡传染性贫血病毒;基因组DNA;聚合酶链式反应;克隆
中国病毒学990115 摘 要 根据已发表的序列,分别在鸡贫血病毒(CAV)环形基因组DNA(全长2.3kb)的EcoRI位点和BamHI位点的两侧选择适当序列合成两对引物,用PCR技术,从斑点杂交检测到病毒核酸的CAV感染的MDCC-RP1细胞基因组DNA中,分别扩增出包含EcoRI和BamHI分割开的病毒基因组两部分(1.5kb和0.8kb)约1.5kb和约1.25kb的两个片段。再将其中相应序列拼接克隆进pUC18载体,获得包含CAV全基因组序列DNA片段的克隆质粒pCAV2.4。酶切分析表明,该质粒具有预期的BamHI位点、PstI位点、HindⅢ位点,而预期的EcoRI位点消失。重组质粒插入DNA片段的两端序列分析表明,质粒pCAV2.4是包含CAV全基因组序列的重组质粒,插入DNA片段序列中的EcoRI位点序列发生了一个碱基突变。
, 百拇医药
Amplification and Cloning of Chicken Anemia Virus whole
genomic DNA by Polymerase Chain Reaction
Chen Weimin Cui Zhizhong Duang Yuyou et al
(College of Animal Science and Veterinary Medicine, Yangzhou University, Yangzhou 225009)
Abstract By two pairs of primers complementory to the published sequences at the both sides of EcoRI site and BamHI site on CAV circular genome, two DNA fragments containing two parts (1.5 kb and 0.8 kb) of CAV genome divided by EcoRI and BamHI sites were amplified by PCR from the circular CAV genome (2.3 kb) detected by dot blot in genomic DNA of CAV-infected RP1 cells. Through re-ligation of the selected sequences from the amplified fragments, the whole CAV genomic DNA was cloned into pUC18, and the recombinant plasmid was designated as pCAV2.4. Restriction endonucleases analysis of pCAV2.4 showed that there were sites of BamHI、PstI、HindⅢ,but no site of EcoRI. Two ends of cloned DNA sequence analysis showed that the recombinant plasmid pCAV2.4 containes the whole genomic DNA sequence of CAV, and that the site sequence of EcoRI in the cloned DNA was changed by one base substitution.
, 百拇医药
Key words Chicken anaemia virus, Genome, PCR, Clone
自Yuasa等[1]报道鸡贫血病毒(CAV)以来,世界各地均陆续证明了该病毒感染的普遍性。CAV属于一个新的病毒科——圆环病毒科[2]。这是一种无囊膜的呈二十面体的小病毒,直径只有20nm左右。CAV的基因组是一条只有2.3kb的单股环状“-”链DNA[3],只产生一个转录子,可编码三个蛋白质,即衣壳蛋白VP1及其相关蛋白VP2、细胞凋亡因子VP3[4]。CAV的致病性在于通过CAV编码产生的细胞凋亡因子造成的细胞程序性死亡[5],主要是造血细胞和淋巴细胞的程序性死亡,进而造成感染鸡的贫血,出血及免疫抑制。在野外,感染CAV几乎不引起典型的临床症状,但由于CAV的免疫抑制作用,CAV感染群易并发或继发其它疾病,以及常引起疫苗免疫失败等情况。CAV的亚临床感染严重影响着养禽业的经济效益[6],然而至今仍没有理想的预防CAV感染的方法。国外将CAV基因组双拷贝串连在一起克隆进载体质粒后,所得到的重组质粒DNA转染MDCC-MSB1细胞后可产生传染性的CAV[7]。这就有可能用基因工程的方法构建出能在体内复制的无致病性CAV毒株。为了尝试构建这样的毒株用作疫苗株,本研究用PCR技术成功地扩增和克隆了鸡贫血病毒全基因组DNA。
, http://www.100md.com
1 材料与方法
1.1 材料
1.1.1 引物 引物1:5′CATGATGAATTCCGAGTGGTTACTA3′
引物2:5′CATGATGGATCCCTCATTCTTAGTG3′
引物3:5′CATGATCGCCAAGATCTCTGTGAACCTG3′
引物4:5′CATGATGCATGCGAGCGAAGTCGAGCGATTCGTCCA3′
引物1和引物2分别对应于Noterborn等[10]发表的CAV环形基因组DNA序列的1~19及互补链的1512~1494序列,用于扩增CAV环形基因组1~1512间1512bp DNA片段。两引物5′端分别具有EcoRI、BamHI酶切位点序列。
, http://www.100md.com
引物3和引物4分别对应于Noteborn等[10]发表的环形基因组DNA序列的1158~1179及互补链的82~58序列,用于扩增CAV环形基因组1158~82间的DNA片段。引物4的5′端人为添加有SphI酶切位点序列。
1.1.2 细胞及病毒
MDCC-RP1细胞系,由本教研室提供;CAVCux-1株,由中国兽医监察所惠赠。
1.1.3 菌株及质粒
大肠杆菌TG1用作质粒转化的受体菌,pUC18质粒用作克隆的载体。二者皆由本教研室保存提供。
1.1.4 分子生物学试剂
Dig DNA labeling and Detection Kit、PCR Kit、连接酶及各种限制性内切酶等均购自商业化公司,CAV VP1基因质粒[9]由本教研室提供。
, 百拇医药
1.2 方法
1.2.1 CAV感染MDCC-RP1基因组DNA的制备
以含10%胎牛血清的RPMI-1640培养液培养MDCC-RP1,CAV Cux-1株感染后,每48h自传一次,分别收集第7、14、19、26代细胞和未接种病毒的RP1细胞,按常规[7,8]提取细胞基因组DNA。
1.2.2 斑点杂交比较CAV感染的MDCC-RP1各代细胞中的病毒核酸的水平
对提取的各代细胞基因组DNA浓度定量后,以一系列已知浓度梯度的CAV VP1基因克隆DNA作为参考,用狄高辛标记的CAV VP1基因作为探针。在斑点杂交中检测和比较CAV感染后各代细胞中的CAV DNA水平。
1.2.3 PCR扩增
, http://www.100md.com
引物1和引物2,引物3和引物4组成两对上下游引物,分别用于扩增包含CAV环形基因组EcoRI和BamHI分割开两部分的两DNA片段,1.5kb及1.25kb片段。其PCR扩增步骤基本一致,即:100μL反应体积中,模板DNA 46ng,引物浓度各为2.5μmol/L,4种dNTP浓度为200μmol/L,10×buffer组成为100mmol/L Tris(pH8.3),500mmol/L KCL,15mmol/L MgCl2。反应混合液98℃变性10min,在室温下退火的同时迅速加入1μL Taq DNA聚合酶,覆盖液体石蜡油100μL,离心数秒,进入PCR循环: 94℃ 60s-55℃ 60s-72℃ 180s,共30个循环,最后一个循环后72℃再延伸10min。取PCR产物用1%琼脂糖凝胶电泳,以λDNA/HindⅢ消化物为标记物,观察扩增片段长度。
1.2.4 1.5kb PCR产物的克隆及重组质粒的酶切分析
将已纯化的1.5kb PCR产物与载体pUC18分别用EcoRI、BamHI双酶消化,低熔点琼脂糖电泳回收后,按常规[8]连接、回收。挑取若干单个白斑,进行质粒扩增、提纯,以EcoRI、BamHI双酶切筛选出重组克隆。对重组质粒分别用EcoRI+BamHI、HindⅢ、PstI等内切酶进行酶切,验证插入片段内部酶切位点是否符合预期的结果。
, http://www.100md.com
1.2.5 两PCR扩增片段相应部分的拼接克隆及重组质粒的酶切分析
将已纯化的1.25kb PCR产物与获得的1.5kb PCR产物的重组质粒分别用BamHI、SphI酶消化后,低熔点琼脂糖电泳后,分别回收0.9kb、4.2kb大小的片段,按常规[8]连接转化。挑取若干单个白斑,分别进行质粒扩增、提取,酶切分析,筛选出包含2.4kb插入片段的重组质粒。对筛选到的重组质粒进一步用EcoRI、BamHI、PstI酶切分析。
1.2.6 包含CAV全基因组的重组质粒中插入片段的两段序列分析
采用双脱氧法。整个测序过程由中国科学院上海植物生理所完成。
2 结果
2.1 斑点杂交比较CAV感染后的各代细胞中CAV DNA水平
, http://www.100md.com
以CAV的VP1-pUC18狄高辛标记探针检测CAV感染MDCC-RP1后各代细胞基因组中CAV DNA水平,结果表明: 自传7代的细胞中CAV DNA水平最高,杂交反应呈阳性,而自传14代,19代,26代以及未感染CAV的RP1细胞基因组DNA杂交反应呈阴性。以一系列已知含量的VP1-pUC18克隆DNA阳性样品对照作参考,表明探针的灵敏度可达1~10pg,估计自传7代的细胞基因组DNA每4μg约含50~100pg左右的CAV DNA(图1)。
图1 斑点杂交检测CAV感染的MDCC-RP1各代细胞中CAV核酸
Fig.1 Detection of viral DNA in MDCC-RP1 cells infected with CAV by dot blot hybridigation
, 百拇医药
B5,B6,A1,A2: 4μg genomic DNA of infected cells respectively in 7th, 14th, 19th, 26th passage; A3:4μg genomic DNA of RP1 cells; A4-A6: lng, 100pg, 10pg, 1pg,100fg, 10fg VP1-pUC18 DNA
2.2 两对引物的PCR产物电泳结果
PCR产物经1%琼脂糖凝胶电泳显示: 以CAV感染RP1后自传第7代细胞基因组DNA为模板,引物1和引物2扩增出约1.5kb的条带,与预期长度1512bp一致;引物3和引物4扩增出约1.25kb的条带,与预期长度1243bp或1222bp一致。
2.3 CAV基因组1.5kb片段重组质粒的酶切分析
随意挑取9个白色菌落,经质粒扩增、提取及EcoRI+BamHI双酶消化,发现9个质粒均重组有1.5kb DNA片段,将此重组质粒命名为pCAV1.5-01~09。对重组质粒pCAV1.5-01进一步酶切分析,EcoRI单酶切产生的片段为4.2kb,EcoRI+BamHI双酶消化产生的两个片段为2.7kb、1.5kb,PstI单酶切产生的两个片段为3.3kb、0.9kb,HindⅢ单酶切产生的两个片段为3.5kb、0.7kb(图2)。这与由已发表的Cux-1株CAV基因组序列所推测的结果一致。
, http://www.100md.com
图2 pCAV1.5-01的酶切分析
Fig.2 Electrophoresis of pCAV1.5-01 digested
with restrication enzymes A. Marker λDNA/EcoRI+HindⅢ;
B. pUC18 digested with EcoRI; C. pUC18 digested with BamHI;
D. Digested with EcoRI/BamHI; E. Digested with HindⅢ;
F. Digested with PstI
2.4 包含CAV全基因组序列的重组质粒的酶切分析
, http://www.100md.com
重组质粒pCAV1.5-01在插入CAV基因组1.5kb片段和CAV1.25 kb PCR产物BamHI+SphI双酶切回收的0.9 kb大片段拼接而成的2.4 kb的大片段,包含了CAV全基因组序列,且比CAV基因组多了一段重复的序列,即Meehan等序列[7]的1-82bp段。随意挑取了10个白色菌落,经扩增、小规模提取质粒,以EcoRI酶切,结果所有质粒都产生了5.1kb大小一条带,初步确定该10个质粒都是包含了CAV 2.4kb插入片段的重组质粒,将这10个质粒分别命名为pCAV2.4-01~10。取重组质粒pCAV2.4-01进一步酶切分析,HindⅢ单酶切产生了三个片段大小分别为约1.5kb、1.0kb、0.6kb, BamHI单酶切产生的片段为5.1kb,EcoRI产生的片段为5.1kb(图3)。HindⅢ、BamHI酶切结果与已发表的CUX-1株CAV基因组序列中酶切位点相符,而EcoRI酶切结果与产生大小为2.8kb、2.3kb两片段的预期推测不符,但EcoRI酶切产生的一条带大小为5.1kb,恰是2.8kb、2.3kb两片段大小之和,由此推测发表的CAV基因组DNA中的EcoRI位点,在该克隆中已消失了,可能发生了碱基的突变。构建的重组质粒位点见图4。
, http://www.100md.com
图3 pCAV2.4-01的酶切电泳结果
Fig.3 Electrophoresis of pCAV2.4-01 digested with restrication enzymes
A. Marker λDNA/HindⅢ; B. Digested with EcoRI;
C. Digested with BamHI; D. Digested with HindⅢ.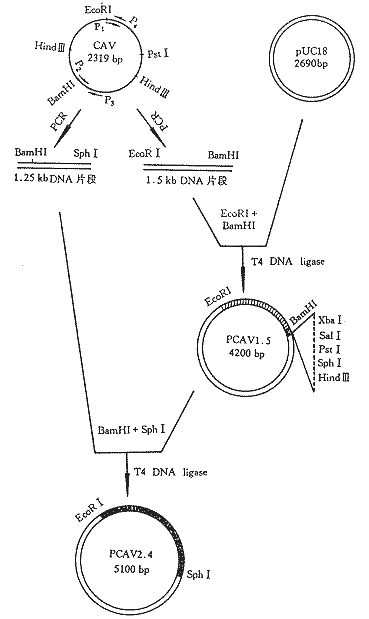
图4 CAV全基因组克隆的构建过程
Fig.4 Construction of recombinant plasmid containing CAV genome
, 百拇医药
2.5 包含CAV全基因组的克隆DNA的两端序列分析
包含CAV全基因组的重组质粒pCAV2.4-01的插入片段的两端DNA序列如图5A、5B。pCAV2.4-01 EcoRI克隆位点即5′端碱基序列如图5A。pCAV24-01 SphI克隆位点端即3′端碱基序列如图5B。
比较所测序列与已发表的Cux-1株CAV环形基因组DNA序列[7],具有高度一致性。稍有不同的是二者共有13处碱基差异,其中6个碱基差异发生在CAV衣壳蛋白VP1基因阅读框架内,但都没有引起相应位置氨基酸的改变。位于CAV环形基因组中的非编码区的EcoRI位点在克隆DNA中确实发生了突变,由GAATTC变为GCATTC(图6A中377-382),这与pCAV2.4质粒EcoRI酶切分析结果相符,证实了该位点已突变的推测,所测的两端重复区序列也完全一致。总之,这些都证实我们克隆到了包含CAV全基因组序列的DNA片段。
, 百拇医药
图5A pCAV2.4-01近EcoRI克隆位点序列
Fig.5A The partial sequence down away the EcoRI site in pCAV2.4-01
图5B pCAV2.4-01近SphI克隆位点序列
Fig.5B The partial sequence up away the SphI site in pCAV2.4-01
3 讨论
CAV的基因组是一条只有2.3kb的单股环状负链DNA,但是在易感细胞,如MDCC-MSB1内亦存在着复制型的线状或环状的双股DNA结构,加之CAV基因组中含EcoRI、BamHI、PstI、HindⅢ酶切位点,国外已从CAV感染细胞中克隆出复制型的CAV全基因组。由于我们没有CAV易感细胞,但具有与CAV易感细胞MSB1相类似的另一类马立克氏病病毒转化T淋巴肿瘤细胞系RP1,因此,尝试用RP1细胞来增殖复制CAV病毒,然后以斑点杂交来检测CAV感染的RP1细胞各代中的病毒核酶水平,结果表明CAV感染的RP1能够不断传代下去,直至26代细胞并没有出现程序性死亡或病变,用灵敏度达1~10pg的探针在斑点杂交中检测到7代细胞中的CAV DNA,据估算约4μg基因组DNA中含50~100pg。为了克隆CAV全基因组,采取了PCR扩增的策略。曾设计了一对反向引物来扩增CAV全基因组,但未能成功。又采取分步PCR扩增覆盖了整个基因组全部序列的两段DNA,再将相应部分拼接克隆的策略。为了拼接克隆的方便,在引物4的5′末端人为添加了一个SphI位点序列,使容易拼接。这样拼接克隆到的片段包含了CAV全基因组序列,但比CAV全基因序列多了一段82bp序列,通过EcoRI酶切,能够获得完整的CAV全基因组的DNA片段。
, 百拇医药
酶切分析和序列分析结果表明,我们已经克隆到包含CAV全基因组序列的DNA片段。但序列分析结果与已发表序列比较,还有少数碱基差异,所测的455个碱基序列中,共有13个碱基变动,包括EcoRI位点中GAATTC→GACTTCC。虽然不能排除少数碱基是由PCR过程中的突变造成的,但我们认为多数碱基差异是毒株长期传代突变的结果。因为所测序列13个碱基差异中有6个发生在VP1基因阅读框架内,但都没有引起相应位置氨基酸的改变。这与PCR随机突变不符。而发生在CAV基因组非编码区中的7个碱基差异,除富含AT的调控序列CACTAT→CACAAT外,其它6个碱基差异都没有发生在目前已知的较为保守的调控序列中。两篇外国文献[7,10]所报道的CuX-1株CAV的基因组序列有很大差异就是毒株自然突变的一个佐证。
本研究获得了CAV全基因组DNA克隆,首先能够提供一个用作CAV检测用的理想探针,其次对该重组质粒克隆片段中突变了的EcoRI位点序列进行校正后,用含有串连着二套CAV基因组的重组质粒DNA转染MSB1,有可能产生具有感染性的病毒,由于CAV基因组很小,其中与感染和致病相关的三个编码基因已完全弄清,因此将可能进一步以定向突变的方法,构建无致病性但仍然能在体内复制组装成有传染性的CAV,作为预防CAV感染的基因工程疫苗。
, 百拇医药
参考文献
[1] Yuasa N, Taniguchi T, Yoshida I.Isolation and characterization of an agent inducing anaemia in chicks. Avian Diseases, 1979,23: 366~385
[2] 陆承平. 最新脊椎动物病毒分类简介. 中国兽医学报,1996,16(1):94~100
[3] Gelderblom H, Kling S, Lurz R. Morphological characterization of chicken anaemia agent (CAA). Arch Virol, 1989,109:115~120
[4] Phenix KV, Meehan BM, Todd D et al. Transcriptional analysis and genome expression of chicken anaemia virus. Journal of General Virology, 1994,75:905~909
, http://www.100md.com
[5] Noteborn MHM, Koch G. Chicken anaemia virus infection: molecular basis of pathogenicity (review). Avian Pathology, 1995,24:11~31
[6] McNulty, MS, McIlroy SG, Bruce DW et al. Economic effects of subclinical chicken anaemia agent infection in broiler chickens. Avian Diseases, 1991,35:263~268
[7] Meehan BM, Todd D, Creelan JL et al. Characterization of viral DNAs from cells infected with chicken anaemia agent: sequence analysis of the cloned replicative form and transfection capabilities of cloned genome fragments. Arichives of Virology, 1992,124:301~319
, 百拇医药
[8] J.萨姆布鲁克,E.F.费里奇,T.曼尼阿蒂斯著. 金冬雁等译.分子克隆(实验指南). 第二版. 北京:科学出版社,1992.
[9] 刘岳龙,崔治中,段玉友. 用PCR扩增和克隆鸡贫血病毒衣壳蛋白基因. 江苏农学院学报,1997,18(2) :49~52
[10] Noteborn MHM, de Boer GF, van Roozelaar DJ et al. Characterization of cloned chicken anemia virus DNA that contains all elements for the infectious replication cycle. Journal of Virology, 1991,65(6): 3131~3139
收稿日期1998-01-18修回日期:1998-06-18, 百拇医药
单位:扬州大学畜牧兽医学院动医系,扬州225009
关键词:鸡传染性贫血病毒;基因组DNA;聚合酶链式反应;克隆
中国病毒学990115 摘 要 根据已发表的序列,分别在鸡贫血病毒(CAV)环形基因组DNA(全长2.3kb)的EcoRI位点和BamHI位点的两侧选择适当序列合成两对引物,用PCR技术,从斑点杂交检测到病毒核酸的CAV感染的MDCC-RP1细胞基因组DNA中,分别扩增出包含EcoRI和BamHI分割开的病毒基因组两部分(1.5kb和0.8kb)约1.5kb和约1.25kb的两个片段。再将其中相应序列拼接克隆进pUC18载体,获得包含CAV全基因组序列DNA片段的克隆质粒pCAV2.4。酶切分析表明,该质粒具有预期的BamHI位点、PstI位点、HindⅢ位点,而预期的EcoRI位点消失。重组质粒插入DNA片段的两端序列分析表明,质粒pCAV2.4是包含CAV全基因组序列的重组质粒,插入DNA片段序列中的EcoRI位点序列发生了一个碱基突变。
, 百拇医药
Amplification and Cloning of Chicken Anemia Virus whole
genomic DNA by Polymerase Chain Reaction
Chen Weimin Cui Zhizhong Duang Yuyou et al
(College of Animal Science and Veterinary Medicine, Yangzhou University, Yangzhou 225009)
Abstract By two pairs of primers complementory to the published sequences at the both sides of EcoRI site and BamHI site on CAV circular genome, two DNA fragments containing two parts (1.5 kb and 0.8 kb) of CAV genome divided by EcoRI and BamHI sites were amplified by PCR from the circular CAV genome (2.3 kb) detected by dot blot in genomic DNA of CAV-infected RP1 cells. Through re-ligation of the selected sequences from the amplified fragments, the whole CAV genomic DNA was cloned into pUC18, and the recombinant plasmid was designated as pCAV2.4. Restriction endonucleases analysis of pCAV2.4 showed that there were sites of BamHI、PstI、HindⅢ,but no site of EcoRI. Two ends of cloned DNA sequence analysis showed that the recombinant plasmid pCAV2.4 containes the whole genomic DNA sequence of CAV, and that the site sequence of EcoRI in the cloned DNA was changed by one base substitution.
, 百拇医药
Key words Chicken anaemia virus, Genome, PCR, Clone
自Yuasa等[1]报道鸡贫血病毒(CAV)以来,世界各地均陆续证明了该病毒感染的普遍性。CAV属于一个新的病毒科——圆环病毒科[2]。这是一种无囊膜的呈二十面体的小病毒,直径只有20nm左右。CAV的基因组是一条只有2.3kb的单股环状“-”链DNA[3],只产生一个转录子,可编码三个蛋白质,即衣壳蛋白VP1及其相关蛋白VP2、细胞凋亡因子VP3[4]。CAV的致病性在于通过CAV编码产生的细胞凋亡因子造成的细胞程序性死亡[5],主要是造血细胞和淋巴细胞的程序性死亡,进而造成感染鸡的贫血,出血及免疫抑制。在野外,感染CAV几乎不引起典型的临床症状,但由于CAV的免疫抑制作用,CAV感染群易并发或继发其它疾病,以及常引起疫苗免疫失败等情况。CAV的亚临床感染严重影响着养禽业的经济效益[6],然而至今仍没有理想的预防CAV感染的方法。国外将CAV基因组双拷贝串连在一起克隆进载体质粒后,所得到的重组质粒DNA转染MDCC-MSB1细胞后可产生传染性的CAV[7]。这就有可能用基因工程的方法构建出能在体内复制的无致病性CAV毒株。为了尝试构建这样的毒株用作疫苗株,本研究用PCR技术成功地扩增和克隆了鸡贫血病毒全基因组DNA。
, http://www.100md.com
1 材料与方法
1.1 材料
1.1.1 引物 引物1:5′CATGATGAATTCCGAGTGGTTACTA3′
引物2:5′CATGATGGATCCCTCATTCTTAGTG3′
引物3:5′CATGATCGCCAAGATCTCTGTGAACCTG3′
引物4:5′CATGATGCATGCGAGCGAAGTCGAGCGATTCGTCCA3′
引物1和引物2分别对应于Noterborn等[10]发表的CAV环形基因组DNA序列的1~19及互补链的1512~1494序列,用于扩增CAV环形基因组1~1512间1512bp DNA片段。两引物5′端分别具有EcoRI、BamHI酶切位点序列。
, http://www.100md.com
引物3和引物4分别对应于Noteborn等[10]发表的环形基因组DNA序列的1158~1179及互补链的82~58序列,用于扩增CAV环形基因组1158~82间的DNA片段。引物4的5′端人为添加有SphI酶切位点序列。
1.1.2 细胞及病毒
MDCC-RP1细胞系,由本教研室提供;CAVCux-1株,由中国兽医监察所惠赠。
1.1.3 菌株及质粒
大肠杆菌TG1用作质粒转化的受体菌,pUC18质粒用作克隆的载体。二者皆由本教研室保存提供。
1.1.4 分子生物学试剂
Dig DNA labeling and Detection Kit、PCR Kit、连接酶及各种限制性内切酶等均购自商业化公司,CAV VP1基因质粒[9]由本教研室提供。
, 百拇医药
1.2 方法
1.2.1 CAV感染MDCC-RP1基因组DNA的制备
以含10%胎牛血清的RPMI-1640培养液培养MDCC-RP1,CAV Cux-1株感染后,每48h自传一次,分别收集第7、14、19、26代细胞和未接种病毒的RP1细胞,按常规[7,8]提取细胞基因组DNA。
1.2.2 斑点杂交比较CAV感染的MDCC-RP1各代细胞中的病毒核酸的水平
对提取的各代细胞基因组DNA浓度定量后,以一系列已知浓度梯度的CAV VP1基因克隆DNA作为参考,用狄高辛标记的CAV VP1基因作为探针。在斑点杂交中检测和比较CAV感染后各代细胞中的CAV DNA水平。
1.2.3 PCR扩增
, http://www.100md.com
引物1和引物2,引物3和引物4组成两对上下游引物,分别用于扩增包含CAV环形基因组EcoRI和BamHI分割开两部分的两DNA片段,1.5kb及1.25kb片段。其PCR扩增步骤基本一致,即:100μL反应体积中,模板DNA 46ng,引物浓度各为2.5μmol/L,4种dNTP浓度为200μmol/L,10×buffer组成为100mmol/L Tris(pH8.3),500mmol/L KCL,15mmol/L MgCl2。反应混合液98℃变性10min,在室温下退火的同时迅速加入1μL Taq DNA聚合酶,覆盖液体石蜡油100μL,离心数秒,进入PCR循环: 94℃ 60s-55℃ 60s-72℃ 180s,共30个循环,最后一个循环后72℃再延伸10min。取PCR产物用1%琼脂糖凝胶电泳,以λDNA/HindⅢ消化物为标记物,观察扩增片段长度。
1.2.4 1.5kb PCR产物的克隆及重组质粒的酶切分析
将已纯化的1.5kb PCR产物与载体pUC18分别用EcoRI、BamHI双酶消化,低熔点琼脂糖电泳回收后,按常规[8]连接、回收。挑取若干单个白斑,进行质粒扩增、提纯,以EcoRI、BamHI双酶切筛选出重组克隆。对重组质粒分别用EcoRI+BamHI、HindⅢ、PstI等内切酶进行酶切,验证插入片段内部酶切位点是否符合预期的结果。
, http://www.100md.com
1.2.5 两PCR扩增片段相应部分的拼接克隆及重组质粒的酶切分析
将已纯化的1.25kb PCR产物与获得的1.5kb PCR产物的重组质粒分别用BamHI、SphI酶消化后,低熔点琼脂糖电泳后,分别回收0.9kb、4.2kb大小的片段,按常规[8]连接转化。挑取若干单个白斑,分别进行质粒扩增、提取,酶切分析,筛选出包含2.4kb插入片段的重组质粒。对筛选到的重组质粒进一步用EcoRI、BamHI、PstI酶切分析。
1.2.6 包含CAV全基因组的重组质粒中插入片段的两段序列分析
采用双脱氧法。整个测序过程由中国科学院上海植物生理所完成。
2 结果
2.1 斑点杂交比较CAV感染后的各代细胞中CAV DNA水平
, http://www.100md.com
以CAV的VP1-pUC18狄高辛标记探针检测CAV感染MDCC-RP1后各代细胞基因组中CAV DNA水平,结果表明: 自传7代的细胞中CAV DNA水平最高,杂交反应呈阳性,而自传14代,19代,26代以及未感染CAV的RP1细胞基因组DNA杂交反应呈阴性。以一系列已知含量的VP1-pUC18克隆DNA阳性样品对照作参考,表明探针的灵敏度可达1~10pg,估计自传7代的细胞基因组DNA每4μg约含50~100pg左右的CAV DNA(图1)。

图1 斑点杂交检测CAV感染的MDCC-RP1各代细胞中CAV核酸
Fig.1 Detection of viral DNA in MDCC-RP1 cells infected with CAV by dot blot hybridigation
, 百拇医药
B5,B6,A1,A2: 4μg genomic DNA of infected cells respectively in 7th, 14th, 19th, 26th passage; A3:4μg genomic DNA of RP1 cells; A4-A6: lng, 100pg, 10pg, 1pg,100fg, 10fg VP1-pUC18 DNA
2.2 两对引物的PCR产物电泳结果
PCR产物经1%琼脂糖凝胶电泳显示: 以CAV感染RP1后自传第7代细胞基因组DNA为模板,引物1和引物2扩增出约1.5kb的条带,与预期长度1512bp一致;引物3和引物4扩增出约1.25kb的条带,与预期长度1243bp或1222bp一致。
2.3 CAV基因组1.5kb片段重组质粒的酶切分析
随意挑取9个白色菌落,经质粒扩增、提取及EcoRI+BamHI双酶消化,发现9个质粒均重组有1.5kb DNA片段,将此重组质粒命名为pCAV1.5-01~09。对重组质粒pCAV1.5-01进一步酶切分析,EcoRI单酶切产生的片段为4.2kb,EcoRI+BamHI双酶消化产生的两个片段为2.7kb、1.5kb,PstI单酶切产生的两个片段为3.3kb、0.9kb,HindⅢ单酶切产生的两个片段为3.5kb、0.7kb(图2)。这与由已发表的Cux-1株CAV基因组序列所推测的结果一致。

, http://www.100md.com
图2 pCAV1.5-01的酶切分析
Fig.2 Electrophoresis of pCAV1.5-01 digested
with restrication enzymes A. Marker λDNA/EcoRI+HindⅢ;
B. pUC18 digested with EcoRI; C. pUC18 digested with BamHI;
D. Digested with EcoRI/BamHI; E. Digested with HindⅢ;
F. Digested with PstI
2.4 包含CAV全基因组序列的重组质粒的酶切分析
, http://www.100md.com
重组质粒pCAV1.5-01在插入CAV基因组1.5kb片段和CAV1.25 kb PCR产物BamHI+SphI双酶切回收的0.9 kb大片段拼接而成的2.4 kb的大片段,包含了CAV全基因组序列,且比CAV基因组多了一段重复的序列,即Meehan等序列[7]的1-82bp段。随意挑取了10个白色菌落,经扩增、小规模提取质粒,以EcoRI酶切,结果所有质粒都产生了5.1kb大小一条带,初步确定该10个质粒都是包含了CAV 2.4kb插入片段的重组质粒,将这10个质粒分别命名为pCAV2.4-01~10。取重组质粒pCAV2.4-01进一步酶切分析,HindⅢ单酶切产生了三个片段大小分别为约1.5kb、1.0kb、0.6kb, BamHI单酶切产生的片段为5.1kb,EcoRI产生的片段为5.1kb(图3)。HindⅢ、BamHI酶切结果与已发表的CUX-1株CAV基因组序列中酶切位点相符,而EcoRI酶切结果与产生大小为2.8kb、2.3kb两片段的预期推测不符,但EcoRI酶切产生的一条带大小为5.1kb,恰是2.8kb、2.3kb两片段大小之和,由此推测发表的CAV基因组DNA中的EcoRI位点,在该克隆中已消失了,可能发生了碱基的突变。构建的重组质粒位点见图4。

, http://www.100md.com
图3 pCAV2.4-01的酶切电泳结果
Fig.3 Electrophoresis of pCAV2.4-01 digested with restrication enzymes
A. Marker λDNA/HindⅢ; B. Digested with EcoRI;
C. Digested with BamHI; D. Digested with HindⅢ.

图4 CAV全基因组克隆的构建过程
Fig.4 Construction of recombinant plasmid containing CAV genome
, 百拇医药
2.5 包含CAV全基因组的克隆DNA的两端序列分析
包含CAV全基因组的重组质粒pCAV2.4-01的插入片段的两端DNA序列如图5A、5B。pCAV2.4-01 EcoRI克隆位点即5′端碱基序列如图5A。pCAV24-01 SphI克隆位点端即3′端碱基序列如图5B。
比较所测序列与已发表的Cux-1株CAV环形基因组DNA序列[7],具有高度一致性。稍有不同的是二者共有13处碱基差异,其中6个碱基差异发生在CAV衣壳蛋白VP1基因阅读框架内,但都没有引起相应位置氨基酸的改变。位于CAV环形基因组中的非编码区的EcoRI位点在克隆DNA中确实发生了突变,由GAATTC变为GCATTC(图6A中377-382),这与pCAV2.4质粒EcoRI酶切分析结果相符,证实了该位点已突变的推测,所测的两端重复区序列也完全一致。总之,这些都证实我们克隆到了包含CAV全基因组序列的DNA片段。
, 百拇医药
图5A pCAV2.4-01近EcoRI克隆位点序列
Fig.5A The partial sequence down away the EcoRI site in pCAV2.4-01

图5B pCAV2.4-01近SphI克隆位点序列
Fig.5B The partial sequence up away the SphI site in pCAV2.4-01
3 讨论
CAV的基因组是一条只有2.3kb的单股环状负链DNA,但是在易感细胞,如MDCC-MSB1内亦存在着复制型的线状或环状的双股DNA结构,加之CAV基因组中含EcoRI、BamHI、PstI、HindⅢ酶切位点,国外已从CAV感染细胞中克隆出复制型的CAV全基因组。由于我们没有CAV易感细胞,但具有与CAV易感细胞MSB1相类似的另一类马立克氏病病毒转化T淋巴肿瘤细胞系RP1,因此,尝试用RP1细胞来增殖复制CAV病毒,然后以斑点杂交来检测CAV感染的RP1细胞各代中的病毒核酶水平,结果表明CAV感染的RP1能够不断传代下去,直至26代细胞并没有出现程序性死亡或病变,用灵敏度达1~10pg的探针在斑点杂交中检测到7代细胞中的CAV DNA,据估算约4μg基因组DNA中含50~100pg。为了克隆CAV全基因组,采取了PCR扩增的策略。曾设计了一对反向引物来扩增CAV全基因组,但未能成功。又采取分步PCR扩增覆盖了整个基因组全部序列的两段DNA,再将相应部分拼接克隆的策略。为了拼接克隆的方便,在引物4的5′末端人为添加了一个SphI位点序列,使容易拼接。这样拼接克隆到的片段包含了CAV全基因组序列,但比CAV全基因序列多了一段82bp序列,通过EcoRI酶切,能够获得完整的CAV全基因组的DNA片段。
, 百拇医药
酶切分析和序列分析结果表明,我们已经克隆到包含CAV全基因组序列的DNA片段。但序列分析结果与已发表序列比较,还有少数碱基差异,所测的455个碱基序列中,共有13个碱基变动,包括EcoRI位点中GAATTC→GACTTCC。虽然不能排除少数碱基是由PCR过程中的突变造成的,但我们认为多数碱基差异是毒株长期传代突变的结果。因为所测序列13个碱基差异中有6个发生在VP1基因阅读框架内,但都没有引起相应位置氨基酸的改变。这与PCR随机突变不符。而发生在CAV基因组非编码区中的7个碱基差异,除富含AT的调控序列CACTAT→CACAAT外,其它6个碱基差异都没有发生在目前已知的较为保守的调控序列中。两篇外国文献[7,10]所报道的CuX-1株CAV的基因组序列有很大差异就是毒株自然突变的一个佐证。
本研究获得了CAV全基因组DNA克隆,首先能够提供一个用作CAV检测用的理想探针,其次对该重组质粒克隆片段中突变了的EcoRI位点序列进行校正后,用含有串连着二套CAV基因组的重组质粒DNA转染MSB1,有可能产生具有感染性的病毒,由于CAV基因组很小,其中与感染和致病相关的三个编码基因已完全弄清,因此将可能进一步以定向突变的方法,构建无致病性但仍然能在体内复制组装成有传染性的CAV,作为预防CAV感染的基因工程疫苗。
, 百拇医药
参考文献
[1] Yuasa N, Taniguchi T, Yoshida I.Isolation and characterization of an agent inducing anaemia in chicks. Avian Diseases, 1979,23: 366~385
[2] 陆承平. 最新脊椎动物病毒分类简介. 中国兽医学报,1996,16(1):94~100
[3] Gelderblom H, Kling S, Lurz R. Morphological characterization of chicken anaemia agent (CAA). Arch Virol, 1989,109:115~120
[4] Phenix KV, Meehan BM, Todd D et al. Transcriptional analysis and genome expression of chicken anaemia virus. Journal of General Virology, 1994,75:905~909
, http://www.100md.com
[5] Noteborn MHM, Koch G. Chicken anaemia virus infection: molecular basis of pathogenicity (review). Avian Pathology, 1995,24:11~31
[6] McNulty, MS, McIlroy SG, Bruce DW et al. Economic effects of subclinical chicken anaemia agent infection in broiler chickens. Avian Diseases, 1991,35:263~268
[7] Meehan BM, Todd D, Creelan JL et al. Characterization of viral DNAs from cells infected with chicken anaemia agent: sequence analysis of the cloned replicative form and transfection capabilities of cloned genome fragments. Arichives of Virology, 1992,124:301~319
, 百拇医药
[8] J.萨姆布鲁克,E.F.费里奇,T.曼尼阿蒂斯著. 金冬雁等译.分子克隆(实验指南). 第二版. 北京:科学出版社,1992.
[9] 刘岳龙,崔治中,段玉友. 用PCR扩增和克隆鸡贫血病毒衣壳蛋白基因. 江苏农学院学报,1997,18(2) :49~52
[10] Noteborn MHM, de Boer GF, van Roozelaar DJ et al. Characterization of cloned chicken anemia virus DNA that contains all elements for the infectious replication cycle. Journal of Virology, 1991,65(6): 3131~3139
收稿日期1998-01-18修回日期:1998-06-18, 百拇医药