一种稳定、易于操作的噬菌体随机肽库载体的构建
作者:万瑛 代佳平 王燕 刘昕
单位:万瑛 代佳平 王燕 刘昕 第三军医大学基础医学部分子生物学教研室 重庆,400038
关键词:噬菌粒随机肽库;pCOMB3;Dot-Plot分析
提 要 目的提 要 目的:拟将噬菌粒pCOMB3改建为更为稳定、高拷贝且易于操作的噬菌体随机肽库载体。方法:对常用噬菌体抗体库呈现载体pCOMB3序列进行点矩阵作图分析(Dot-Plot分析 ),切除其中重复区,纠正其多代培养后自删除特性。原克隆位点63bp的填充序列增加到1 435 bp以便于酶切鉴定和电泳回收。结果:构建了命名为pTMB2的噬菌粒载体。结论:pTMB2的噬菌粒载体更为稳定且易于操作。
中图法分类号 R373.9;R394-3
Establishment of a stable and easily manipulated vector for macrophage random peptide library
, 百拇医药
Wan Ying, Dai Jiaping, Wang Yan
Department of Molecular Biology, Third Military Medical University, Chongqing,400038
Abstract Objective: To modify a combinational library display vector, pCOMB3 and to provide a stable, easily manipulated and highly copied vector for the display of a random peptide library. Methods: According to Dot-Plot analysis of pCOMB3 nucleic acid sequences, the phagmid pCOMB3 was modified to eliminate the propensity of accumulated deletions in the region of cloning site. Furthermore, a 1375 bp DNA fragment from phage was cloned in the small 63 bp stuffer at the cloning site. Results: A 5129 vector named pTMB2 was produced. Conclusion: pTMB2 holds the appropriate characteristics for random peptide phage display with improved stability and ease of manipulation.
, http://www.100md.com
Key words phage random peptide library; pCOMB3; Dot-Plot analysis
噬菌体显示库技术是通过在丝状噬菌体表面呈现多样化的多肽,经过筛选获得与特定配基结合的肽类的一种新兴技术。此技术已广泛用于新药开发和蛋白质结构与功能等研究领域[1~3]。pCOMB3是Barbas等[4]于1991年构建的噬菌体抗体库显示载体,其含有AmpR基因和丝状噬菌体小衣壳蛋白geneⅢ C末端序列,具有高拷贝数(>200拷贝/细胞)且易于操作。但此噬菌粒多代培养后易出现外源DNA片段和载体600~800 bp的自删除[5,6],并且其填充序列较短不便于证实限制性内切酶酶切结果和电泳回收。本研究采用Dot-Plot方法分析pCOMB3序列[7,8],推测其自删除突变机制,并对pCOMB3进行相应地改造,为进一步构建丝状噬菌体随机肽库奠定基础。
, 百拇医药
1 材料与方法
1.1 菌株和质粒
Escherichia coli XLl-Blue [F′ proAB, lacIq ZM15,Tn19(tetr)]和pCOMB3分别由本校微生物教研室万泽生博士和本校免疫教研室王喜良博士惠赠。
1.2 主要试剂
XbaI、EcoRI和HindⅢ购于华美生物工程公司,NheI购与New England Biolabs公司,T4连接酶购于Boehrinoer Mannheim公司。λ-DNA/HindⅢ+EcoRI核酶分子量标准构自华美公司(DNA片段大小依此为:21 226,5 148,4 973,4 268,3 530,2 027,1 904,1 584,1 375,947,831和564 bp),小分子量核酶分子量标准购自复华公司(DNA片段大小依此为:501/498,404,331,242,190,147和111/110 bp)。DNA操作参照文献和公司产品指导进行[9]。
, 百拇医药
1.3 多代培养自删除突变检测
构建的pTMB2噬菌粒转化细菌后,挑单菌落接种培养(LB培养基,37℃,16 h),稀释后涂布平板(含氨苄青霉素),随机选取20个克隆抽提DNA,HindⅢ和EcoRI酶切鉴定是否出现外源片段的自删除突变。重复培养、随机选取和酶切鉴定20代。将带有862 bp xho2+spe2外源片段的PCOMB3作为对照,同上鉴定外源片段的自删除突变。
1.4 Dot-Plot序列分析软件
Microsoft ANTHEPROT V2.9g由Gilbert Deleage(France)开发,通过匿名FTP下载。
2 结果
2.1 Microsoft ANTHEPROT V2.9 g分析结果
, 百拇医药
利用ANTHEPROT V2.9g的Dot-Plot分析模块对pCOMB3和pTMB1全序列进行点矩阵作图分析。采用单位矩阵打分,即相同为1,不相同为0。采用窗口过滤技术减弱背景噪音,窗值设为11,阈值设为9,超过阈值的窗口在其中间核苷酸位置打点,结果见图1。图1A中横坐标和纵坐标均为pCOMB3,图1B中横坐标和纵坐标均为pTMB1.我们发现pCOMB3中有两对序列相同的重复区域:一个82 bp(nt 24~105和1037~1118),一个148 bp(nt 871~1017和3809~3952),图中以与对角线平行斜线示之,而构建的pTMB1无明显的重复序列。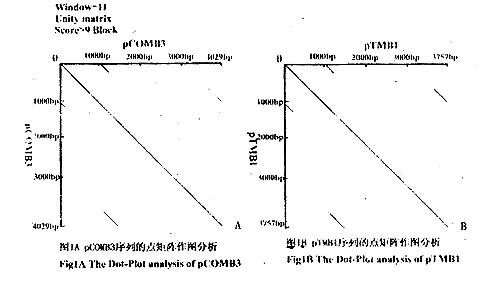
图1 点矩阵作图分析
A:pCOMB3序列 B:pTMB1序列
, 百拇医药
Fig 1 The Dot-Plot analysis
A:pCOMB3 B:pTMB1
2.2 pTMB2的构建
Nhel和XbaI分别位于pCOMB3的859 bp和1 131 bp,故双酶切噬菌粒可切下长约272 bp的DNA片段。低熔点琼脂糖法回收pCOMB3剩余片段,由于XbaI、NheI酶切后产生互补的粘性末端,自连即得称之为pTMB1的噬菌粒,其XbaI、NheI酶切位点消失,可采用XbaI和NheI酶切鉴定重组子。将λ噬菌体1 375 bp的HindⅢ、EcoRI酶切片段定向克隆进pTMB1的克隆位点,增加其填充序列的长度,所得噬菌粒称之为pTMB2,见图2、3。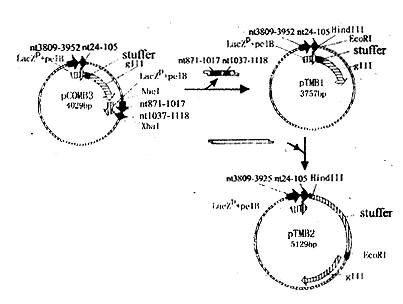
, 百拇医药
图2 噬菌粒pTMB2的构建
Fig 2 Construction of plasmid pTMB2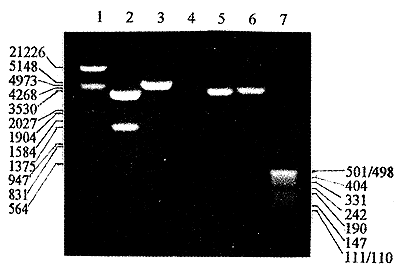
图3 噬菌粒pTMB2的酶切鉴定
1:核酸分子量标准λ HindⅢ, EcoRI 2:pTMB2 EcoRI,HindⅢ双酶切 3:pTMB2 HindⅢ单酶切 4:pTMB1 HindⅢ单酶切 5:pCOMB3 NheI, XbeI双酶切 6:pCOMB3 HindⅢ单酶切 7:复华公司小片段核酸分子量标准
Fig 3 Restriction analysis of phagmid pTMB2
, 百拇医药
Lane l:λ DNA cleaved with HindⅢ and EcoRI as the molecular weight marker; Lane 2:pTMB2 digested with EcoRI and HindⅢ; Lane 3: pTMB2 digested with HindⅢ; Lane 4:pTMB1 digested with HindⅢ; Lane 5:pCOMB3 digested with NheI and XbaI; Lane 6: pCOMB3 digested HindⅢ; Lane 7: Molecular weight marker from Corp FuHua
2.3 多代培养自删除突变的检测
pTMB2经20代培养,每代随机挑选20个菌落检测,400个菌落中均未发现自删除突变。而作为对照含有λ噬菌体1375 bpHindⅢ、EcoRI酶切片段的pCOMB3经五代培养后,即在20随机挑选的菌落中发现有两个菌落出现自删除突变。
, 百拇医药
3 讨论
随机丝状噬菌体肽库的构建首先需要合适的载体,现常用的以小衣壳蛋白为目标的载体系统有两种,一种是直接在丝状噬菌体geneⅢ序列引入克隆位点,另一种则是在利用噬菌粒表达肽与geneⅢ蛋白C末端的融合蛋白。前者虽然较易于操作,但却存在不能引入较长的多肽和感染率下降等缺点。故为了下一步构建20肽的随机肽库,我们选择噬菌粒pCOMB3。pCOMB3是Barbas等[4]于1991年构建的噬菌体抗体库呈现载体。为同时表达抗体的轻链和重链,pCOMB3拥有两个lacZ的启动子和引导序列pel B,见图3。近年在pCOMB3构建的抗体库筛选中,常有报道此噬菌粒多代(3~5代)培养后易出现外源DNA片段和载体600~800 bp的自删除突变[5,6]。采用ANTHEPROT V2.9 g的Dot-Plot分析模块对pCOMB3全序列进行点矩阵作图分析,发现pCOMB3中有两对序列相同的重复区域:一个82 bp(nt24~105和nt1 037~1 118),一个148 bp(nt871~1 017和nt3 809~3 952)。我们认为上经重复区的存在与噬菌粒pCOMB3在复制中发生遗传重组有关,导致其出现自删除突变。XbaI和NheI双酶切切除272 bp片段(nt859~1 131),其中包含2个重复区(nt871~1 017,nt1 037~1 118),见图2,多代培养自删除突变的检测显示所得噬菌粒pTMB2更为稳定。为便于证实限制性内切酶酶切结果和电泳回收,我们将λ噬菌体1375 bp的HindⅢ、EcoRI酶切片段定向克隆进pTMB1的克隆位点,增加其填充序列的长度。这样我们就得到较pCOMB3更为稳定且易于操作的噬菌体随机肽库载体pTMB2,进一步构建丝状噬菌体随机肽库奠定了基础。
, http://www.100md.com
作者简介:万瑛,男,27岁,助教,硕士研究生
参考文献
1 McGregor D. Selection of proteins and peptides from libraries displayed on filamentous bacteriophage. Mol biotechol,1996,6(2):155
2 Gho Y S. Development of antiangiogenin peptide using a phage-displayed peptide library. Cancer Res,1997,57(17):3733
3 Fack F. Epitope mapping by phage display:random versus gene-fragment libraries. J Immunol Methods,1997,206(1~2):43
, 百拇医药
4 Barbas Ⅲ C F, Kang A S, Lerner R A. Assembly of combinatorial antibody libraries on phage surfaces:The gene Ⅲ site. Proc Natl Acad Sci USA,1991,88(21):7 978
5 Barbas Ⅲ C F, Lerner R A. Combinatorial immunoglobulin libraries on the surface of phage (Phabs):rapid selection of antigen-specific Fabs. Method Companion Methods Enzymol,1991,2(1):119
6 张广发,王 琰,王雅明,等.噬菌体抗体库的构建及抗肿瘤单抗的筛选.中国肿瘤杂志,1995,17(4):258
, 百拇医药
7 McDonnell M T, Colwell R R. Dot-matrix analysis of nucleic acid sequences: an interactive microcomputer program. J Microbiol Meth,1985,3(1):247
8 王槐春.蛋白质与核酸序列分析基础.北京:人民科学出版社,1994.45~58
9 Sambrook J, Fritsch E F, Maniatis T. 分子克隆实验指南.第2版.北京:科学出版社,1992:34~69
收稿:1998-09-15
修回:1999-01-22, 百拇医药
单位:万瑛 代佳平 王燕 刘昕 第三军医大学基础医学部分子生物学教研室 重庆,400038
关键词:噬菌粒随机肽库;pCOMB3;Dot-Plot分析
提 要 目的提 要 目的:拟将噬菌粒pCOMB3改建为更为稳定、高拷贝且易于操作的噬菌体随机肽库载体。方法:对常用噬菌体抗体库呈现载体pCOMB3序列进行点矩阵作图分析(Dot-Plot分析 ),切除其中重复区,纠正其多代培养后自删除特性。原克隆位点63bp的填充序列增加到1 435 bp以便于酶切鉴定和电泳回收。结果:构建了命名为pTMB2的噬菌粒载体。结论:pTMB2的噬菌粒载体更为稳定且易于操作。
中图法分类号 R373.9;R394-3
Establishment of a stable and easily manipulated vector for macrophage random peptide library
, 百拇医药
Wan Ying, Dai Jiaping, Wang Yan
Department of Molecular Biology, Third Military Medical University, Chongqing,400038
Abstract Objective: To modify a combinational library display vector, pCOMB3 and to provide a stable, easily manipulated and highly copied vector for the display of a random peptide library. Methods: According to Dot-Plot analysis of pCOMB3 nucleic acid sequences, the phagmid pCOMB3 was modified to eliminate the propensity of accumulated deletions in the region of cloning site. Furthermore, a 1375 bp DNA fragment from phage was cloned in the small 63 bp stuffer at the cloning site. Results: A 5129 vector named pTMB2 was produced. Conclusion: pTMB2 holds the appropriate characteristics for random peptide phage display with improved stability and ease of manipulation.
, http://www.100md.com
Key words phage random peptide library; pCOMB3; Dot-Plot analysis
噬菌体显示库技术是通过在丝状噬菌体表面呈现多样化的多肽,经过筛选获得与特定配基结合的肽类的一种新兴技术。此技术已广泛用于新药开发和蛋白质结构与功能等研究领域[1~3]。pCOMB3是Barbas等[4]于1991年构建的噬菌体抗体库显示载体,其含有AmpR基因和丝状噬菌体小衣壳蛋白geneⅢ C末端序列,具有高拷贝数(>200拷贝/细胞)且易于操作。但此噬菌粒多代培养后易出现外源DNA片段和载体600~800 bp的自删除[5,6],并且其填充序列较短不便于证实限制性内切酶酶切结果和电泳回收。本研究采用Dot-Plot方法分析pCOMB3序列[7,8],推测其自删除突变机制,并对pCOMB3进行相应地改造,为进一步构建丝状噬菌体随机肽库奠定基础。
, 百拇医药
1 材料与方法
1.1 菌株和质粒
Escherichia coli XLl-Blue [F′ proAB, lacIq ZM15,Tn19(tetr)]和pCOMB3分别由本校微生物教研室万泽生博士和本校免疫教研室王喜良博士惠赠。
1.2 主要试剂
XbaI、EcoRI和HindⅢ购于华美生物工程公司,NheI购与New England Biolabs公司,T4连接酶购于Boehrinoer Mannheim公司。λ-DNA/HindⅢ+EcoRI核酶分子量标准构自华美公司(DNA片段大小依此为:21 226,5 148,4 973,4 268,3 530,2 027,1 904,1 584,1 375,947,831和564 bp),小分子量核酶分子量标准购自复华公司(DNA片段大小依此为:501/498,404,331,242,190,147和111/110 bp)。DNA操作参照文献和公司产品指导进行[9]。
, 百拇医药
1.3 多代培养自删除突变检测
构建的pTMB2噬菌粒转化细菌后,挑单菌落接种培养(LB培养基,37℃,16 h),稀释后涂布平板(含氨苄青霉素),随机选取20个克隆抽提DNA,HindⅢ和EcoRI酶切鉴定是否出现外源片段的自删除突变。重复培养、随机选取和酶切鉴定20代。将带有862 bp xho2+spe2外源片段的PCOMB3作为对照,同上鉴定外源片段的自删除突变。
1.4 Dot-Plot序列分析软件
Microsoft ANTHEPROT V2.9g由Gilbert Deleage(France)开发,通过匿名FTP下载。
2 结果
2.1 Microsoft ANTHEPROT V2.9 g分析结果
, 百拇医药
利用ANTHEPROT V2.9g的Dot-Plot分析模块对pCOMB3和pTMB1全序列进行点矩阵作图分析。采用单位矩阵打分,即相同为1,不相同为0。采用窗口过滤技术减弱背景噪音,窗值设为11,阈值设为9,超过阈值的窗口在其中间核苷酸位置打点,结果见图1。图1A中横坐标和纵坐标均为pCOMB3,图1B中横坐标和纵坐标均为pTMB1.我们发现pCOMB3中有两对序列相同的重复区域:一个82 bp(nt 24~105和1037~1118),一个148 bp(nt 871~1017和3809~3952),图中以与对角线平行斜线示之,而构建的pTMB1无明显的重复序列。

图1 点矩阵作图分析
A:pCOMB3序列 B:pTMB1序列
, 百拇医药
Fig 1 The Dot-Plot analysis
A:pCOMB3 B:pTMB1
2.2 pTMB2的构建
Nhel和XbaI分别位于pCOMB3的859 bp和1 131 bp,故双酶切噬菌粒可切下长约272 bp的DNA片段。低熔点琼脂糖法回收pCOMB3剩余片段,由于XbaI、NheI酶切后产生互补的粘性末端,自连即得称之为pTMB1的噬菌粒,其XbaI、NheI酶切位点消失,可采用XbaI和NheI酶切鉴定重组子。将λ噬菌体1 375 bp的HindⅢ、EcoRI酶切片段定向克隆进pTMB1的克隆位点,增加其填充序列的长度,所得噬菌粒称之为pTMB2,见图2、3。

, 百拇医药
图2 噬菌粒pTMB2的构建
Fig 2 Construction of plasmid pTMB2

图3 噬菌粒pTMB2的酶切鉴定
1:核酸分子量标准λ HindⅢ, EcoRI 2:pTMB2 EcoRI,HindⅢ双酶切 3:pTMB2 HindⅢ单酶切 4:pTMB1 HindⅢ单酶切 5:pCOMB3 NheI, XbeI双酶切 6:pCOMB3 HindⅢ单酶切 7:复华公司小片段核酸分子量标准
Fig 3 Restriction analysis of phagmid pTMB2
, 百拇医药
Lane l:λ DNA cleaved with HindⅢ and EcoRI as the molecular weight marker; Lane 2:pTMB2 digested with EcoRI and HindⅢ; Lane 3: pTMB2 digested with HindⅢ; Lane 4:pTMB1 digested with HindⅢ; Lane 5:pCOMB3 digested with NheI and XbaI; Lane 6: pCOMB3 digested HindⅢ; Lane 7: Molecular weight marker from Corp FuHua
2.3 多代培养自删除突变的检测
pTMB2经20代培养,每代随机挑选20个菌落检测,400个菌落中均未发现自删除突变。而作为对照含有λ噬菌体1375 bpHindⅢ、EcoRI酶切片段的pCOMB3经五代培养后,即在20随机挑选的菌落中发现有两个菌落出现自删除突变。
, 百拇医药
3 讨论
随机丝状噬菌体肽库的构建首先需要合适的载体,现常用的以小衣壳蛋白为目标的载体系统有两种,一种是直接在丝状噬菌体geneⅢ序列引入克隆位点,另一种则是在利用噬菌粒表达肽与geneⅢ蛋白C末端的融合蛋白。前者虽然较易于操作,但却存在不能引入较长的多肽和感染率下降等缺点。故为了下一步构建20肽的随机肽库,我们选择噬菌粒pCOMB3。pCOMB3是Barbas等[4]于1991年构建的噬菌体抗体库呈现载体。为同时表达抗体的轻链和重链,pCOMB3拥有两个lacZ的启动子和引导序列pel B,见图3。近年在pCOMB3构建的抗体库筛选中,常有报道此噬菌粒多代(3~5代)培养后易出现外源DNA片段和载体600~800 bp的自删除突变[5,6]。采用ANTHEPROT V2.9 g的Dot-Plot分析模块对pCOMB3全序列进行点矩阵作图分析,发现pCOMB3中有两对序列相同的重复区域:一个82 bp(nt24~105和nt1 037~1 118),一个148 bp(nt871~1 017和nt3 809~3 952)。我们认为上经重复区的存在与噬菌粒pCOMB3在复制中发生遗传重组有关,导致其出现自删除突变。XbaI和NheI双酶切切除272 bp片段(nt859~1 131),其中包含2个重复区(nt871~1 017,nt1 037~1 118),见图2,多代培养自删除突变的检测显示所得噬菌粒pTMB2更为稳定。为便于证实限制性内切酶酶切结果和电泳回收,我们将λ噬菌体1375 bp的HindⅢ、EcoRI酶切片段定向克隆进pTMB1的克隆位点,增加其填充序列的长度。这样我们就得到较pCOMB3更为稳定且易于操作的噬菌体随机肽库载体pTMB2,进一步构建丝状噬菌体随机肽库奠定了基础。
, http://www.100md.com
作者简介:万瑛,男,27岁,助教,硕士研究生
参考文献
1 McGregor D. Selection of proteins and peptides from libraries displayed on filamentous bacteriophage. Mol biotechol,1996,6(2):155
2 Gho Y S. Development of antiangiogenin peptide using a phage-displayed peptide library. Cancer Res,1997,57(17):3733
3 Fack F. Epitope mapping by phage display:random versus gene-fragment libraries. J Immunol Methods,1997,206(1~2):43
, 百拇医药
4 Barbas Ⅲ C F, Kang A S, Lerner R A. Assembly of combinatorial antibody libraries on phage surfaces:The gene Ⅲ site. Proc Natl Acad Sci USA,1991,88(21):7 978
5 Barbas Ⅲ C F, Lerner R A. Combinatorial immunoglobulin libraries on the surface of phage (Phabs):rapid selection of antigen-specific Fabs. Method Companion Methods Enzymol,1991,2(1):119
6 张广发,王 琰,王雅明,等.噬菌体抗体库的构建及抗肿瘤单抗的筛选.中国肿瘤杂志,1995,17(4):258
, 百拇医药
7 McDonnell M T, Colwell R R. Dot-matrix analysis of nucleic acid sequences: an interactive microcomputer program. J Microbiol Meth,1985,3(1):247
8 王槐春.蛋白质与核酸序列分析基础.北京:人民科学出版社,1994.45~58
9 Sambrook J, Fritsch E F, Maniatis T. 分子克隆实验指南.第2版.北京:科学出版社,1992:34~69
收稿:1998-09-15
修回:1999-01-22, 百拇医药