乙型肝炎病毒全基因组研究的新方法
作者:何建文 何丽芳 姚忻 闻玉梅
单位:200032 上海 医科大学卫生部医学分子病毒学重点实验室
关键词:肝炎病毒,乙型;基因组;聚合酶链反应
中华传染病杂980203 【摘要】 目的 为从全基因水平研究乙型肝炎病毒变异与致病性的关系,建立血清标本经聚合酶链反应(PCR)加酶切方法扩增及克隆HBV全基因组的新方法。方法 设计了含SpeⅠ,SalⅠ及SapⅠ酶切点的引物,分别扩增1.15kb和2.05kb的单链及双链DNA区。经酶切及连接后获得HBV全基因克隆。结果 用新建立的方法直接从少量血清中克隆了HBV全基因,转染HepG2细胞可表达抗原并有胞内复制。结论 该法可用于对临床上HBV毒株的基因结构和功能的研究。
A novel method to study the full-length genome of hepatitis B virus He Jianwen, He Lifang, Yao Xin, et al. Department of Molecular Virology, Shanghai Medical University, Shanghai 200032
, http://www.100md.com
【Abstract】 Objective To study the association between pathogenicity and variants of hepatitis B virus(HBV) by full-length HBV genome, a novel method of amplification of the whole genome of HBV strains from serum samples of patients by PCR, followed by enzyme digestion and cloning was established. Methods Two sets of primers were designed: one with SpeⅠ restriction site, the other with restriction sites of SalⅠ and SapⅠ. PCR products were amplified separately from the single-stranded and the double-stranded regions of the genome, and the two PCR products obtained were 1.15kb and 2.05kb. After cleavage and ligation, the full-length HBV genome was used to transfect HepG2 cell line. Results Expression of viral antigens and intracellular replication of HBV were observed with the full-length HBV genome by the novel method. Conclusion This novel method could serve to study the structure and function of HBV genome from viral strains of clinical samples.
, http://www.100md.com
【Key words】 Hepatitis B virus Genome PCR
对乙型肝炎病毒(HBV)基因变异的研究提示,一些基因位点的变异可能与临床及流行病有关[1,2]。由于多数研究仅限于对某一基因片段的研究,而HBV基因内部有调控基因,基因片段间又相互重叠,研究单一片段难以说明问题。以往对HBV全基因的克隆需从大量血清中提取HBV,故难以对不同患者单一毒株作HBV全基因克隆。本研究根据Gunther等[3]的报道(Gunther法)并加以改进后,建立了一种聚合酶链反应(PCR)加酶切及酶连接后,从少量血清中一次克隆HBV全基因的方法(二片段法)。用获得的全基因转染细胞,可短期表达HBsAg和HBeAg并有胞内HBV复制。这一新方法为研究HBV毒株的基因结构与功能提供了可行的途径。
材料和方法
一、血清HBV DNA提取
, http://www.100md.com
方法见参考文献[4]。主要步骤:100μl血清加TES(10mmol/L Tris-HCl,pH8.0,5mmol/L EDTA,0.5%SDS,150μg/ml蛋白酶K),65°C 3小时,常规酚/氯仿提取,乙醇沉淀,DNA溶解于20μl双蒸水中。
二、引物设计
设计两对引物从不同方向分别扩增单链区及双链区,扩增片段分别为1.15kb和2.05kb长短。通过对已发表的36株HBV全基因组的同源及酶切点分析,选择含单一酶切点(SpeⅠ)的共有序列(nt669-699)分别合成正链和负链引物(PSP1和PSP2)。此外,根据我国HBVadr株存在HindⅢ酶切点但无SalⅠ酶切点,将Gunther设计的原引物酶切序列由HindⅢ改为SalⅠ,分别合成了P3S1和P3S2(nt1821-1841,nt1806-1825)。各引物序列为:
PSP15′-GGCTCAGTTTACTAGTGCCATTTGTTCA-
, 百拇医药
SpeⅠ
GTG-3′正链nt 669-699
PSP25′-CACTGAACAAATGGCACTAGTAAACTGA-
SalⅠ SapⅠ
GCC-3′负链nt 699-669
P3S1 5′-CCGGCGTCGACGAGCTCTTCTTTTTCACC-
SalⅠ SapⅠ
TCTGCCTAATCA-3′正链nt1821-1841
P3S2 5′-CCGGCGTCGACGAGCTCTTCAAAAAGTTGCATGGTGCTGG-3′负链nt1806-1825
, http://www.100md.com
三、PCR扩增
自血清标本提取的DNA分作二份模板,分别用P3S1和PSP2,P3S2和PSP1扩增,反应条件为100μl反应体积中包括引物各100pmol/L,4×dNTP各0.2mmol/L,MgCl2 1.5mmol/L,1×缓冲液及TaqDNA聚合酶2.5U(Gibco公司)。PCR于94°C 4分钟后,94°C 50秒,52°C 90秒,72°C90秒(每循环自动延伸10秒),30个循环后,于72°C延伸10分钟。
四、扩增产物的酶切及克隆
扩增产物经纯化后,用SalⅠ和SpeⅠ进行双酶切,同时用SalⅠ酶切载体pUC19。酶切后电泳并从凝胶中回收酶切片段,用连接酶(Promega公司)连接过夜后转化大肠杆菌JM109,37℃培养过夜,挑取白色菌落,提取质粒DNA,用酶切法及核苷酸自动测序鉴定。
, http://www.100md.com
五、HBV全基因片段的回收
经证实插入全基因片段的克隆转种于100ml培养液,用Qiagen midi(Qiagen公司)纯化质粒DNA,用SapⅠ(Gene-Lab公司)酶切(0.5U/μg DNA)16~20小时,低溶点胶回收并纯化3.2kb酶切片段,按1μg/μl溶于0.1×TE中。
六、HBV全基因转染细胞及培养上清HBsAg和HBeAg的检测
同前报道[5]按10微克/皿全基因DNA通过磷酸钙沉淀转染HepG2细胞,每3天换液并收集上清,用Abbott试剂检测(EIA)上清HBsAg和HBeAg。
七、转染细胞内HBV DNA的Southern印迹检测
同参考文献[3]。
, http://www.100md.com
结果
一、二片段法和Gunther法扩增全基因效率的比较
用二片段法对18份保存1~5年的乙型肝炎患者血清标本扩增后,14份出现了1.15 kb和2.05 kb扩增片段,而用Gunther法仅有二份出现3.2 kb扩增产物。二片段法的扩增效率明显高于Gunther法。图1是分别用二种方法扩增同一例携带者血清标本的结果,通过条带密度定量分析,二片段的扩增产物比3.2 kb产物高20~40倍。
用酶切法和对HBV全基因克隆位点序列分析显示酶切位点和HBV插入序列与设计相符。
二、转染细胞培养上清中HBsAg和HBeAg的表达
分别用Abbott试剂检测转染细胞培养上清中HBsAg和HBeAg。图2、3分别显示用二片段法克隆的全基因转染HepG2细胞可有HBsAg和HBeAg的表达。由于该法扩增的HBV全基因的二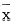
, 百拇医药
1:DNA分子量标志(1kb marker);2:Gunther法扩增产物;
3和4:二片段法扩增产物
图1 二片段法和Gunther法扩增HBV基因组产物的电泳图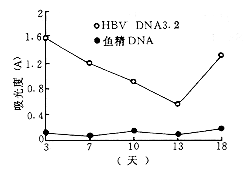
HBV DNA3.2:二片段法制备的SapⅠ粘末端HBV全基因;鱼精DNA:阴性对照
图2 用HBV全基因组转染HepG2细胞培养
上清HBsAg的表达
, http://www.100md.com
HBV DNA3.2:二片段法制备的SapⅠ粘末端HBV全基因;p3.8Ⅱ:插入1.2倍HBV全基因重组质粒(阳性对照);pCMVHBs:插入HBV S基因片段的重组质粒(HBeAg阴性对照);鱼精DNA:阴性对照 图3 用HBV全基因组转染HepG2细胞培养上清HBsAg的表达
端位于HBV C区读码框架起始点下游,故测及HBeAg提示二片段连接成的3.2kb全基因可在细胞内自身环化,并可有HBV复制。
Southern印迹杂交显示有小于3.2kb拖带杂交信号,反映获得的HBV全基因克隆可以在细胞内有复制过程。
讨论
HBV基因组的特殊结构使扩增全基因极为困难。应用本方法由于将HBV全基因组分为两个片段扩增,不要求模板为完整的3.2kb全基因组,降低了对模板的要求,提高了扩增效率,获得阳性PCR产物的机率增高。然而,由于HBV单链区的扩增有时会呈阴性,故应尽量采用较新鲜的血清。据Gunther报道用Expand High Fidelity(Boehinger Mannheim)的扩增系统扩增1fg的HBV DNA,当PCR产物为1μg时,人为造成的突变核苷酸数仅为8.3kb中1个核苷酸突变。因此,人为因素造成的突变率很低,但为保证扩增序列的可靠性,需用有校阅作用的酶作扩增。
, 百拇医药
在已发表的HBV全基因序列中,均有单一SpeⅠ酶切点,设计的引物PSP1和PSP2也均含有一个SpeⅠ酶切点,可将获得的HBV基因片段相互连接。SalⅠ为分子克隆中常用的限制性内切酶,而所发表的HBV全基因序列均无SalⅠ酶切点。引物中设计的SalⅠ酶切点有利于将扩增的片段克隆入载体中。由于SalⅠ和SpeⅠ酶切条件一致,便于对扩增片段作双酶切,简化了操作步骤。
SapⅠ的识别位点与酶切位点不同,故可通过用SapⅠ酶切克隆载体的HBV全基因而不改变HBV基因序列,从而获得完整的HBV全基因。在已发表的36株HBV全基因中,仅有2株序列中含SapⅠ酶切点。因此,用SapⅠ酶切HBV全基因克隆以获得HBV完整的全基因适用于大多数HBV毒株。用本文报道的方法已成功地克隆了患者血清HBV毒株全基因,并证明可在细胞中短暂表达抗原及在胞内复制。根据需要,还可对有意义的毒株进行全基因测序。这一新方法的建立将大大推进对HBV毒株的结构和功能的研究。
本课题为国家自然科学基金资助项目(39630020)
, 百拇医药
参考文献
1 Carman WF, Zanetti AR, Karayiannis P, et al. Vaccine-induced escape mutant of hepatitis B virus. Lancet,1990,336:325-329.
2 Sato S, Suzuki K, Akahane Y, et al. Hepatitis B virus strains with mutations in the core promoter in patients with fulminant hepatitis. Ann Intern Med,1995,122:241-246.
3 Gunther S, Li BC, Miska S, et al. A novel method for efficient amplification of whole hepatitis B virus genomes permits rapid functional analysis and reveals deletion mutant in immunosuppressed patients. J Virol,1995,69:5437-5444.
4 何建文,吕晴,朱启FDAC,等.乙型肝炎疫苗免疫失败病毒表面抗原‘a’决定簇基因变异的研究.中华实验和临床病毒学杂志,1996,10:308-311.
5 闻玉梅,何丽芳,楼惠珍,等.克隆乙型肝炎病毒DNA在HepG2细胞中的表达.中华医学杂志,1989,69:260-263., 百拇医药
单位:200032 上海 医科大学卫生部医学分子病毒学重点实验室
关键词:肝炎病毒,乙型;基因组;聚合酶链反应
中华传染病杂980203 【摘要】 目的 为从全基因水平研究乙型肝炎病毒变异与致病性的关系,建立血清标本经聚合酶链反应(PCR)加酶切方法扩增及克隆HBV全基因组的新方法。方法 设计了含SpeⅠ,SalⅠ及SapⅠ酶切点的引物,分别扩增1.15kb和2.05kb的单链及双链DNA区。经酶切及连接后获得HBV全基因克隆。结果 用新建立的方法直接从少量血清中克隆了HBV全基因,转染HepG2细胞可表达抗原并有胞内复制。结论 该法可用于对临床上HBV毒株的基因结构和功能的研究。
A novel method to study the full-length genome of hepatitis B virus He Jianwen, He Lifang, Yao Xin, et al. Department of Molecular Virology, Shanghai Medical University, Shanghai 200032
, http://www.100md.com
【Abstract】 Objective To study the association between pathogenicity and variants of hepatitis B virus(HBV) by full-length HBV genome, a novel method of amplification of the whole genome of HBV strains from serum samples of patients by PCR, followed by enzyme digestion and cloning was established. Methods Two sets of primers were designed: one with SpeⅠ restriction site, the other with restriction sites of SalⅠ and SapⅠ. PCR products were amplified separately from the single-stranded and the double-stranded regions of the genome, and the two PCR products obtained were 1.15kb and 2.05kb. After cleavage and ligation, the full-length HBV genome was used to transfect HepG2 cell line. Results Expression of viral antigens and intracellular replication of HBV were observed with the full-length HBV genome by the novel method. Conclusion This novel method could serve to study the structure and function of HBV genome from viral strains of clinical samples.
, http://www.100md.com
【Key words】 Hepatitis B virus Genome PCR
对乙型肝炎病毒(HBV)基因变异的研究提示,一些基因位点的变异可能与临床及流行病有关[1,2]。由于多数研究仅限于对某一基因片段的研究,而HBV基因内部有调控基因,基因片段间又相互重叠,研究单一片段难以说明问题。以往对HBV全基因的克隆需从大量血清中提取HBV,故难以对不同患者单一毒株作HBV全基因克隆。本研究根据Gunther等[3]的报道(Gunther法)并加以改进后,建立了一种聚合酶链反应(PCR)加酶切及酶连接后,从少量血清中一次克隆HBV全基因的方法(二片段法)。用获得的全基因转染细胞,可短期表达HBsAg和HBeAg并有胞内HBV复制。这一新方法为研究HBV毒株的基因结构与功能提供了可行的途径。
材料和方法
一、血清HBV DNA提取
, http://www.100md.com
方法见参考文献[4]。主要步骤:100μl血清加TES(10mmol/L Tris-HCl,pH8.0,5mmol/L EDTA,0.5%SDS,150μg/ml蛋白酶K),65°C 3小时,常规酚/氯仿提取,乙醇沉淀,DNA溶解于20μl双蒸水中。
二、引物设计
设计两对引物从不同方向分别扩增单链区及双链区,扩增片段分别为1.15kb和2.05kb长短。通过对已发表的36株HBV全基因组的同源及酶切点分析,选择含单一酶切点(SpeⅠ)的共有序列(nt669-699)分别合成正链和负链引物(PSP1和PSP2)。此外,根据我国HBVadr株存在HindⅢ酶切点但无SalⅠ酶切点,将Gunther设计的原引物酶切序列由HindⅢ改为SalⅠ,分别合成了P3S1和P3S2(nt1821-1841,nt1806-1825)。各引物序列为:
PSP15′-GGCTCAGTTTACTAGTGCCATTTGTTCA-
, 百拇医药
SpeⅠ
GTG-3′正链nt 669-699
PSP25′-CACTGAACAAATGGCACTAGTAAACTGA-
SalⅠ SapⅠ
GCC-3′负链nt 699-669
P3S1 5′-CCGGCGTCGACGAGCTCTTCTTTTTCACC-
SalⅠ SapⅠ
TCTGCCTAATCA-3′正链nt1821-1841
P3S2 5′-CCGGCGTCGACGAGCTCTTCAAAAAGTTGCATGGTGCTGG-3′负链nt1806-1825
, http://www.100md.com
三、PCR扩增
自血清标本提取的DNA分作二份模板,分别用P3S1和PSP2,P3S2和PSP1扩增,反应条件为100μl反应体积中包括引物各100pmol/L,4×dNTP各0.2mmol/L,MgCl2 1.5mmol/L,1×缓冲液及TaqDNA聚合酶2.5U(Gibco公司)。PCR于94°C 4分钟后,94°C 50秒,52°C 90秒,72°C90秒(每循环自动延伸10秒),30个循环后,于72°C延伸10分钟。
四、扩增产物的酶切及克隆
扩增产物经纯化后,用SalⅠ和SpeⅠ进行双酶切,同时用SalⅠ酶切载体pUC19。酶切后电泳并从凝胶中回收酶切片段,用连接酶(Promega公司)连接过夜后转化大肠杆菌JM109,37℃培养过夜,挑取白色菌落,提取质粒DNA,用酶切法及核苷酸自动测序鉴定。
, http://www.100md.com
五、HBV全基因片段的回收
经证实插入全基因片段的克隆转种于100ml培养液,用Qiagen midi(Qiagen公司)纯化质粒DNA,用SapⅠ(Gene-Lab公司)酶切(0.5U/μg DNA)16~20小时,低溶点胶回收并纯化3.2kb酶切片段,按1μg/μl溶于0.1×TE中。
六、HBV全基因转染细胞及培养上清HBsAg和HBeAg的检测
同前报道[5]按10微克/皿全基因DNA通过磷酸钙沉淀转染HepG2细胞,每3天换液并收集上清,用Abbott试剂检测(EIA)上清HBsAg和HBeAg。
七、转染细胞内HBV DNA的Southern印迹检测
同参考文献[3]。
, http://www.100md.com
结果
一、二片段法和Gunther法扩增全基因效率的比较
用二片段法对18份保存1~5年的乙型肝炎患者血清标本扩增后,14份出现了1.15 kb和2.05 kb扩增片段,而用Gunther法仅有二份出现3.2 kb扩增产物。二片段法的扩增效率明显高于Gunther法。图1是分别用二种方法扩增同一例携带者血清标本的结果,通过条带密度定量分析,二片段的扩增产物比3.2 kb产物高20~40倍。
用酶切法和对HBV全基因克隆位点序列分析显示酶切位点和HBV插入序列与设计相符。
二、转染细胞培养上清中HBsAg和HBeAg的表达
分别用Abbott试剂检测转染细胞培养上清中HBsAg和HBeAg。图2、3分别显示用二片段法克隆的全基因转染HepG2细胞可有HBsAg和HBeAg的表达。由于该法扩增的HBV全基因的二
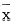
, 百拇医药
1:DNA分子量标志(1kb marker);2:Gunther法扩增产物;
3和4:二片段法扩增产物
图1 二片段法和Gunther法扩增HBV基因组产物的电泳图

HBV DNA3.2:二片段法制备的SapⅠ粘末端HBV全基因;鱼精DNA:阴性对照
图2 用HBV全基因组转染HepG2细胞培养
上清HBsAg的表达

, http://www.100md.com
HBV DNA3.2:二片段法制备的SapⅠ粘末端HBV全基因;p3.8Ⅱ:插入1.2倍HBV全基因重组质粒(阳性对照);pCMVHBs:插入HBV S基因片段的重组质粒(HBeAg阴性对照);鱼精DNA:阴性对照 图3 用HBV全基因组转染HepG2细胞培养上清HBsAg的表达
端位于HBV C区读码框架起始点下游,故测及HBeAg提示二片段连接成的3.2kb全基因可在细胞内自身环化,并可有HBV复制。
Southern印迹杂交显示有小于3.2kb拖带杂交信号,反映获得的HBV全基因克隆可以在细胞内有复制过程。
讨论
HBV基因组的特殊结构使扩增全基因极为困难。应用本方法由于将HBV全基因组分为两个片段扩增,不要求模板为完整的3.2kb全基因组,降低了对模板的要求,提高了扩增效率,获得阳性PCR产物的机率增高。然而,由于HBV单链区的扩增有时会呈阴性,故应尽量采用较新鲜的血清。据Gunther报道用Expand High Fidelity(Boehinger Mannheim)的扩增系统扩增1fg的HBV DNA,当PCR产物为1μg时,人为造成的突变核苷酸数仅为8.3kb中1个核苷酸突变。因此,人为因素造成的突变率很低,但为保证扩增序列的可靠性,需用有校阅作用的酶作扩增。
, 百拇医药
在已发表的HBV全基因序列中,均有单一SpeⅠ酶切点,设计的引物PSP1和PSP2也均含有一个SpeⅠ酶切点,可将获得的HBV基因片段相互连接。SalⅠ为分子克隆中常用的限制性内切酶,而所发表的HBV全基因序列均无SalⅠ酶切点。引物中设计的SalⅠ酶切点有利于将扩增的片段克隆入载体中。由于SalⅠ和SpeⅠ酶切条件一致,便于对扩增片段作双酶切,简化了操作步骤。
SapⅠ的识别位点与酶切位点不同,故可通过用SapⅠ酶切克隆载体的HBV全基因而不改变HBV基因序列,从而获得完整的HBV全基因。在已发表的36株HBV全基因中,仅有2株序列中含SapⅠ酶切点。因此,用SapⅠ酶切HBV全基因克隆以获得HBV完整的全基因适用于大多数HBV毒株。用本文报道的方法已成功地克隆了患者血清HBV毒株全基因,并证明可在细胞中短暂表达抗原及在胞内复制。根据需要,还可对有意义的毒株进行全基因测序。这一新方法的建立将大大推进对HBV毒株的结构和功能的研究。
本课题为国家自然科学基金资助项目(39630020)
, 百拇医药
参考文献
1 Carman WF, Zanetti AR, Karayiannis P, et al. Vaccine-induced escape mutant of hepatitis B virus. Lancet,1990,336:325-329.
2 Sato S, Suzuki K, Akahane Y, et al. Hepatitis B virus strains with mutations in the core promoter in patients with fulminant hepatitis. Ann Intern Med,1995,122:241-246.
3 Gunther S, Li BC, Miska S, et al. A novel method for efficient amplification of whole hepatitis B virus genomes permits rapid functional analysis and reveals deletion mutant in immunosuppressed patients. J Virol,1995,69:5437-5444.
4 何建文,吕晴,朱启FDAC,等.乙型肝炎疫苗免疫失败病毒表面抗原‘a’决定簇基因变异的研究.中华实验和临床病毒学杂志,1996,10:308-311.
5 闻玉梅,何丽芳,楼惠珍,等.克隆乙型肝炎病毒DNA在HepG2细胞中的表达.中华医学杂志,1989,69:260-263., 百拇医药