应用噬菌体随机肽库研究丙型肝炎病毒核心蛋白B细胞抗原表位
作者:杜勇 王海涛
单位:100071 北京,军事医学科学院微生物流行病研究所
关键词:肝炎病毒;丙型;病毒核心蛋白质类;抗原决定簇;噬菌体;肽;基因文库
中华微生物学和免疫学杂志990102 【 摘要 】 目的 探索利用特异多克隆抗体结合噬菌体递呈(phage display)随机肽库,进行病原体蛋白质的B细胞抗原表位研究。 方法 采取特异抗原→特异多克隆抗体→相应抗原表位的技术路线。所选研究对象为丙型肝炎病毒(HCV)核心蛋白。实验分三步:(1)抗HCV核心蛋白多克隆抗体的制备。用活化的Sepharose 4B耦联重组的HCV核心蛋白,亲和层析纯化丙型肝炎病人血清中特异的抗核心蛋白多克隆抗体。(2)随机肽库的生物淘洗(biopanning)。以纯化的多克隆抗体作为筛选分子,生物淘洗噬菌体递呈的随机七肽库。(3)阳性噬菌体克隆的鉴定。将筛选的噬菌体克隆进行ELISA检测、DNA测序以及竞争抑制试验等,最后分析所获资料。 结果 七肽氨基酸序列分析表明,HCV核心蛋白存在着至少3个B细胞抗原位点,其中19~25aa间(PQDVKFP)的线性表位是该蛋白的优势表位,证实了本研究策略的可行性。 结论 本技术路线可以有效地进行HCV核心蛋白的B细胞抗原表位研究。由此推论,此方法也可移植于其它病原微生物抗原或自身抗原的表位研究,继而为基于抗原表位水平的特异诊断试剂的研制、疫苗的设计提供依据。
, http://www.100md.com
Epitope mapping of polyclonal antibodies to HCV core protein using a phage peptide library DU Yong, WANG Haitao. Institute of Microbiology and Epidemiology, Beijing 100071
【 Abstract 】 Objective To design and testify a novel strategy for epitope mapping by using disease-specific polyclonal antibodies and phage peptide library. Methods The core protein of hepatitis C virus(HCV) was chosen as target protein. The strategy included three steps: (1) Preparation of specific anti-core polyclonal antibodies. The recombinant core protein of HCV was linked with CNBr-Sepharose 4B. The anti-core polyclonal antibodies in HCV-positive sera were purified by affinity chromatography. (2) Biopanning of a phage peptide library. By using the specific polyclonal antibodies as selective moleculars, a 7 meres phage peptide library was biopanned, and the positive clones were selected. (3) Identification of the epitopes: the positive clones were confirmed by ELISA , competition assay and DNA sequencing. Amino acid sequences of positive clones were compared with the aa sequence of HCV core protein. Results 31 positive clones were chosen and DNA sequenced. By experiments and sequence comparison, three epitopes were conformed in the N-terminal(1-125aa) of HCV core protein, and the linear epitope at residues 19-25 was the dominant epitope. These results agreed to other reports using overlapping peptides to make epitope mapping. Conclusions The designed strategy starting with target protein can be effectively used for epitope mapping, thus providing a new approach for some other pathogens specific epitope discovery and provide the potential for developing diagnostic reagents or vaccine.
, 百拇医药
【 Subject words 】 Hepatitis C virus Epitope mapping Phage peptide library Polyclonal antibodies HCV core protein
病原体的蛋白质抗原表位分析既是研究抗原分子的结构和功能、抗原-抗体反应机制的基础,又可为多肽疫苗研制、药物筛选以及特异血清学诊断方法的建立提供重要的线索和依据。分析抗原表位的传统方法主要是利用抗血清筛选重叠(overlapping)合成肽,但此方法存在几方面的缺陷〔1,2〕,首先是合成肽的成本高;其次是该方法的应用必须先确定蛋白质的氨基酸序列,才能合成相应的肽,而在实际研究中,许多蛋白质的序列并不清楚,故限制了此方法的应用;更主要的是,完整蛋白产生的抗血清中,抗构象型表位的抗体占90%,只有10%属线性表位抗体,而线性的合成肽仅能模拟蛋白质的线性表位,无疑会丢失众多的构象型表位信息。
近年来,一种新的生物技术——噬菌体递呈随机肽库(random peptide libraries,RPL)被逐渐用于蛋白质抗原表位分析。这种技术弥补了合成肽筛选的不足〔3,4〕,在研究线性短肽、折叠的蛋白质,甚至非蛋白分子等受体的相应配体时,噬菌体递呈的随机肽库被认为是配体的“万能库”。这些配体不一定和天然的配体完全相同,但是能模拟天然配体的结合特性(mimotope)。基于此理论,在抗原-抗体的识别研究中,不必源于天然抗原,就可从随机肽库中直接筛选出能和抗体结合的表位,这些表位既可以是线性表位,也可以是模拟的构象型表位。而且,此种方法不必预先确定蛋白质的氨基酸序列。
, 百拇医药
本研究中,我们设计了利用噬菌体递呈随机肽库结合特异多克隆抗体研究蛋白质B细胞抗原表位的技术路线:首先用具有抗原活性的HCV核心蛋白耦联Sepharose 4B,亲和层析纯化丙型肝炎病人血清中特异的抗HCV核心蛋白多克隆抗体。再以此多克隆抗体作为受体分子,筛选噬菌体递呈的随机七肽库,进行了HCV核心蛋白B细胞抗原表位的研究。
材料与方法
1.随机肽库:噬菌体递呈随机肽库(Ph.D.TMphage peptide library)购自美国NEW ENGLAND Biolabs公司。肽库的滴度为2×1013PFU/ml,随机多样性为2×109。受体菌为E.coli ER2537。测序引物(-28 gⅢsequencing primer):5'-GTA TGG GAT TTT GCT AAA CAA C-3'。
2.试验用血清:采自河北省固安县的4名慢性HCV携带者,每人静脉取血200ml,分离血清,-20℃冻存。经ELISA鉴定,抗HCV抗体均阳性。
, 百拇医药
3.主要试剂:CNBr-Sepharose 4B购自Pharmacia公司。HCV核心蛋白(P19)由本实验室在大肠杆菌中表达、纯化。HRP标记兔抗M13噬菌体IgG抗体、HRP标记羊抗人IgG抗体由本所制备、标记。合成肽CP9(5~46aa)由本室唐时幸博士赠送。
4.亲和层析纯化特异抗体:首先以50%,33%的饱和硫酸铵分步沉淀400ml丙型肝炎病人血清中的IgG抗体。将提取的IgG溶液混悬已耦联HCV核心蛋白P19的Sepharose 4B,室温下充分亲和吸附。将样品装柱,PBS洗去未结合的蛋白。用0.2mol/L甘氨酸-盐酸缓冲液(pH2.2)洗脱结合的特异蛋白。SDS-PAGE分析洗脱液,并按以下公式计算蛋白浓度:
蛋白浓度(mg/ml)=A280×1.55-A260×0.74
5.肽库的生物淘洗(biopanning):将抗HCV核心蛋白多克隆抗体稀释至150μg/ml,每孔150μl包被微孔,3%BSA封闭。用300μl TBST缓冲液稀释10μl肽库原液,按100μl/孔加到微孔内,室温缓慢摇荡1小时。TBST缓冲液洗涤10次。每孔加入100μl 0.2mol/L甘氨酸-盐酸(pH2.2),室温10分钟洗脱结合的噬菌体。加入20μl 2mol/L Tris中和洗脱液。测定洗脱液噬菌体滴度。余下的洗脱液加到20ml E.coli ER2537培养液中(A600=0.5~1.0)。37℃ 250r/min培养5小时。制备繁殖后的噬菌体,并测定滴度。计算噬菌体产量:%产率=(洗脱噬菌体数÷淘洗用噬菌体数)×100%。按上述步骤再淘洗2次。第三次淘洗的噬菌体感染E.coli ER2537后,铺制平板。挑取单个噬斑,制备原种。经ELISA鉴定阳性克隆。对阳性克隆提取单链DNA,测序。
, 百拇医药
6.噬菌体的ELISA检测:抗HCV核心蛋白多克隆抗体包被酶联板。3%BSA封闭后,加入稀释液和噬菌体原种各50μl,37℃孵育1小时,洗涤缓冲液洗板3次。加入1∶1000稀释的酶标兔抗M13噬菌体抗体,37℃反应1小时。OPD显色,测定吸光度A490值。
7.竞争试验:将制备的噬菌体稀释至1011/ml,包被酶联板。3%BSA封闭。将合成肽CP9(20μg/ml)先与125I标记的抗HCV核心抗体在37℃孵育1小时,再加到封闭后的酶联板孔中,37℃作用1小时,洗涤,测定cpm。计算抑制率:
抑制率=(抑制前cpm-抑制后cpm)/抑制前cpm×100%
8.序列测定:采取Sanger双脱氧链终止法测序。提取噬菌体单链DNA,测序程序按试剂盒说明书操作。
结 果
, 百拇医药
1.抗HCV核心蛋白多克隆抗体的制备与特异性检测:本试验所用的HCV重组核心蛋白为在大肠杆菌中表达,经Ni-NTA层析纯化、自然复性,纯度达到电泳级,ELISA检测可以和丙型肝炎病人血清发生特异反应。
经定量测定,用上述HCV核心蛋白从400ml HCV感染者血清中亲和层析纯化了1.86mg抗HCV核心蛋白抗体。与血清中抗体滴度相比,此特异多克隆抗体大约浓缩了3200倍。
ELISA方法比较制备的HCV核心抗体、艾滋病人IgG抗体、静脉用混合人IgG抗体与P19蛋白的免疫反应。从图1可以看出,纯化的抗体和P19的反应呈高度特异性,稀释至7.4ng/ml仍有很强的反应,而对照用的两种抗体和P19基本无反应。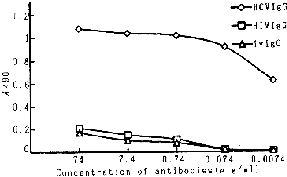
图1 三种抗体与P19蛋白的ELISA反应
, 百拇医药
Fig 1. Immunoreaction of P19 with three kinds of antibodies by ELISA
2.阳性噬菌体克隆的筛选:每次淘洗均加入2×1011的噬菌体,并滴定洗脱下的噬菌体数量。表1显示,淘洗中有较高程度的富集(enrichment),第三轮淘洗比第一轮淘洗的产量高出近百倍。
表1 噬菌体肽库淘洗产量(%)
Table 1. Yields(%) of washed phages by biopanning No.of
biopanning
Phages added
Phages washed
, 百拇医药
Yield(%)
1
2×1011
7.2×106
3.6×10-5
2
2×1011
4.4×107
2.2×10-4
3
2×1011
, http://www.100md.com
3.6×108
1.8×10-3
第三轮淘洗下的噬菌体感染细菌后,随机挑取79个噬菌斑,制备原种,ELISA检测其与上述三种抗体的反应性,并用淘洗前的肽库和辅助噬菌体VCM13原种作阴性对照。相对两种阴性对照的阳性率分别为87.3%(69/79)和94.9%(75/79)。另外,在包被的3种抗体中,噬菌体特异地与抗HCV核心抗体发生反应,呈高度的特异性。
3.阳性噬菌体DNA序列测定及编码氨基酸序列分析:测定了31株阳性噬菌体克隆的递呈DNA片段,推导这些DNA序列编码的氨基酸残基,即为噬菌体递呈的七肽序列(图2)。依据氨基酸序列特征,可以将31个阳性噬菌体递呈的七肽序列分为五种类型:第一种类型的序列模式为xQxV(I)xFP,此类型数量最多,占总数的77%(24/31)其组成特点为第二位的氨基酸(P36为第一位)均为谷氨酰胺、第四位为芳香族氨基酸的缬氨酸或异亮氨酸、第六、七位氨基酸(P36 为五、六位)分别为苯丙氨酸和脯氨酸。将这24个氨基酸序列和HCV核心蛋白比较,其和核心蛋白第19~25位氨基酸序列(PQDVKFP)有很高的同源性,说明该区域存在一个优势线性表位。第二种类型(P25/P48/P68)为SxxxQYV,占10%(3/31);第三种类型(P11/P51)为WYQLPLR,占6%(2/31);第四种类型(P35)为HQSL RWA;第五种类型(P39)为ANSLLIT,后两种类型各占3%(1/31)。后四种类型的序列和HCV核心蛋白无明显的同源序列,这些序列所递呈的表位可能有两种情况:一是这些序列为线性表位的模拟位(mimotope),即这些序列的空间构型与某线性表位的空间构型相近,以致引起了特异的血清学交叉反应;另一种情况是这些表位可能为蛋白质抗原的非连续性表位,即在蛋白质一级结构上不连续,而在高级结构上却毗邻的氨基酸残基所组成的表位。这些模拟位可能均为非优势表位。
, 百拇医药
图2 阳性噬菌体递呈七肽氨基酸序列比较
Fig 2.Comparison of sequences of amino acid displayed by phages
4.测序噬菌体克隆的抗原性检测:ELISA检测上述测序的噬菌体克隆与抗 HCV核心蛋白多克隆抗体、艾滋病人IgG抗体、静脉用人IgG抗体的反应(图3),阳性噬菌体均可与HCV核心抗体发生特异反应,而与其它两种抗体无明显反应,进一步证明了淘洗的特异性。
图3 阳性噬菌体与三种抗体的ELISA反应(A490)
Fig 3. Immunoreaction of positive-phages with three kinds of antibodies by ELISA(A490)
, 百拇医药
5.HCV核心蛋白优势表位(P19~25)氨基酸变异频数分布:在测定的HCV核心蛋白优势表位(P19~25)的24个序列中,除第二位(Q)、第六位(F)和第七位(P)氨基酸保守外,其余位置的氨基酸均发生了变异,但这些变异的氨基酸并不是随机发生的,每一位置的变异氨基酸分布仍有一定的聚集性。
6.竞争试验:用合成肽CP9和8个克隆的噬菌体同125I标记的抗HCV核心抗体进行竞争试验(图4)。可以看出,抑制率最高且相近的4个样品的序列,均为与CP9有高同源性的序列,即xQxDVxFP;CP9对P11和P25的抑制率最低,对P35和P39的抑制率相近。这一结果进一步显示上述序列比较分析的正确性,同时也说明,HCV核心蛋白至少有3个以上B细胞表位。
图4 合成肽CP9与阳性噬菌体的竞争抑制
, http://www.100md.com
Fig 4. Competition of peptide CP9 with positive-phages by ELISA
The inhibit rates are 37.8%(P11),81.4%(P16),75.4%
(P18),72.3%(P20),66.6%(P24),32.6%(P25),57.2%(P35),60.9%(P39),respectively
讨 论
对很多人类的过敏性疾病、传染性疾病以及自身免疫性疾病而言,人体内存在着种类繁多的、针对过敏原、病原微生物或自身抗原的抗体,对这众多抗体的相应表位研究和分析可以为阐明病原体性质及病原体与人体的关系,进而研制相应的疫苗和诊断试剂提供基础资料。1994年,Folgori〔4〕首先进行了实验性尝试:实验者将乙型肝炎病毒表面抗原(HBsAg)免疫人体,待产生高滴度抗体后,用血清作筛选分子,从随机肽库中筛选出了两个抗原表位,证实了利用随机肽库研究血清中某种病原体蛋白质抗原表位的可行性。随后,Dybwad〔5〕也进行了相似的试验:将17个氨基酸的多肽免疫兔后,从兔血清中筛选出了特异的抗原表位。除以上人为的实验研究外,有的学者亦将这种实验方法用于自然感染的病人血清中的抗原表位分析,取得了较满意的结果〔6,7〕。
, http://www.100md.com
分析上述试验途径不难发现,在实验技术中存在着一个很大的障碍:由于病人血清中抗体种类繁多,且滴度高低不同,单纯用血清作筛选分子进行生物淘洗时,不可避免地同时使大量的非特异噬菌体也随之“富集”,进而掩盖了特异噬菌体的筛选〔3〕。尤其对一些免疫原性差的病原体,如丙型肝炎病毒,其引起的宿主免疫反应很弱,相应的抗体滴度低,在血清抗体中所占的比例很小,筛选这类病原体的抗原表位难度很大。为克服这一障碍,我们选择HCV核心蛋白为研究对象,设计了一条从抗原出发,利用噬菌体递呈随机肽库结合特异多克隆抗体,研究B细胞抗原表位的技术路线:首先以HCV核心蛋白亲和层析纯化了病人血清中特异的抗HCV核心蛋白多克隆抗体(此步骤可以极大地浓缩特异抗体,本实验中,特异抗体浓缩了大约3200倍,并同时去除了非特异抗体),然后再以这些特异的多克隆抗体作为筛选分子,对随机肽库进行了生物淘洗和免疫学检测,最后获得了能和筛选用抗体特异结合的递呈多肽序列。和同类研究相比,Prezzi等〔7〕用抗血清直接淘洗肽库,筛选的阳性率仅为千分之二,而我们在试验中,首先以亲和层析纯化了特异抗体,这一方面浓缩了所需的多克隆抗体,同时又去除了无关抗体的干扰,其结果大大提高了筛选随机肽库的敏感性和特异性,经过3轮淘洗,筛选的阳性率可达87.3%。
, 百拇医药
HCV核心蛋白是病毒的主要结构蛋白,能引起宿主较强的体液免疫反应。以往的研究表明,HCV核心蛋白的氨基端(1~125aa)存在着多个6~10肽的序列依赖性及构象依赖性抗原表位〔8-12〕。因此,该蛋白是检测HCV感染的主要诊断试剂。大肠杆菌表达的HCV核心蛋白是目前国内外组装试剂盒的主要试剂之一,我们表达的核心蛋白经纯化、复性后,能和丙型肝炎病人血清特异反应,而和其它血清,如艾滋病人血清抗体、正常人血清抗体无明显反应,说明其已接近正确折叠,具有天然HCV核心蛋白的抗原性,而受非天然蛋白结构的影响不大。显然,用这种蛋白进行表位研究,如前言中所述,应明显优于合成肽筛选。
本实验结果表明,我们所用的这条技术路线能有效地进行病人血清中抗蛋白质多克隆抗体的相应表位分析。可以推论,此方法也可移植用于众多的已知病原体蛋白质的抗原表位研究,为其基于抗原表位水平的特异诊断试剂、疫苗的研制打下基础。
本课题受国家自然科学基金(No.39630020)和总后重点基金(No.9608030)资助
, 百拇医药
参考文献
[1] Cason J.Strategies for mapping and imitating viral B-cell epitopes.J Virol Methods,1994,49∶209-220.
[2] Van Regenmortel MHV.Mapping epitope structure and activity:from one-dimensional prediction to four-dimensional description of antigenic specificity.Methods:A companion to Methods in Enzymology,1996,9∶465-472.
[3] Daniels DA,Lane DP.Phage peptide libraries.Methods Enzymol,1996,9∶494-507.
, 百拇医药
[4] Folgori A,Tafi R,Meola A,et al.A general strategy to identify mimotopes of pathological antigens using only random peptide libraries and human sera.EMBO J,1994,13(9)∶2236-2243.
[5] Dybwad A,Bogen B,Natvig JB,et al.peptide phage libraries can be an efficient tool for identifying antibody ligands for polyclonal antisera.Clin Exp Immunol,1995,102∶438-442.
[6] Germaschewski V,Murray K.Identification of polyclonal serum specificities with phagedisplay libraries.J Virol Methods,1996,58∶21-32.
, 百拇医药
[7] Prezzi C,Nuzzo M, Meola A,et al.Selection of antigenic and immunogenic mimics of hepatitis C virus using sera from patients.J Immunol,1996,4 504-4 513.
[8] Sallberg M,Pumpen P,Zhang ZX,et al.Locations of antibody binding sites within conserved regions of the HCV core protein.J Med Virol,1994,43∶62-68.
[9] Kotwal GJ,Baroudy BM,Kuramoto IK,et al.Detection of acute hepatitis C virus infection by ELISA using a synthetic peptide comprising a structural epitope.Proc Natl Acad Sci USA,1992,89∶4 486-4 489.
, 百拇医药
[10] Okamoto H,Tsuda F,Machida A,et al.Antibodies against synthetic oligopeptides deduced from the putative core gene for the diagnosis of HCV infection.Hepatology,1992,15∶180-186.
[11] Nasoff MS,Zebedee SL,Inchauspe G,et al.Identification of an immunodominant epitope within the capsid protein of HCV.Proc Natl Acad Sci USA,1991,88∶5 462-5 466.
[12] Goeser T,Muller HM,Ye J,et al.Characterization of antigenic determinants in the core antigen of the hepatitis C virus.virology,1994,205∶462-469.
(收稿:1997-08-07 修回:1998-03-12), 百拇医药
单位:100071 北京,军事医学科学院微生物流行病研究所
关键词:肝炎病毒;丙型;病毒核心蛋白质类;抗原决定簇;噬菌体;肽;基因文库
中华微生物学和免疫学杂志990102 【 摘要 】 目的 探索利用特异多克隆抗体结合噬菌体递呈(phage display)随机肽库,进行病原体蛋白质的B细胞抗原表位研究。 方法 采取特异抗原→特异多克隆抗体→相应抗原表位的技术路线。所选研究对象为丙型肝炎病毒(HCV)核心蛋白。实验分三步:(1)抗HCV核心蛋白多克隆抗体的制备。用活化的Sepharose 4B耦联重组的HCV核心蛋白,亲和层析纯化丙型肝炎病人血清中特异的抗核心蛋白多克隆抗体。(2)随机肽库的生物淘洗(biopanning)。以纯化的多克隆抗体作为筛选分子,生物淘洗噬菌体递呈的随机七肽库。(3)阳性噬菌体克隆的鉴定。将筛选的噬菌体克隆进行ELISA检测、DNA测序以及竞争抑制试验等,最后分析所获资料。 结果 七肽氨基酸序列分析表明,HCV核心蛋白存在着至少3个B细胞抗原位点,其中19~25aa间(PQDVKFP)的线性表位是该蛋白的优势表位,证实了本研究策略的可行性。 结论 本技术路线可以有效地进行HCV核心蛋白的B细胞抗原表位研究。由此推论,此方法也可移植于其它病原微生物抗原或自身抗原的表位研究,继而为基于抗原表位水平的特异诊断试剂的研制、疫苗的设计提供依据。
, http://www.100md.com
Epitope mapping of polyclonal antibodies to HCV core protein using a phage peptide library DU Yong, WANG Haitao. Institute of Microbiology and Epidemiology, Beijing 100071
【 Abstract 】 Objective To design and testify a novel strategy for epitope mapping by using disease-specific polyclonal antibodies and phage peptide library. Methods The core protein of hepatitis C virus(HCV) was chosen as target protein. The strategy included three steps: (1) Preparation of specific anti-core polyclonal antibodies. The recombinant core protein of HCV was linked with CNBr-Sepharose 4B. The anti-core polyclonal antibodies in HCV-positive sera were purified by affinity chromatography. (2) Biopanning of a phage peptide library. By using the specific polyclonal antibodies as selective moleculars, a 7 meres phage peptide library was biopanned, and the positive clones were selected. (3) Identification of the epitopes: the positive clones were confirmed by ELISA , competition assay and DNA sequencing. Amino acid sequences of positive clones were compared with the aa sequence of HCV core protein. Results 31 positive clones were chosen and DNA sequenced. By experiments and sequence comparison, three epitopes were conformed in the N-terminal(1-125aa) of HCV core protein, and the linear epitope at residues 19-25 was the dominant epitope. These results agreed to other reports using overlapping peptides to make epitope mapping. Conclusions The designed strategy starting with target protein can be effectively used for epitope mapping, thus providing a new approach for some other pathogens specific epitope discovery and provide the potential for developing diagnostic reagents or vaccine.
, 百拇医药
【 Subject words 】 Hepatitis C virus Epitope mapping Phage peptide library Polyclonal antibodies HCV core protein
病原体的蛋白质抗原表位分析既是研究抗原分子的结构和功能、抗原-抗体反应机制的基础,又可为多肽疫苗研制、药物筛选以及特异血清学诊断方法的建立提供重要的线索和依据。分析抗原表位的传统方法主要是利用抗血清筛选重叠(overlapping)合成肽,但此方法存在几方面的缺陷〔1,2〕,首先是合成肽的成本高;其次是该方法的应用必须先确定蛋白质的氨基酸序列,才能合成相应的肽,而在实际研究中,许多蛋白质的序列并不清楚,故限制了此方法的应用;更主要的是,完整蛋白产生的抗血清中,抗构象型表位的抗体占90%,只有10%属线性表位抗体,而线性的合成肽仅能模拟蛋白质的线性表位,无疑会丢失众多的构象型表位信息。
近年来,一种新的生物技术——噬菌体递呈随机肽库(random peptide libraries,RPL)被逐渐用于蛋白质抗原表位分析。这种技术弥补了合成肽筛选的不足〔3,4〕,在研究线性短肽、折叠的蛋白质,甚至非蛋白分子等受体的相应配体时,噬菌体递呈的随机肽库被认为是配体的“万能库”。这些配体不一定和天然的配体完全相同,但是能模拟天然配体的结合特性(mimotope)。基于此理论,在抗原-抗体的识别研究中,不必源于天然抗原,就可从随机肽库中直接筛选出能和抗体结合的表位,这些表位既可以是线性表位,也可以是模拟的构象型表位。而且,此种方法不必预先确定蛋白质的氨基酸序列。
, 百拇医药
本研究中,我们设计了利用噬菌体递呈随机肽库结合特异多克隆抗体研究蛋白质B细胞抗原表位的技术路线:首先用具有抗原活性的HCV核心蛋白耦联Sepharose 4B,亲和层析纯化丙型肝炎病人血清中特异的抗HCV核心蛋白多克隆抗体。再以此多克隆抗体作为受体分子,筛选噬菌体递呈的随机七肽库,进行了HCV核心蛋白B细胞抗原表位的研究。
材料与方法
1.随机肽库:噬菌体递呈随机肽库(Ph.D.TMphage peptide library)购自美国NEW ENGLAND Biolabs公司。肽库的滴度为2×1013PFU/ml,随机多样性为2×109。受体菌为E.coli ER2537。测序引物(-28 gⅢsequencing primer):5'-GTA TGG GAT TTT GCT AAA CAA C-3'。
2.试验用血清:采自河北省固安县的4名慢性HCV携带者,每人静脉取血200ml,分离血清,-20℃冻存。经ELISA鉴定,抗HCV抗体均阳性。
, 百拇医药
3.主要试剂:CNBr-Sepharose 4B购自Pharmacia公司。HCV核心蛋白(P19)由本实验室在大肠杆菌中表达、纯化。HRP标记兔抗M13噬菌体IgG抗体、HRP标记羊抗人IgG抗体由本所制备、标记。合成肽CP9(5~46aa)由本室唐时幸博士赠送。
4.亲和层析纯化特异抗体:首先以50%,33%的饱和硫酸铵分步沉淀400ml丙型肝炎病人血清中的IgG抗体。将提取的IgG溶液混悬已耦联HCV核心蛋白P19的Sepharose 4B,室温下充分亲和吸附。将样品装柱,PBS洗去未结合的蛋白。用0.2mol/L甘氨酸-盐酸缓冲液(pH2.2)洗脱结合的特异蛋白。SDS-PAGE分析洗脱液,并按以下公式计算蛋白浓度:
蛋白浓度(mg/ml)=A280×1.55-A260×0.74
5.肽库的生物淘洗(biopanning):将抗HCV核心蛋白多克隆抗体稀释至150μg/ml,每孔150μl包被微孔,3%BSA封闭。用300μl TBST缓冲液稀释10μl肽库原液,按100μl/孔加到微孔内,室温缓慢摇荡1小时。TBST缓冲液洗涤10次。每孔加入100μl 0.2mol/L甘氨酸-盐酸(pH2.2),室温10分钟洗脱结合的噬菌体。加入20μl 2mol/L Tris中和洗脱液。测定洗脱液噬菌体滴度。余下的洗脱液加到20ml E.coli ER2537培养液中(A600=0.5~1.0)。37℃ 250r/min培养5小时。制备繁殖后的噬菌体,并测定滴度。计算噬菌体产量:%产率=(洗脱噬菌体数÷淘洗用噬菌体数)×100%。按上述步骤再淘洗2次。第三次淘洗的噬菌体感染E.coli ER2537后,铺制平板。挑取单个噬斑,制备原种。经ELISA鉴定阳性克隆。对阳性克隆提取单链DNA,测序。
, 百拇医药
6.噬菌体的ELISA检测:抗HCV核心蛋白多克隆抗体包被酶联板。3%BSA封闭后,加入稀释液和噬菌体原种各50μl,37℃孵育1小时,洗涤缓冲液洗板3次。加入1∶1000稀释的酶标兔抗M13噬菌体抗体,37℃反应1小时。OPD显色,测定吸光度A490值。
7.竞争试验:将制备的噬菌体稀释至1011/ml,包被酶联板。3%BSA封闭。将合成肽CP9(20μg/ml)先与125I标记的抗HCV核心抗体在37℃孵育1小时,再加到封闭后的酶联板孔中,37℃作用1小时,洗涤,测定cpm。计算抑制率:
抑制率=(抑制前cpm-抑制后cpm)/抑制前cpm×100%
8.序列测定:采取Sanger双脱氧链终止法测序。提取噬菌体单链DNA,测序程序按试剂盒说明书操作。
结 果
, 百拇医药
1.抗HCV核心蛋白多克隆抗体的制备与特异性检测:本试验所用的HCV重组核心蛋白为在大肠杆菌中表达,经Ni-NTA层析纯化、自然复性,纯度达到电泳级,ELISA检测可以和丙型肝炎病人血清发生特异反应。
经定量测定,用上述HCV核心蛋白从400ml HCV感染者血清中亲和层析纯化了1.86mg抗HCV核心蛋白抗体。与血清中抗体滴度相比,此特异多克隆抗体大约浓缩了3200倍。
ELISA方法比较制备的HCV核心抗体、艾滋病人IgG抗体、静脉用混合人IgG抗体与P19蛋白的免疫反应。从图1可以看出,纯化的抗体和P19的反应呈高度特异性,稀释至7.4ng/ml仍有很强的反应,而对照用的两种抗体和P19基本无反应。

图1 三种抗体与P19蛋白的ELISA反应
, 百拇医药
Fig 1. Immunoreaction of P19 with three kinds of antibodies by ELISA
2.阳性噬菌体克隆的筛选:每次淘洗均加入2×1011的噬菌体,并滴定洗脱下的噬菌体数量。表1显示,淘洗中有较高程度的富集(enrichment),第三轮淘洗比第一轮淘洗的产量高出近百倍。
表1 噬菌体肽库淘洗产量(%)
Table 1. Yields(%) of washed phages by biopanning No.of
biopanning
Phages added
Phages washed
, 百拇医药
Yield(%)
1
2×1011
7.2×106
3.6×10-5
2
2×1011
4.4×107
2.2×10-4
3
2×1011
, http://www.100md.com
3.6×108
1.8×10-3
第三轮淘洗下的噬菌体感染细菌后,随机挑取79个噬菌斑,制备原种,ELISA检测其与上述三种抗体的反应性,并用淘洗前的肽库和辅助噬菌体VCM13原种作阴性对照。相对两种阴性对照的阳性率分别为87.3%(69/79)和94.9%(75/79)。另外,在包被的3种抗体中,噬菌体特异地与抗HCV核心抗体发生反应,呈高度的特异性。
3.阳性噬菌体DNA序列测定及编码氨基酸序列分析:测定了31株阳性噬菌体克隆的递呈DNA片段,推导这些DNA序列编码的氨基酸残基,即为噬菌体递呈的七肽序列(图2)。依据氨基酸序列特征,可以将31个阳性噬菌体递呈的七肽序列分为五种类型:第一种类型的序列模式为xQxV(I)xFP,此类型数量最多,占总数的77%(24/31)其组成特点为第二位的氨基酸(P36为第一位)均为谷氨酰胺、第四位为芳香族氨基酸的缬氨酸或异亮氨酸、第六、七位氨基酸(P36 为五、六位)分别为苯丙氨酸和脯氨酸。将这24个氨基酸序列和HCV核心蛋白比较,其和核心蛋白第19~25位氨基酸序列(PQDVKFP)有很高的同源性,说明该区域存在一个优势线性表位。第二种类型(P25/P48/P68)为SxxxQYV,占10%(3/31);第三种类型(P11/P51)为WYQLPLR,占6%(2/31);第四种类型(P35)为HQSL RWA;第五种类型(P39)为ANSLLIT,后两种类型各占3%(1/31)。后四种类型的序列和HCV核心蛋白无明显的同源序列,这些序列所递呈的表位可能有两种情况:一是这些序列为线性表位的模拟位(mimotope),即这些序列的空间构型与某线性表位的空间构型相近,以致引起了特异的血清学交叉反应;另一种情况是这些表位可能为蛋白质抗原的非连续性表位,即在蛋白质一级结构上不连续,而在高级结构上却毗邻的氨基酸残基所组成的表位。这些模拟位可能均为非优势表位。

, 百拇医药
图2 阳性噬菌体递呈七肽氨基酸序列比较
Fig 2.Comparison of sequences of amino acid displayed by phages
4.测序噬菌体克隆的抗原性检测:ELISA检测上述测序的噬菌体克隆与抗 HCV核心蛋白多克隆抗体、艾滋病人IgG抗体、静脉用人IgG抗体的反应(图3),阳性噬菌体均可与HCV核心抗体发生特异反应,而与其它两种抗体无明显反应,进一步证明了淘洗的特异性。

图3 阳性噬菌体与三种抗体的ELISA反应(A490)
Fig 3. Immunoreaction of positive-phages with three kinds of antibodies by ELISA(A490)
, 百拇医药
5.HCV核心蛋白优势表位(P19~25)氨基酸变异频数分布:在测定的HCV核心蛋白优势表位(P19~25)的24个序列中,除第二位(Q)、第六位(F)和第七位(P)氨基酸保守外,其余位置的氨基酸均发生了变异,但这些变异的氨基酸并不是随机发生的,每一位置的变异氨基酸分布仍有一定的聚集性。
6.竞争试验:用合成肽CP9和8个克隆的噬菌体同125I标记的抗HCV核心抗体进行竞争试验(图4)。可以看出,抑制率最高且相近的4个样品的序列,均为与CP9有高同源性的序列,即xQxDVxFP;CP9对P11和P25的抑制率最低,对P35和P39的抑制率相近。这一结果进一步显示上述序列比较分析的正确性,同时也说明,HCV核心蛋白至少有3个以上B细胞表位。

图4 合成肽CP9与阳性噬菌体的竞争抑制
, http://www.100md.com
Fig 4. Competition of peptide CP9 with positive-phages by ELISA
The inhibit rates are 37.8%(P11),81.4%(P16),75.4%
(P18),72.3%(P20),66.6%(P24),32.6%(P25),57.2%(P35),60.9%(P39),respectively
讨 论
对很多人类的过敏性疾病、传染性疾病以及自身免疫性疾病而言,人体内存在着种类繁多的、针对过敏原、病原微生物或自身抗原的抗体,对这众多抗体的相应表位研究和分析可以为阐明病原体性质及病原体与人体的关系,进而研制相应的疫苗和诊断试剂提供基础资料。1994年,Folgori〔4〕首先进行了实验性尝试:实验者将乙型肝炎病毒表面抗原(HBsAg)免疫人体,待产生高滴度抗体后,用血清作筛选分子,从随机肽库中筛选出了两个抗原表位,证实了利用随机肽库研究血清中某种病原体蛋白质抗原表位的可行性。随后,Dybwad〔5〕也进行了相似的试验:将17个氨基酸的多肽免疫兔后,从兔血清中筛选出了特异的抗原表位。除以上人为的实验研究外,有的学者亦将这种实验方法用于自然感染的病人血清中的抗原表位分析,取得了较满意的结果〔6,7〕。
, http://www.100md.com
分析上述试验途径不难发现,在实验技术中存在着一个很大的障碍:由于病人血清中抗体种类繁多,且滴度高低不同,单纯用血清作筛选分子进行生物淘洗时,不可避免地同时使大量的非特异噬菌体也随之“富集”,进而掩盖了特异噬菌体的筛选〔3〕。尤其对一些免疫原性差的病原体,如丙型肝炎病毒,其引起的宿主免疫反应很弱,相应的抗体滴度低,在血清抗体中所占的比例很小,筛选这类病原体的抗原表位难度很大。为克服这一障碍,我们选择HCV核心蛋白为研究对象,设计了一条从抗原出发,利用噬菌体递呈随机肽库结合特异多克隆抗体,研究B细胞抗原表位的技术路线:首先以HCV核心蛋白亲和层析纯化了病人血清中特异的抗HCV核心蛋白多克隆抗体(此步骤可以极大地浓缩特异抗体,本实验中,特异抗体浓缩了大约3200倍,并同时去除了非特异抗体),然后再以这些特异的多克隆抗体作为筛选分子,对随机肽库进行了生物淘洗和免疫学检测,最后获得了能和筛选用抗体特异结合的递呈多肽序列。和同类研究相比,Prezzi等〔7〕用抗血清直接淘洗肽库,筛选的阳性率仅为千分之二,而我们在试验中,首先以亲和层析纯化了特异抗体,这一方面浓缩了所需的多克隆抗体,同时又去除了无关抗体的干扰,其结果大大提高了筛选随机肽库的敏感性和特异性,经过3轮淘洗,筛选的阳性率可达87.3%。
, 百拇医药
HCV核心蛋白是病毒的主要结构蛋白,能引起宿主较强的体液免疫反应。以往的研究表明,HCV核心蛋白的氨基端(1~125aa)存在着多个6~10肽的序列依赖性及构象依赖性抗原表位〔8-12〕。因此,该蛋白是检测HCV感染的主要诊断试剂。大肠杆菌表达的HCV核心蛋白是目前国内外组装试剂盒的主要试剂之一,我们表达的核心蛋白经纯化、复性后,能和丙型肝炎病人血清特异反应,而和其它血清,如艾滋病人血清抗体、正常人血清抗体无明显反应,说明其已接近正确折叠,具有天然HCV核心蛋白的抗原性,而受非天然蛋白结构的影响不大。显然,用这种蛋白进行表位研究,如前言中所述,应明显优于合成肽筛选。
本实验结果表明,我们所用的这条技术路线能有效地进行病人血清中抗蛋白质多克隆抗体的相应表位分析。可以推论,此方法也可移植用于众多的已知病原体蛋白质的抗原表位研究,为其基于抗原表位水平的特异诊断试剂、疫苗的研制打下基础。
本课题受国家自然科学基金(No.39630020)和总后重点基金(No.9608030)资助
, 百拇医药
参考文献
[1] Cason J.Strategies for mapping and imitating viral B-cell epitopes.J Virol Methods,1994,49∶209-220.
[2] Van Regenmortel MHV.Mapping epitope structure and activity:from one-dimensional prediction to four-dimensional description of antigenic specificity.Methods:A companion to Methods in Enzymology,1996,9∶465-472.
[3] Daniels DA,Lane DP.Phage peptide libraries.Methods Enzymol,1996,9∶494-507.
, 百拇医药
[4] Folgori A,Tafi R,Meola A,et al.A general strategy to identify mimotopes of pathological antigens using only random peptide libraries and human sera.EMBO J,1994,13(9)∶2236-2243.
[5] Dybwad A,Bogen B,Natvig JB,et al.peptide phage libraries can be an efficient tool for identifying antibody ligands for polyclonal antisera.Clin Exp Immunol,1995,102∶438-442.
[6] Germaschewski V,Murray K.Identification of polyclonal serum specificities with phagedisplay libraries.J Virol Methods,1996,58∶21-32.
, 百拇医药
[7] Prezzi C,Nuzzo M, Meola A,et al.Selection of antigenic and immunogenic mimics of hepatitis C virus using sera from patients.J Immunol,1996,4 504-4 513.
[8] Sallberg M,Pumpen P,Zhang ZX,et al.Locations of antibody binding sites within conserved regions of the HCV core protein.J Med Virol,1994,43∶62-68.
[9] Kotwal GJ,Baroudy BM,Kuramoto IK,et al.Detection of acute hepatitis C virus infection by ELISA using a synthetic peptide comprising a structural epitope.Proc Natl Acad Sci USA,1992,89∶4 486-4 489.
, 百拇医药
[10] Okamoto H,Tsuda F,Machida A,et al.Antibodies against synthetic oligopeptides deduced from the putative core gene for the diagnosis of HCV infection.Hepatology,1992,15∶180-186.
[11] Nasoff MS,Zebedee SL,Inchauspe G,et al.Identification of an immunodominant epitope within the capsid protein of HCV.Proc Natl Acad Sci USA,1991,88∶5 462-5 466.
[12] Goeser T,Muller HM,Ye J,et al.Characterization of antigenic determinants in the core antigen of the hepatitis C virus.virology,1994,205∶462-469.
(收稿:1997-08-07 修回:1998-03-12), 百拇医药