猪瘟病毒石门株NS2-3基因片段的序列测定及比较
作者:黄茜华 张楚瑜 王 宁 傅烈振 王家富
单位:武汉大学生命科学学院,武汉 430072
关键词:猪瘟病毒石门株;NS2-3基因片段;克隆;序列分析
猪瘟病毒石门株NS2 摘 要 根据猪瘟病毒C株的序列,以计算机辅助设计,化学合成1对引物(PF 5648/PR 6604),应用RT-PCR技术从感染猪血中成功地扩增了我国猪瘟病毒强毒石门株NS2-3基因片段,大小为957 bp,位于NS3基因的中部NTPase和Helicase活性区。克隆后测序,结果表明该段基因产物具有解旋酶超家族全部七个特征性保守序列,包括共同的NTP结合基序A位点(GXGKT/S)和B位点(3 hy,2 x)D。序列同源性比较表明,石门株与日本的ALD和GPE-株同源性最高,与其它3株猪瘟病毒(C株、Brescia株和Alfort株)的同源性也很高,并与2株牛病毒性腹泻病毒(BVDV)(NADL株和SD-1株)也有较高的同源性,尤其是由核苷酸序列推导的氨基酸序列,同源性均大于90%,是瘟病毒属基因组中最保守的区段,这与该基因产物在病毒复制及聚蛋白前体加工过程中所具有的重要功能是一致的。
, http://www.100md.com
Sequencing and Comparison of NS2-3 Gene Fragment of Classical Swine Fever Virus Shimen Strain
Huang Qianhua Zhang Chuyu Wang Ning Fu Liezhen Wang Jiafu
(Institute of Virology, Wuhan University, Wuhan 430072)
Abstract A couple of CSFV specific primer (PF 5648 and PR 6604) was designed with the aid of computer primer designation software and synthesized based upon the relative conserved regions of published sequences of C strain. NS2-3 gene (p125 gene) fragment of CSFV Shimen strain was amplified successfully by RT-PCR from the anticoagulant blood of infected pig. The product length is 957 bp, located in the central of NS3, a putative NTPase and helicase domain. The obtained PCR product was cloned and then sequenced. The sequence showed that this fragment contained all of seven typical conserved segments of helicase superfamily, including two common NTP-binding motifs, namely, “A” site (GXGKT/S) and “B” site (3hy, 2x) D. Sequence homology analysis revealed that Shimen strain had the highest homology with Japanese strains (ALD and GPE-), and slightly lower homology with other three CSFV strains (C, BresciaandAlfort). Shimen strain had also significant homology with two BVDV strains (NADL and SD-1). The deduced amino acid sequence homology of Shimen strain with five CSFV and two BVDV strains was all upper than 90%. It is further confirmed that this fragment is the most conserved in pestivirus amino acid sequence. It is consistent with its essential function in replication and translation of virus genome and in processing of polyprotein precursor.
, 百拇医药
Key words Classical swine fever virus Shimen strain, NS2-3 gene fragment, Clone, Sequence analysis
猪瘟是危害养猪业最重要的传染病之一。猪瘟的病原是猪瘟病毒(CSFV),属于黄病毒科瘟病毒属,同属成员还包括牛病毒性腹泻病毒(BVDV)和绵羊边界病病毒(BDV)。瘟病毒基因组为单股正链RNA,长度约为12.3 kb,含有一个大的开放读码框架(ORF),编码一个由约4000个氨基酸残基组成的多聚蛋白。非致细胞病变(NCP)瘟病毒的多聚蛋白经过共翻译或翻译后切割机制,由病毒和宿主细胞酶的作用而形成11种结构蛋白和非结构蛋白,各产物按顺序排列为NH2-NPro-C-E0-E1-E2-p7-NS2-3-NS4A-NS4B-NS5A-NS5B-COOH[1]。其中NS2-3基因产物(p125)是一个多功能蛋白,NS3部分具有丝氨酸类蛋白酶活性[1,2],核苷三磷酸酶活性[1,3]及解旋酶活性[1,4,5],其中蛋白酶活性对下游各基因产物的切割至关重要[1,2],而由RNA激活的NTP酶/RNA解旋酶活性可以在病毒RNA复制、翻译等过程中解开病毒基因组的二级结构[5]。因此,该蛋白在病毒生命周期中至关重要。
, http://www.100md.com
猪瘟病毒石门株是我国猪瘟的标准强毒,1945年在石家庄解放区分离获得,是我国制造灭活苗及鉴定、攻毒用标准强毒株。对其序列的测定有助于阐明猪瘟病毒的致病机理、演变规律,并为有用基因的利用提供分子基础。我们扩增了猪瘟病毒石门株NS2-3基因片段,对其进行了克隆和测序,并与国外报道的毒株序列作了比较,为我室对石门株NS2-3基因全序列的测定工作奠定了基础,并为以后的基因表达及功能研究提供了依据。
1 材料和方法
1.1 病毒的增殖 猪瘟病毒石门株AV1411(04/8/97)购自中国兽药监察所。用生理盐水1∶10稀释后耳静脉接种2月龄断奶的猪瘟非免疫猪(购自湖北省生药厂)。每天3次检测体温,当体温升到最高而开始下降时,约第6-7天,前腔静脉采取抗凝血,-30℃冻存。
1.2 猪瘟病毒RNA提取 采用异硫氰酸胍-酚-氯仿一步法从猪瘟抗凝血中提取总RNA。
, 百拇医药
1.3 引物设计 根据已公开报道的猪瘟病毒C株核苷酸序列[6],借助计算机辅助设计了一对引物PF5648(正向引物,位置5648~5669)和PR6604(反向引物,位置6581~6604),序列如下:PF5648:5′GCA TCA AGT GGA AGG GTA GTC G3′;PR6604:5′CGT CAC TAT GAA GGG CAT CTT AGG 3′。由上海生工生物工程有限公司合成,纯度为测序级。
1.4 RT-PCR 按照常规方法进行,同时设阴性对照。
1.5 扩增产物的克隆 按照Promega公司的pGEM-T vector system说明书进行。受体菌为TG1,由本室保存。经蓝/白斑筛选,碱裂解法提取质粒,经限制性内切酶(SABC)酶切及PCR方法鉴定阳性克隆。
1.6 序列测定 采用T7和SP6正向和反向引物进行全自动测序。
, 百拇医药
1.7 序列分析和同源性比较 采用DNASIS和PROSIS软件(Hitachi Software Engineering Co.Ltd.)。
2 结果
2.1 PCR扩增及鉴定 PF5648和PR6604对应的扩增片段长957bp,位于NS3的中部,NTPase及Helicase活性区[1]。PCR产物经2%琼脂糖凝胶电泳,可见一条清晰的扩增带,大小与预期的一致,无非特异性扩增。而阴性对照无扩增。我们还采用PCR技术以扩增片段为模板进行二次扩增,也得到了大小一致的目的片段,并与C株的相应阳性片段作比较,进一步证实已获得目的片段(见图1)。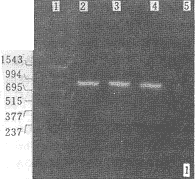
图1 957 bp片段的扩增
, 百拇医药
Fig.1 Amplification of 957bp fragment
1.PCR分子量标准 2.RT-PCR产物 3.二次扩增产物 4.阳性对照 5.阴性对照
1.PCR Marker 2.Product of RT-PCR 3.Product of the second amplification 4.Positive control 5.Negtive control
2.2 扩增产物的克隆及鉴定 PCR产物经低融点琼脂糖纯化后按Promega公司的pGEM-T vector system说明书进行克隆,命名为pGEM957。采用酶切鉴定阳性克隆。根据已报道的C株序列[6]可推测该基因片段内部存在单一的BamH Ⅰ (6436)和HindⅢ (5711)切点及两个EcoRI切点(5798,6242),而pGEM-T vector无上述切点。酶切结果显示BamHⅠ、HindⅢ、EocRⅠ均只有单一切点,推测有一个EcoRI位点已经变异。该克隆应该是阳性克隆(见图2)。序列分析结果表示推测正确,6247位碱基由C变为T,致使该处EcoRI切点消失。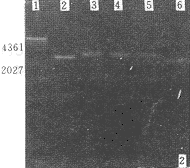
, 百拇医药
图2 pGEM957克隆的酶切鉴定
Fig.2 Identification of pGEM957 clone by restriction enzyme
1.λDNA HindⅢ酶切分子量标准 2.pGEM957 BamHⅠ和HindⅢ双酶切产物 3.pGEM957 BamHⅠ酶切产物 4.pGEM957 HindⅢ酶切产物 5.pGEM957 EcoRⅠ酶切产物 6.pGEM-T vector Pst I酶切产物
1.λDNA/HindⅢ Marker 2.pGEM957/BamHⅠ+HindⅢ 3.pGEM957/BamHⅠ 4.pGEM957/HindⅢ 5.pGEM957/EcoRⅠ 6.pGEM-T vector/PstⅠ
2.3 核苷酸序列的测定 石门株NS2-3基因片段的核苷酸序列及推导的氨基酸序列见图3。该片段具有解旋酶超家族的七个保守区段,其中包括一个截短的“A”位点:GXGKT/S(X:任意氨基酸残基)和“B”位点:(3hy,2X) D (hy:疏水性氨基酸残基),形成独立的β-链-β-转角-α-螺旋结构(β-strand-β-turn-α-helix),是推测的NTP结合区[5,7~9]。
, 百拇医药
图3 猪瘟病毒石门株NS2-3基因片段的核苷酸序列及推导的氨基酸序列
Fig.3 The nucleotide and deduced amino acid sequence of NS2-3 gene fragment of CSFV Shimen strain
2.4 NS2-3基因片段的核苷酸和氨基酸序列与国外报道的5株CSFV及2株BVDV的同源性比较 结果见表1。石门株与日本的CSFV ALD、GPE-株同源性最高,与另3株的同源性次之,与2株BVDV的同源性也较高,各株的氨基酸序列同源性均大于90%,显示了该段基因的高度保守性。如石门株与ALD、GPE-株比较在该段多肽内只有2个氨基酸残基的替换,其中一个为保守性替换。即使与2株BVDV比较也只有28个氨基酸残基的替换,其中26个是保守性替换。“A”、“B”两个NTP结合位点基序完全保守,可以推测各毒株该段多肽具有共同的高级结构。
, 百拇医药
表1 石门株NS2-3基因片段与其他瘟病毒的核苷酸和氨基酸序列同源性比较
Table 1 Nucleotide and deduced amino acid sequence homology analysis of NS2-3 gene fragment of CSFV Shimen strain with other pestiviruses
CSFV
BVDV
ALD
GPE-
C
Brescia
Alfort
, 百拇医药
SD-1
NADL
石门株NS2-3基因片段核苷酸序列同源性
Nucleotide sequence homology of NS2-3 gene
fragment of Shimen strain
97.7%
97.9%
96.3%
94.6%
87.2%
78.2%
, 百拇医药
77.7%
石门株NS2-3蛋白氨基酸序列同源性
Amino acid sequence
homology of NS2-3protein of Shimen strain
99.3%
99.3%
98.4%
98.0%
98.0%
90.1%
90.1%
, 百拇医药
3 讨论 猪瘟病毒石门株是我国的标准强毒,我们采用RT-PCR技术扩增了NS2-3基因的部分片段,序列分析表明该段基因高度保守,氨基酸序列同源性很高,两个推测的NTP结合位点完全保守,属于一个包括从大肠杆菌到人的多种解旋酶的超家族[5,7~9]。在病毒的复制、翻译等过程中解开病毒基因组RNA的二级结构,在病毒的生命周期中起重要作用。而且,GPE-和C株都是弱毒株,其余五株为强毒株,它们在该段基因区域内的高度保守性说明该区域与病毒的毒力关系不大。
值得注意的是,同源性比较显示石门株与中国猪瘟兔化弱毒疫苗株C株的序列同源性反而低于与日本ALD、GPE-株的同源性。石门株与C株在该段基因上的核苷酸序列同源性为96.3%,氨基酸序列同源性为98.4%。共有6个氨基酸残基的替换,其中五个是保守性替换。而石门株与ALD、GPE-株的核苷酸序列同源性分别为97.7%和97.9%,氨基酸序列同源性均为99.3%,只有两个氨基酸残基的替换,其中一个是保守性替换。我们还对石门株E2基因的序列进行了测定,同源性比较结果相同(结果待发表)。相反,日本的ALD及其致弱的毒株GPE-在该段基因上同源性很高(97.8%),而氨基酸的同源性更是高达100%[10]。虽然C株是用从中国分离的强毒株适应兔体培育而成的,但该结果提示石门株与C株之间的亲缘关系可能比日本的两株更远。这一设想尚需进一步的实验证实。
, 百拇医药
* 张楚瑜:通讯作者
参考文献
[1] Meyers G, Thiel H-J. Molecular characterization of pestivirues. Adv Virus Res, 1996,47:53~118
[2] Wiskerchen M, Collett M S. Pestivirus gene expression: protein p80 of bovine viral diarrhea virus is a proteinase involved in polyprotein processing. Virology, 1991,184(1):341~350
[3] Tamura J K, Warrener P, Collett M S et al. RNA-stimulated NTPase activity associated with the p80 protein of the pestivirus bovine viral diarrhea virus. Virology, 1993,193(1):1~10
, http://www.100md.com
[4] Warrener P, Collett M S. Pestivirus NS3 (p80) protein possesses RNA helicase activity. J Virol, 1995,69(3):1720~1726
[5] Gorbalenya A E, Koonin V, Donchenko A P et al. Two related superfamilies of putative helicases involved in replication, recombination, repair and expression of DNA and RNA genomes. Nucleic Acid Res, 1989,17(12):4713~4729
[6] Moormann R J M, van Gennip H G P, Miedema G K W et al. Infectious RNA transcribed from an engineered full-length cDNA template of the genome of a pestivirus. J Virol, 1996,70(2):763~770
, http://www.100md.com
[7] Gorbalenya A E, Donchenko A P, Koonin V et al. A conserved NTP-motif in putative helicases. Nature, 1988,333(5):22
[8] Hodgman T C. A new superfamily of replicative proteins. Nature, 1988,333(5):22~23
[9] Linder P, Lasko P F, Leroy P et al. Birth of the D-E-A-D box. Nature, 1989,337(6202):121~122
[10] Isbikawa K, Nagai H, Katayama K et al. Comparison of the entire nucleotide and deduced amino acid sequences of the attenuated hog cholera vaccine strain GPE- and the wild-type parental strain ALD. Arch Virol, 1995,140(8):1385~1391
收稿日期:1998-04-21,修回日期:1998-07-23, 百拇医药
单位:武汉大学生命科学学院,武汉 430072
关键词:猪瘟病毒石门株;NS2-3基因片段;克隆;序列分析
猪瘟病毒石门株NS2 摘 要 根据猪瘟病毒C株的序列,以计算机辅助设计,化学合成1对引物(PF 5648/PR 6604),应用RT-PCR技术从感染猪血中成功地扩增了我国猪瘟病毒强毒石门株NS2-3基因片段,大小为957 bp,位于NS3基因的中部NTPase和Helicase活性区。克隆后测序,结果表明该段基因产物具有解旋酶超家族全部七个特征性保守序列,包括共同的NTP结合基序A位点(GXGKT/S)和B位点(3 hy,2 x)D。序列同源性比较表明,石门株与日本的ALD和GPE-株同源性最高,与其它3株猪瘟病毒(C株、Brescia株和Alfort株)的同源性也很高,并与2株牛病毒性腹泻病毒(BVDV)(NADL株和SD-1株)也有较高的同源性,尤其是由核苷酸序列推导的氨基酸序列,同源性均大于90%,是瘟病毒属基因组中最保守的区段,这与该基因产物在病毒复制及聚蛋白前体加工过程中所具有的重要功能是一致的。
, http://www.100md.com
Sequencing and Comparison of NS2-3 Gene Fragment of Classical Swine Fever Virus Shimen Strain
Huang Qianhua Zhang Chuyu Wang Ning Fu Liezhen Wang Jiafu
(Institute of Virology, Wuhan University, Wuhan 430072)
Abstract A couple of CSFV specific primer (PF 5648 and PR 6604) was designed with the aid of computer primer designation software and synthesized based upon the relative conserved regions of published sequences of C strain. NS2-3 gene (p125 gene) fragment of CSFV Shimen strain was amplified successfully by RT-PCR from the anticoagulant blood of infected pig. The product length is 957 bp, located in the central of NS3, a putative NTPase and helicase domain. The obtained PCR product was cloned and then sequenced. The sequence showed that this fragment contained all of seven typical conserved segments of helicase superfamily, including two common NTP-binding motifs, namely, “A” site (GXGKT/S) and “B” site (3hy, 2x) D. Sequence homology analysis revealed that Shimen strain had the highest homology with Japanese strains (ALD and GPE-), and slightly lower homology with other three CSFV strains (C, BresciaandAlfort). Shimen strain had also significant homology with two BVDV strains (NADL and SD-1). The deduced amino acid sequence homology of Shimen strain with five CSFV and two BVDV strains was all upper than 90%. It is further confirmed that this fragment is the most conserved in pestivirus amino acid sequence. It is consistent with its essential function in replication and translation of virus genome and in processing of polyprotein precursor.
, 百拇医药
Key words Classical swine fever virus Shimen strain, NS2-3 gene fragment, Clone, Sequence analysis
猪瘟是危害养猪业最重要的传染病之一。猪瘟的病原是猪瘟病毒(CSFV),属于黄病毒科瘟病毒属,同属成员还包括牛病毒性腹泻病毒(BVDV)和绵羊边界病病毒(BDV)。瘟病毒基因组为单股正链RNA,长度约为12.3 kb,含有一个大的开放读码框架(ORF),编码一个由约4000个氨基酸残基组成的多聚蛋白。非致细胞病变(NCP)瘟病毒的多聚蛋白经过共翻译或翻译后切割机制,由病毒和宿主细胞酶的作用而形成11种结构蛋白和非结构蛋白,各产物按顺序排列为NH2-NPro-C-E0-E1-E2-p7-NS2-3-NS4A-NS4B-NS5A-NS5B-COOH[1]。其中NS2-3基因产物(p125)是一个多功能蛋白,NS3部分具有丝氨酸类蛋白酶活性[1,2],核苷三磷酸酶活性[1,3]及解旋酶活性[1,4,5],其中蛋白酶活性对下游各基因产物的切割至关重要[1,2],而由RNA激活的NTP酶/RNA解旋酶活性可以在病毒RNA复制、翻译等过程中解开病毒基因组的二级结构[5]。因此,该蛋白在病毒生命周期中至关重要。
, http://www.100md.com
猪瘟病毒石门株是我国猪瘟的标准强毒,1945年在石家庄解放区分离获得,是我国制造灭活苗及鉴定、攻毒用标准强毒株。对其序列的测定有助于阐明猪瘟病毒的致病机理、演变规律,并为有用基因的利用提供分子基础。我们扩增了猪瘟病毒石门株NS2-3基因片段,对其进行了克隆和测序,并与国外报道的毒株序列作了比较,为我室对石门株NS2-3基因全序列的测定工作奠定了基础,并为以后的基因表达及功能研究提供了依据。
1 材料和方法
1.1 病毒的增殖 猪瘟病毒石门株AV1411(04/8/97)购自中国兽药监察所。用生理盐水1∶10稀释后耳静脉接种2月龄断奶的猪瘟非免疫猪(购自湖北省生药厂)。每天3次检测体温,当体温升到最高而开始下降时,约第6-7天,前腔静脉采取抗凝血,-30℃冻存。
1.2 猪瘟病毒RNA提取 采用异硫氰酸胍-酚-氯仿一步法从猪瘟抗凝血中提取总RNA。
, 百拇医药
1.3 引物设计 根据已公开报道的猪瘟病毒C株核苷酸序列[6],借助计算机辅助设计了一对引物PF5648(正向引物,位置5648~5669)和PR6604(反向引物,位置6581~6604),序列如下:PF5648:5′GCA TCA AGT GGA AGG GTA GTC G3′;PR6604:5′CGT CAC TAT GAA GGG CAT CTT AGG 3′。由上海生工生物工程有限公司合成,纯度为测序级。
1.4 RT-PCR 按照常规方法进行,同时设阴性对照。
1.5 扩增产物的克隆 按照Promega公司的pGEM-T vector system说明书进行。受体菌为TG1,由本室保存。经蓝/白斑筛选,碱裂解法提取质粒,经限制性内切酶(SABC)酶切及PCR方法鉴定阳性克隆。
1.6 序列测定 采用T7和SP6正向和反向引物进行全自动测序。
, 百拇医药
1.7 序列分析和同源性比较 采用DNASIS和PROSIS软件(Hitachi Software Engineering Co.Ltd.)。
2 结果
2.1 PCR扩增及鉴定 PF5648和PR6604对应的扩增片段长957bp,位于NS3的中部,NTPase及Helicase活性区[1]。PCR产物经2%琼脂糖凝胶电泳,可见一条清晰的扩增带,大小与预期的一致,无非特异性扩增。而阴性对照无扩增。我们还采用PCR技术以扩增片段为模板进行二次扩增,也得到了大小一致的目的片段,并与C株的相应阳性片段作比较,进一步证实已获得目的片段(见图1)。

图1 957 bp片段的扩增
, 百拇医药
Fig.1 Amplification of 957bp fragment
1.PCR分子量标准 2.RT-PCR产物 3.二次扩增产物 4.阳性对照 5.阴性对照
1.PCR Marker 2.Product of RT-PCR 3.Product of the second amplification 4.Positive control 5.Negtive control
2.2 扩增产物的克隆及鉴定 PCR产物经低融点琼脂糖纯化后按Promega公司的pGEM-T vector system说明书进行克隆,命名为pGEM957。采用酶切鉴定阳性克隆。根据已报道的C株序列[6]可推测该基因片段内部存在单一的BamH Ⅰ (6436)和HindⅢ (5711)切点及两个EcoRI切点(5798,6242),而pGEM-T vector无上述切点。酶切结果显示BamHⅠ、HindⅢ、EocRⅠ均只有单一切点,推测有一个EcoRI位点已经变异。该克隆应该是阳性克隆(见图2)。序列分析结果表示推测正确,6247位碱基由C变为T,致使该处EcoRI切点消失。

, 百拇医药
图2 pGEM957克隆的酶切鉴定
Fig.2 Identification of pGEM957 clone by restriction enzyme
1.λDNA HindⅢ酶切分子量标准 2.pGEM957 BamHⅠ和HindⅢ双酶切产物 3.pGEM957 BamHⅠ酶切产物 4.pGEM957 HindⅢ酶切产物 5.pGEM957 EcoRⅠ酶切产物 6.pGEM-T vector Pst I酶切产物
1.λDNA/HindⅢ Marker 2.pGEM957/BamHⅠ+HindⅢ 3.pGEM957/BamHⅠ 4.pGEM957/HindⅢ 5.pGEM957/EcoRⅠ 6.pGEM-T vector/PstⅠ
2.3 核苷酸序列的测定 石门株NS2-3基因片段的核苷酸序列及推导的氨基酸序列见图3。该片段具有解旋酶超家族的七个保守区段,其中包括一个截短的“A”位点:GXGKT/S(X:任意氨基酸残基)和“B”位点:(3hy,2X) D (hy:疏水性氨基酸残基),形成独立的β-链-β-转角-α-螺旋结构(β-strand-β-turn-α-helix),是推测的NTP结合区[5,7~9]。

, 百拇医药
图3 猪瘟病毒石门株NS2-3基因片段的核苷酸序列及推导的氨基酸序列
Fig.3 The nucleotide and deduced amino acid sequence of NS2-3 gene fragment of CSFV Shimen strain
2.4 NS2-3基因片段的核苷酸和氨基酸序列与国外报道的5株CSFV及2株BVDV的同源性比较 结果见表1。石门株与日本的CSFV ALD、GPE-株同源性最高,与另3株的同源性次之,与2株BVDV的同源性也较高,各株的氨基酸序列同源性均大于90%,显示了该段基因的高度保守性。如石门株与ALD、GPE-株比较在该段多肽内只有2个氨基酸残基的替换,其中一个为保守性替换。即使与2株BVDV比较也只有28个氨基酸残基的替换,其中26个是保守性替换。“A”、“B”两个NTP结合位点基序完全保守,可以推测各毒株该段多肽具有共同的高级结构。
, 百拇医药
表1 石门株NS2-3基因片段与其他瘟病毒的核苷酸和氨基酸序列同源性比较
Table 1 Nucleotide and deduced amino acid sequence homology analysis of NS2-3 gene fragment of CSFV Shimen strain with other pestiviruses
CSFV
BVDV
ALD
GPE-
C
Brescia
Alfort
, 百拇医药
SD-1
NADL
石门株NS2-3基因片段核苷酸序列同源性
Nucleotide sequence homology of NS2-3 gene
fragment of Shimen strain
97.7%
97.9%
96.3%
94.6%
87.2%
78.2%
, 百拇医药
77.7%
石门株NS2-3蛋白氨基酸序列同源性
Amino acid sequence
homology of NS2-3protein of Shimen strain
99.3%
99.3%
98.4%
98.0%
98.0%
90.1%
90.1%
, 百拇医药
3 讨论 猪瘟病毒石门株是我国的标准强毒,我们采用RT-PCR技术扩增了NS2-3基因的部分片段,序列分析表明该段基因高度保守,氨基酸序列同源性很高,两个推测的NTP结合位点完全保守,属于一个包括从大肠杆菌到人的多种解旋酶的超家族[5,7~9]。在病毒的复制、翻译等过程中解开病毒基因组RNA的二级结构,在病毒的生命周期中起重要作用。而且,GPE-和C株都是弱毒株,其余五株为强毒株,它们在该段基因区域内的高度保守性说明该区域与病毒的毒力关系不大。
值得注意的是,同源性比较显示石门株与中国猪瘟兔化弱毒疫苗株C株的序列同源性反而低于与日本ALD、GPE-株的同源性。石门株与C株在该段基因上的核苷酸序列同源性为96.3%,氨基酸序列同源性为98.4%。共有6个氨基酸残基的替换,其中五个是保守性替换。而石门株与ALD、GPE-株的核苷酸序列同源性分别为97.7%和97.9%,氨基酸序列同源性均为99.3%,只有两个氨基酸残基的替换,其中一个是保守性替换。我们还对石门株E2基因的序列进行了测定,同源性比较结果相同(结果待发表)。相反,日本的ALD及其致弱的毒株GPE-在该段基因上同源性很高(97.8%),而氨基酸的同源性更是高达100%[10]。虽然C株是用从中国分离的强毒株适应兔体培育而成的,但该结果提示石门株与C株之间的亲缘关系可能比日本的两株更远。这一设想尚需进一步的实验证实。
, 百拇医药
* 张楚瑜:通讯作者
参考文献
[1] Meyers G, Thiel H-J. Molecular characterization of pestivirues. Adv Virus Res, 1996,47:53~118
[2] Wiskerchen M, Collett M S. Pestivirus gene expression: protein p80 of bovine viral diarrhea virus is a proteinase involved in polyprotein processing. Virology, 1991,184(1):341~350
[3] Tamura J K, Warrener P, Collett M S et al. RNA-stimulated NTPase activity associated with the p80 protein of the pestivirus bovine viral diarrhea virus. Virology, 1993,193(1):1~10
, http://www.100md.com
[4] Warrener P, Collett M S. Pestivirus NS3 (p80) protein possesses RNA helicase activity. J Virol, 1995,69(3):1720~1726
[5] Gorbalenya A E, Koonin V, Donchenko A P et al. Two related superfamilies of putative helicases involved in replication, recombination, repair and expression of DNA and RNA genomes. Nucleic Acid Res, 1989,17(12):4713~4729
[6] Moormann R J M, van Gennip H G P, Miedema G K W et al. Infectious RNA transcribed from an engineered full-length cDNA template of the genome of a pestivirus. J Virol, 1996,70(2):763~770
, http://www.100md.com
[7] Gorbalenya A E, Donchenko A P, Koonin V et al. A conserved NTP-motif in putative helicases. Nature, 1988,333(5):22
[8] Hodgman T C. A new superfamily of replicative proteins. Nature, 1988,333(5):22~23
[9] Linder P, Lasko P F, Leroy P et al. Birth of the D-E-A-D box. Nature, 1989,337(6202):121~122
[10] Isbikawa K, Nagai H, Katayama K et al. Comparison of the entire nucleotide and deduced amino acid sequences of the attenuated hog cholera vaccine strain GPE- and the wild-type parental strain ALD. Arch Virol, 1995,140(8):1385~1391
收稿日期:1998-04-21,修回日期:1998-07-23, 百拇医药