c-met原癌基因的表达及其与人肝细胞癌转移关系的研究
作者:张国新 徐顺福 赵志泉 倪 黎 王学浩
单位:张国新 徐顺福 赵志泉 南京医科大学第一附属医院 消化科 邮政编码:210029;倪 黎 南京医科大学第一附属医院 中心实验室 邮政编码:210029;王学浩 南京医科大学第一附属医院 普外科 邮政编码:210029
关键词:原癌基因 人肝细胞 基因表达 聚合酶链反应
江苏医药990806
摘要 以β-actin为内标,用逆转录-多聚酶链反应(RT-PCR)技术,研究c-met mRNA在32例肝癌组织、26例癌旁肝组织中的表达。结果表明,32例肝癌组织、26例癌旁肝组织中均有c-met基因表达(表达率100%),但肝癌c-met基因的表达值显著高于癌旁肝组织(46.75%±13.03%对25.31%±11.03%,p<0.001)。32例肝癌中c-met基因过度表达者有14例,统计分析发现c-met基因过度表达与肝细胞癌的分化程度及肝内转移有关。结果提示c-met基因在人肝细胞癌中高表达,并可能与肝癌的侵袭转移有关。
, 百拇医药
c-met原癌基因是一具有酪氨酸酶活性的生长因子受体,研究表明它与肿瘤转移有关。我们以β-actin基因为内参照,采用半定量逆转录-聚合酶链反应(RT-PCR)技术研究肝癌中c-met原癌基因的表达,探讨c-met基因表达与肝细胞癌浸润转移的关系。
材料和方法
一、实验材料
1、人肝细胞癌标本取自1997年4月至1998年2月在我院普外科及江苏省肿瘤医院普外科所接受手术切除并经病理证实为肝细胞癌的患者,共32例,男24例、女8例,平均年龄55岁。其中26例有癌旁组织,病理证实均无癌细胞浸润。
2、主要试剂:AMV逆转录酶及TaqDNA聚合酶均购自Promega公司。c-met和β-actin基因引物由上海细胞研究所合成,扩增片段大小分别为655bp和206bp,β-actin基因用作内对照扩增。引物序列分别为:
, 百拇医药
c-met 上游引物
5’ACAGTGGCATGTCAACATCG-3’
下游引物
5’-GCTCGGTAGTCTACAGATTC-3’
β-actin 上游引物
5’-GGCGGCACCACCATGTACCCT-3’
下游引物
5’-AGGGGCCGGACTCGTCATCATACT-3’
3、主要仪器:DNA扩增仪(PE公司),光密度扫描仪(Bio-Rad公司)
二、实验方法
, 百拇医药
1、总RNA提取:避开癌症坏死区,切下标本约100mg立即液氮保存,参照Chomczynski等[1]一步法:标本加入异硫氰酸胍裂解液后,依次用酚、氯仿/异戊醇抽提,异丙醇沉淀RNA,最后用乙醇洗脱,提取的RNA溶于适量DEPC水中,测定A260吸光度值定量RNA。
2、RT-PCR:20μl逆转录反应体系中依次加入RT缓冲液、1.0μg总RNA、0.2μgOligo dT、2μl dNTP(10mM)、20U RNasin及其AMV 20U,混匀后42℃反应1小时,然后95℃8分钟以灭活AMV。取10μl cDNA依次加入PCR缓冲液、c-met基因和β-actin基因上、下游引物各20pmol/L、TaqDNA聚合酶1.5U及双蒸水至反应总体积为50μl。PCR循环参数:94℃45秒、56℃45秒、72℃60秒,循环30次后72℃7分钟,分别扩增c-met及β-actin基因片段。
3、PCR产物半定量分析:使用含有溴化乙啶的2%琼脂糖凝胶电泳分析PCR产物,并用标准PCR Maker对照,见655bp、206bp三个片段,照相记录特异性条带,摄影底片用激光密度扫描仪分析PCR产物带,测定其光密度值。
, 百拇医药
4、统计学分析:经方差齐性检验后,两组间均数的比较采用t检验;两个样本率的比较用χ2检验。Ρ<0.05时判定有显著性差异。
结 果
一、肝癌和癌旁肝组织中c-met mRNA表达(见附图)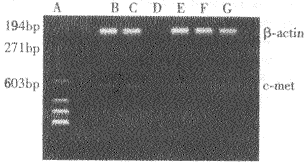
附图 c-met基因扩增图谱
注:A,Marker;B、C,肝癌;D,阴性对照;
E、F、G,癌旁组织
c-met基因在32例肝癌组织、26例相应癌旁肝组织中均有表达,表达率为100%(32/32、26/26);肝癌中c-met基因的表达值(表达值=目的基因光密度值/β-actin光密度值×100%)为46.75%±13.03%,而26例癌旁组织中的表达值为25.31%±11.03%,经统计学分析,两者有显著性差异(t=7.64,P<0.001)。
, http://www.100md.com
附表 c-met mRNA表达与肝癌临床病理特征间的关系 特 征
高c-met HCC
(n=14例)
低c-met HCC
(n=18例)
P值
年龄
60.6±21.3
53.8±17.5
>0.05
性别(男:女)
11:3
, http://www.100md.com
13:5
>0.05
HBsAg阳性
5/14(35.7%)
6/18(33.3%)
>0.05
肝硬化
6/14(42.8%)
6/18(33.3%)
>0.05
AFP(≥20ng/ml)
4/14(28.6)
, 百拇医药
5/18(27.8%)
>0.05
肿瘤大小(cm)
3.6±1.4
3.9±1.2
>0.05
癌栓形成
5/14(35.6%)
4/18(22.2%)
>0.05
肝内转移
9/14(64.4%)
, http://www.100md.com
5/18(27.8%)
<0.05#
病理分化(中~高)
11/14(79.5%)
6/18(33.3%)
<0.05*
#χ2=4.26;*χ2=6.47
二、c-met表达与肝癌临床病理特征的关系
以肝癌中c-met表达的平均值为界将32例肝癌分成两组;高c-met肝癌组(14例)和低c-met肝癌组(18例)。结果表明:高c-met肝癌与癌症肝内转移、病理分化程度中或差有关(见附表)。
, 百拇医药
讨 论
近年,随着对肿瘤转移机理研究的深入,发现一些细胞因子参与了肿瘤细胞的转移,这些细胞因子通过与受体结合并经信息传导而引发癌细胞的运动。c-met原癌基因是肝细胞生长因子(Hepatocyte Growth Factor, HGF)编码的酪氨酸激酶受体,体内外研究证实,HGF不仅促进肿瘤生长,而且与肿瘤浸润和转移有关[3]。c-met原癌基因产物(c-Met)的生成一个剪切为α、β两个亚单位的190-KD前体的合成有关,成熟受体的激活需要与HGF结合[4]。某些癌细胞,在剪切过程中有缺陷,190-KD的前体无需结合配基就有活性,因此,这些细胞失去了HGF的控制,且外源性HGF缺乏时运动增加。已证实c-met基因在胃癌、甲状腺癌等实体瘤中有过度表达,肝癌中也有关于c-met表达的报道,但它们之间的确切关系尚不清楚。本文用半定量RT-PCR技术检测肝细胞癌中c-met的表达,以探讨它们与肝癌侵袭转移的关系。
, http://www.100md.com 本组资料显示,肝癌组织、癌旁肝组织中均有c-met mRNA表达,两组表达率无差异;但肝癌中c-met mRNA表达值明显高于癌旁肝组织(p<0.001)。如果以肝癌病人的平均表达值为界,将之分为“高表达c-met基因”及“低表达c-met基因”肝癌,结果发现:高表达c-met基因的肝癌病人其癌细胞肝内转移的机会更多、肝癌的病理分化程度更差。Suzuki等[5]认为c-Met蛋白表达与肝细胞癌的分化有关,Ueki等[6]研究发现c-met基因表达与肝癌的肝内转移有关,但也有人报告c-met基因表达与肝癌的临床及病理特征无关[7]。这一结果支持Ueki及Suzuki的观点。提示c-met与其配基HGF的相互作用促进了癌细胞的运动,并诱导了肝癌转移。其作用机理为:HGF与其受体c-met结合通过信号传导系统引起酪氨酸磷酸化,改变了细胞内游离钙离子水平,进而影响细胞游走、生长及表型变化[8]。
在高表达c-met的肝癌中,HGF起促运动作用,它通过旁分泌途径刺激癌细胞的运动。HGF能上调c-met基因的表达,Giordano[9]发现当用HGF刺激时,受体c-met可使那些原HGF阴性的细胞出现活动能力并侵犯基底膜。Rong[10]证明用c-met基因转染表达HGF的细胞后,能增加细胞的致瘤性,故具有过度表达c-met mRNA的癌细胞对HGF更为敏感、更具活动性,从而使癌细胞发生浸润和转移。
, http://www.100md.com
我们发现,c-met基因在肝癌中有过度表达,并在肝癌的浸润和转移过程中起一定作用。
参考文献
[1] Chomczynski P, Sacchi N.Analytical Biochemistry 1987;162:2524.
[2] Stoker M and Gherardi E.Biochem Biophys Acta 1991;1072.
[3] Weidner KM, et al.Proc Natl Acad Sci USA 1991;88;7001.
[4] Prat M,et al. Cell 1986;45:895.
[5] Suzuki K,et al.Hepatology 1994;20:1231.
, 百拇医药
[6] Ueki T, et al. Hepatology 1997;25:619.
[7] Biox L,et al. Hepatology 1994;19:88.
[8] Weidner DM,et al.J Cell Biol 1993;121:145.
[9] Giordano S,et al.Nature 1989;339:155.
[10] Rong S,et al. Mol Cell Biol 1992;12:5152.
(1998年12月2日收稿 1999年1月5日修回), http://www.100md.com
单位:张国新 徐顺福 赵志泉 南京医科大学第一附属医院 消化科 邮政编码:210029;倪 黎 南京医科大学第一附属医院 中心实验室 邮政编码:210029;王学浩 南京医科大学第一附属医院 普外科 邮政编码:210029
关键词:原癌基因 人肝细胞 基因表达 聚合酶链反应
江苏医药990806
摘要 以β-actin为内标,用逆转录-多聚酶链反应(RT-PCR)技术,研究c-met mRNA在32例肝癌组织、26例癌旁肝组织中的表达。结果表明,32例肝癌组织、26例癌旁肝组织中均有c-met基因表达(表达率100%),但肝癌c-met基因的表达值显著高于癌旁肝组织(46.75%±13.03%对25.31%±11.03%,p<0.001)。32例肝癌中c-met基因过度表达者有14例,统计分析发现c-met基因过度表达与肝细胞癌的分化程度及肝内转移有关。结果提示c-met基因在人肝细胞癌中高表达,并可能与肝癌的侵袭转移有关。
, 百拇医药
c-met原癌基因是一具有酪氨酸酶活性的生长因子受体,研究表明它与肿瘤转移有关。我们以β-actin基因为内参照,采用半定量逆转录-聚合酶链反应(RT-PCR)技术研究肝癌中c-met原癌基因的表达,探讨c-met基因表达与肝细胞癌浸润转移的关系。
材料和方法
一、实验材料
1、人肝细胞癌标本取自1997年4月至1998年2月在我院普外科及江苏省肿瘤医院普外科所接受手术切除并经病理证实为肝细胞癌的患者,共32例,男24例、女8例,平均年龄55岁。其中26例有癌旁组织,病理证实均无癌细胞浸润。
2、主要试剂:AMV逆转录酶及TaqDNA聚合酶均购自Promega公司。c-met和β-actin基因引物由上海细胞研究所合成,扩增片段大小分别为655bp和206bp,β-actin基因用作内对照扩增。引物序列分别为:
, 百拇医药
c-met 上游引物
5’ACAGTGGCATGTCAACATCG-3’
下游引物
5’-GCTCGGTAGTCTACAGATTC-3’
β-actin 上游引物
5’-GGCGGCACCACCATGTACCCT-3’
下游引物
5’-AGGGGCCGGACTCGTCATCATACT-3’
3、主要仪器:DNA扩增仪(PE公司),光密度扫描仪(Bio-Rad公司)
二、实验方法
, 百拇医药
1、总RNA提取:避开癌症坏死区,切下标本约100mg立即液氮保存,参照Chomczynski等[1]一步法:标本加入异硫氰酸胍裂解液后,依次用酚、氯仿/异戊醇抽提,异丙醇沉淀RNA,最后用乙醇洗脱,提取的RNA溶于适量DEPC水中,测定A260吸光度值定量RNA。
2、RT-PCR:20μl逆转录反应体系中依次加入RT缓冲液、1.0μg总RNA、0.2μgOligo dT、2μl dNTP(10mM)、20U RNasin及其AMV 20U,混匀后42℃反应1小时,然后95℃8分钟以灭活AMV。取10μl cDNA依次加入PCR缓冲液、c-met基因和β-actin基因上、下游引物各20pmol/L、TaqDNA聚合酶1.5U及双蒸水至反应总体积为50μl。PCR循环参数:94℃45秒、56℃45秒、72℃60秒,循环30次后72℃7分钟,分别扩增c-met及β-actin基因片段。
3、PCR产物半定量分析:使用含有溴化乙啶的2%琼脂糖凝胶电泳分析PCR产物,并用标准PCR Maker对照,见655bp、206bp三个片段,照相记录特异性条带,摄影底片用激光密度扫描仪分析PCR产物带,测定其光密度值。
, 百拇医药
4、统计学分析:经方差齐性检验后,两组间均数的比较采用t检验;两个样本率的比较用χ2检验。Ρ<0.05时判定有显著性差异。
结 果
一、肝癌和癌旁肝组织中c-met mRNA表达(见附图)

附图 c-met基因扩增图谱
注:A,Marker;B、C,肝癌;D,阴性对照;
E、F、G,癌旁组织
c-met基因在32例肝癌组织、26例相应癌旁肝组织中均有表达,表达率为100%(32/32、26/26);肝癌中c-met基因的表达值(表达值=目的基因光密度值/β-actin光密度值×100%)为46.75%±13.03%,而26例癌旁组织中的表达值为25.31%±11.03%,经统计学分析,两者有显著性差异(t=7.64,P<0.001)。
, http://www.100md.com
附表 c-met mRNA表达与肝癌临床病理特征间的关系 特 征
高c-met HCC
(n=14例)
低c-met HCC
(n=18例)
P值
年龄
60.6±21.3
53.8±17.5
>0.05
性别(男:女)
11:3
, http://www.100md.com
13:5
>0.05
HBsAg阳性
5/14(35.7%)
6/18(33.3%)
>0.05
肝硬化
6/14(42.8%)
6/18(33.3%)
>0.05
AFP(≥20ng/ml)
4/14(28.6)
, 百拇医药
5/18(27.8%)
>0.05
肿瘤大小(cm)
3.6±1.4
3.9±1.2
>0.05
癌栓形成
5/14(35.6%)
4/18(22.2%)
>0.05
肝内转移
9/14(64.4%)
, http://www.100md.com
5/18(27.8%)
<0.05#
病理分化(中~高)
11/14(79.5%)
6/18(33.3%)
<0.05*
#χ2=4.26;*χ2=6.47
二、c-met表达与肝癌临床病理特征的关系
以肝癌中c-met表达的平均值为界将32例肝癌分成两组;高c-met肝癌组(14例)和低c-met肝癌组(18例)。结果表明:高c-met肝癌与癌症肝内转移、病理分化程度中或差有关(见附表)。
, 百拇医药
讨 论
近年,随着对肿瘤转移机理研究的深入,发现一些细胞因子参与了肿瘤细胞的转移,这些细胞因子通过与受体结合并经信息传导而引发癌细胞的运动。c-met原癌基因是肝细胞生长因子(Hepatocyte Growth Factor, HGF)编码的酪氨酸激酶受体,体内外研究证实,HGF不仅促进肿瘤生长,而且与肿瘤浸润和转移有关[3]。c-met原癌基因产物(c-Met)的生成一个剪切为α、β两个亚单位的190-KD前体的合成有关,成熟受体的激活需要与HGF结合[4]。某些癌细胞,在剪切过程中有缺陷,190-KD的前体无需结合配基就有活性,因此,这些细胞失去了HGF的控制,且外源性HGF缺乏时运动增加。已证实c-met基因在胃癌、甲状腺癌等实体瘤中有过度表达,肝癌中也有关于c-met表达的报道,但它们之间的确切关系尚不清楚。本文用半定量RT-PCR技术检测肝细胞癌中c-met的表达,以探讨它们与肝癌侵袭转移的关系。
, http://www.100md.com 本组资料显示,肝癌组织、癌旁肝组织中均有c-met mRNA表达,两组表达率无差异;但肝癌中c-met mRNA表达值明显高于癌旁肝组织(p<0.001)。如果以肝癌病人的平均表达值为界,将之分为“高表达c-met基因”及“低表达c-met基因”肝癌,结果发现:高表达c-met基因的肝癌病人其癌细胞肝内转移的机会更多、肝癌的病理分化程度更差。Suzuki等[5]认为c-Met蛋白表达与肝细胞癌的分化有关,Ueki等[6]研究发现c-met基因表达与肝癌的肝内转移有关,但也有人报告c-met基因表达与肝癌的临床及病理特征无关[7]。这一结果支持Ueki及Suzuki的观点。提示c-met与其配基HGF的相互作用促进了癌细胞的运动,并诱导了肝癌转移。其作用机理为:HGF与其受体c-met结合通过信号传导系统引起酪氨酸磷酸化,改变了细胞内游离钙离子水平,进而影响细胞游走、生长及表型变化[8]。
在高表达c-met的肝癌中,HGF起促运动作用,它通过旁分泌途径刺激癌细胞的运动。HGF能上调c-met基因的表达,Giordano[9]发现当用HGF刺激时,受体c-met可使那些原HGF阴性的细胞出现活动能力并侵犯基底膜。Rong[10]证明用c-met基因转染表达HGF的细胞后,能增加细胞的致瘤性,故具有过度表达c-met mRNA的癌细胞对HGF更为敏感、更具活动性,从而使癌细胞发生浸润和转移。
, http://www.100md.com
我们发现,c-met基因在肝癌中有过度表达,并在肝癌的浸润和转移过程中起一定作用。
参考文献
[1] Chomczynski P, Sacchi N.Analytical Biochemistry 1987;162:2524.
[2] Stoker M and Gherardi E.Biochem Biophys Acta 1991;1072.
[3] Weidner KM, et al.Proc Natl Acad Sci USA 1991;88;7001.
[4] Prat M,et al. Cell 1986;45:895.
[5] Suzuki K,et al.Hepatology 1994;20:1231.
, 百拇医药
[6] Ueki T, et al. Hepatology 1997;25:619.
[7] Biox L,et al. Hepatology 1994;19:88.
[8] Weidner DM,et al.J Cell Biol 1993;121:145.
[9] Giordano S,et al.Nature 1989;339:155.
[10] Rong S,et al. Mol Cell Biol 1992;12:5152.
(1998年12月2日收稿 1999年1月5日修回), http://www.100md.com