一种基因表达的系统分析的方法
作者:周津 王秀琴 刘芝华 吴旻*
单位:100021北京,中国医学科学院肿瘤研究所分子肿瘤学国家重点实验室;
关键词:
一种基因表达的系统分析的方法 随着人类基因组全部核苷酸测序计划的即将完成,研究的焦点逐渐由结构基因组学转向功能基因组学,而全基因组表达分析也成为功能基因组研究的重要组成部分[1]。以往表达序列标签(EST)测序、mRNA差异显示、消减杂交及差异杂交等在克隆差异表达基因方面做出了巨大贡献,但由于它们的随机性均无法描绘出基因表达模式的全貌,所获得的多限于高丰度或表达差异大的基因[2-5],且无法得知丰度的信息以及与其他差异表达基因相比的相对重要性。而目前用于定量表达分析的Northern杂交、逆转录PCR、竞争PCR(competitive PCR)等方法[6],分析的基因数量很有限。DNA芯片技术虽可一次分析大量基因的表达状况,但其价格昂贵且对设备有特殊要求,目前尚不能推广。1995年Velculescu等[7]发表了基因表达的系统分析(serial analysis of gene expression,SAGE),这是目前唯一以测序为基础的定量分析全基因组表达模式的技术,它充分利用了已有的测序结果,在定量分析各转录子表达水平及检测低丰度表达基因方面具有明显的优势,而且在有PCR仪、手工测序设备及计算机的实验室均可实施。该技术已成功地用于酵母转录组分析、正常与肿瘤细胞表达模式比较,以及p53诱导的凋亡机理的研究中。现就SAGE原理及有代表性的应用实例进行综述。
, 百拇医药
1 SAGE的原理[7]
SAGE的主要原理:(1)以转录子特定区域的一个9~11bp的寡核苷酸短序列(即标签,tag)特异地代表该转录子。因为一个10bp的寡核苷酸有1 000 000多种(410)排列方式,而目前所知人类基因组也只有大约100 000个不同的转录子,这种10bp标签所代表的复杂性超出了人类以及任何其他哺乳动物细胞中的基因数目,它含有足够的信息代表该转录子。(2)将多个短序列标签串连在一个克隆中,通过测序以连续的方式同时分析多个标签及其代表的转录子,明显提高了分析效率。(3)根据测序结果,计算某一标签出现的次数来确定相应转录子的拷贝数与表达水平。
这一技术的基本流程见图1[8]。该项技术中的两个酶:锚定酶(anchoring enzyme,AE)常采用4个碱基识别位点的限制性内切酶,因为理论上每256个碱基就有1个这种酶的切点,使绝大多数转录子至少被切割1次;标签酶(tagging enzyme,TE)为Ⅱs型限制性内切酶(如BsmFⅠ),其切点距识别位点10~14bp,切割后即产生与连接子相连的9~11bp的短cDNA tag。在连接与扩增步骤(5、6)中,只有tag呈尾-尾相连(ditag)的分子才能得到扩增,(因为PCR的引物是依据连接子的序列设计的),这一步骤除扩增ditag的数目外,对于最后分析中确定tag的方向,ditag之间的界限均具有重要意义。最后所得测序结果用SAGE软件包进行分析,从粗序列中提取tag序列及其拷贝数的信息,每一个tag可通过对比已知核苷酸序列数据库确认其所代表的基因。筛选cDNA文库获得全长。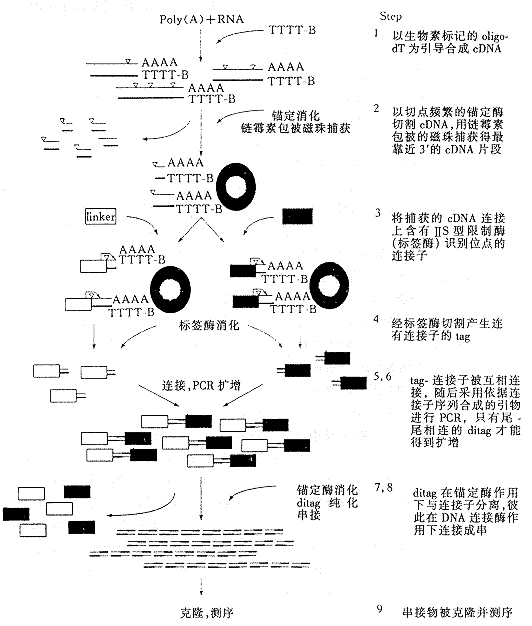
, 百拇医药
图1 SAGE方法流程图
Velculescu等[7]最早应用SAGE分析了胰腺细胞的基因表达,测定了1 000个tag,通过比较核苷酸数据库,发现95%以上的9bp tag具有很好的代表性,以一些胰腺特异高表达的基因为探针筛选来自同一组织标本的cDNA文库,所获得的克隆数与SAGE分析得出的相对丰度相吻合,表明SAGE能够良好地反映原初组织中的表达状况,而且用tag序列作寡核苷酸探针筛选cDNA文库,也获得了良好的杂交信号。
2 SAGE的应用
2.1 应用SAGE进行转录组分析 所谓转录组(transcriptome)是指一定类型细胞内所有转录的基因及其丰度,它决定着细胞的表型,与基因组不同,转录组是不稳定的,不同种类的细胞具有不同的转录组。1997年,Velculescu等[9]应用SAGE成功地分析了酵母的转录组,这也是迄今为止第1个详尽的真核生物转录组。酵母基因组经序列分析预计有6121个基因[10]。应用SAGE对酵母在3种生长状态下——对数生长期、S期阻滞和G2/M阻滞的表达状况进行分析,测序共获得60 633个tag,其中93%(56 291)代表来自酵母基因组的转录子,对应于4665个表达基因,占酵母基因总数的75%,丰度每个细胞0.3~200个拷贝不等,其余来自线粒体或质粒。Monte Carlo分析表明,该分析检测到每个细胞1个拷贝的转录子的可信度达97%。利用这些转录子及其丰度的信息,绘制的虚拟Rot曲线与RNA-DNA饱和杂交所绘制的Rot曲线非常吻合,某些基因(SUP44/RPS4)的定量杂交分析结果也与SAGE所得出的丰度极为相似[11]。这些都表明SAGE分析能够真实、全面地反映酵母的基因表达 状况。此外,还发现了160多个在以前序列分析时未能预测出的开放读框(ORF),其中包括一些高丰度的基因,这对于酵母基因组是很好的补充。依据表达基因及其丰度与它们在染色体上的位置绘制的各染色体表达分布图,为分析染色体不同部位的转录活性以及基因的表达调控提供了重要而直观的信息,这也是迄今为止唯一以cDNA序列及丰度为基础的染色体的表达分布图。
, 百拇医药
2.2 SAGE在分析正常组织与癌组织基因表达差异中的应用 Zhang等[12]以结肠癌为例,用SAGE对正常与肿瘤细胞间的基因表达差异进行了分析。测序来自正常结肠粘膜、原发结肠癌、结肠癌细胞系、原发胰腺癌、胰腺癌细胞系共300 000多个tag,代表约50 000个基因,丰度自1~5000个拷贝/细胞不等。通过比较发现正常结肠细胞与肿瘤细胞及不同结肠癌细胞系之间,大多数基因表达水平相似,只有约500个基因在肿瘤细胞中呈10倍以上的明显改变,其中300个以上为已知,大多数在以往的文献中未见与肿瘤相关的表达改变的报道。通过对比胰腺癌、结肠癌与正常结肠粘膜,发现除胰腺癌特异表达的基因外,有约100多个基因表达水平在两种肿瘤中均呈上调,二者有47%的共性。这一研究结果除提供了从低丰度到高丰度基因在肿瘤细胞中的表达变化的信息外,还为解释每一个差异表达基因的意义提供了整体的框架,以便更好地了解它们的相对重要性,为的肿瘤发生机理与诊断治疗的应用性进一步研究奠定了良好的基础。
2.3 SAGE在研究由抑癌基因p53介导的细胞周期调控与凋亡中的应用 肿瘤抑制基因p53是人类肿瘤中最常发生突变的基因,它可以在DNA受损的细胞中引起细胞周期阻滞或凋亡[13,14],它主要是通过直接或间接调节其他一系列基因的转录来发挥作用。最近Madden等[15]应用SAGE对一个受Ha-ras和温度敏感p53转化的鼠胚胎成纤维细胞(REF)突变体模型,进行了诱导p53基因表达前后其他基因表达改变的分析。共检测了60 000个转录子,代表15 000个不同的基因,其中除8个在以前研究中已确定的P53蛋白诱导表达的基因外,还首次发现另外24个受P53蛋白调控的基因,利用这些tag作为探针分离到了一些与P53蛋白介导的细胞周期调控有关的新基因。Polyak等[16]通过分析100 000多个转录子,发现了14个由P53蛋白诱导表达的与凋亡相关的基因,并提出了P53所介导的凋亡的可能机理。Hermeking 等[17]对P53在G2/M阻滞作用的研究中,分析了电离辐射引起的G2/M阻滞前后共约50 000个转录子,它们来自约20 000个不同的基因,从中发现了与G2/M控制机理有关的基因14-3-3σ,并推测了在真核生物进化中保守和可能的G2/M监测点机理。SAGE技术在上述P53介导的基因表达改变的研究中的应用,显示其不仅可以从整体上分析细胞的基因表达状况,而且在了解与某些特殊生命现象相关的表达基因中也具有很大的潜能。
, 百拇医药
除上述应用外,SAGE还可用于对多个类型或多个发育阶段细胞基因表达模式的解剖分析,早期诊断的生物标志物及治疗靶点的发现,不同疗效患者基因表达差异的分析,药物的发现与筛选等多个领域,SAGE作为一种能够提供生物学和临床研究中最基本生物信息的技术,具有广阔的应用前景。
*通信联系人
参考文献
1 Collins FS,Patrinos A,Jordan E,et al.New goals for the US human genome project:1998-2003.Science,1998,282(5389)∶682-689.
2 Diatchenko L,Lau YC,Campbell AP,et al.Suppression subtractive hybridization:a method for generating differentially regulated or tissue-specific cDNA probes and libraries.Proc Natl Acad Sci U S A ,1996,93(12)∶6025-6030.
, 百拇医药
3 Liang P,pardee AB.Differential display of eukaryotic messenger RNA by means of the polymerase chain reaction.Science,1992,257(5072)∶967-971.
4 Bertiolo DJ,Schlichter UH,Adams MJ,et al.An analysis of differential display shows a strong bias towards high copy number mRNAs.Nucleic Acids Res,1995,23(21)∶4520-4523.
5 Adams MD,Kelley JM,Gocayne JD,et al.Complementary DNA sequencing:expres-sed sequence tags and human genome project.Science,1991,252(5013)∶1651-1656.
, 百拇医药
6 WilleyJC,Crawford EL,Jackson CM,et al.Expression measurement of many genes simultaneously by quantitative RT-PCR using standardized mixtures of competitive templates.Am J Resir Cell Mol Biol,1998,19(1)∶6-17.
7 Velculescu VE,Zhang L,Vogelstein B,et al.Serial analysis of gene expression.Science,1995,270(5235)∶484-487.
8 Bertelsen AH,Velculescu VE.High-throughput gene expression analysis using SAGE.Elsevier trends J,1998,3(4)∶152-159.
, 百拇医药
9 Velculescu VE,Zhang L,Zhou W,et al.Characterization of the yeast trans-criptome.Cell,1997,88(2)∶243-251.
10 Goffeau A,Barrell BG,Bussey H,et al.Life with 6000 genes.Science,1996,274(5287)∶546-567.
11 Lyer V,Struhl K.Absolute mRNA levels and transcriptional initiation rates in saccharomyces cerevisiae.Proc Natl Acad Sci U S A,1996,93(11)∶5208-5212.
12 Zhang L,Zhou W,Velculescu VE,et al.Gene expression profiles in normal and cancer cells.Science,1997,276(5316)∶1268-1272.
, http://www.100md.com
13 Kastan MB.Participation of P53 protein in the cellular response to DNA damage.Cancer Res,1991,51(23 pt 1)∶6304-6311.
14 Shaw P,Bovey R,Tardy S,et al.Introduction of apoptosis by wild-type p53 in a human colon tumor-derived cell line.Proc Natl Acad Sci U S A,1992,89(10)∶4495-4499.
15 Madden SL,Gallella EA,Zhu J,et al.SAGE transcript profiles for p53-dependent growth regulation.Oncogene,1997,15(9)∶1079-1085.
16 Polyak K,Xia Y,Zweier JL,et al.A model for p53-induced apoatosis.Nature,1997,389(6648)∶300-305.
17 Hermeking H,Lengauer C,Polyak K,et al.14-3-3 is a p53-regulated inhibitor of G2/M progression.Mol Cell,1997,1(1)∶3-11.
(收稿:1999-02-20 修回:1999-06-23), 百拇医药(周津 王秀琴 刘芝华 吴旻*)
单位:100021北京,中国医学科学院肿瘤研究所分子肿瘤学国家重点实验室;
关键词:
一种基因表达的系统分析的方法 随着人类基因组全部核苷酸测序计划的即将完成,研究的焦点逐渐由结构基因组学转向功能基因组学,而全基因组表达分析也成为功能基因组研究的重要组成部分[1]。以往表达序列标签(EST)测序、mRNA差异显示、消减杂交及差异杂交等在克隆差异表达基因方面做出了巨大贡献,但由于它们的随机性均无法描绘出基因表达模式的全貌,所获得的多限于高丰度或表达差异大的基因[2-5],且无法得知丰度的信息以及与其他差异表达基因相比的相对重要性。而目前用于定量表达分析的Northern杂交、逆转录PCR、竞争PCR(competitive PCR)等方法[6],分析的基因数量很有限。DNA芯片技术虽可一次分析大量基因的表达状况,但其价格昂贵且对设备有特殊要求,目前尚不能推广。1995年Velculescu等[7]发表了基因表达的系统分析(serial analysis of gene expression,SAGE),这是目前唯一以测序为基础的定量分析全基因组表达模式的技术,它充分利用了已有的测序结果,在定量分析各转录子表达水平及检测低丰度表达基因方面具有明显的优势,而且在有PCR仪、手工测序设备及计算机的实验室均可实施。该技术已成功地用于酵母转录组分析、正常与肿瘤细胞表达模式比较,以及p53诱导的凋亡机理的研究中。现就SAGE原理及有代表性的应用实例进行综述。
, 百拇医药
1 SAGE的原理[7]
SAGE的主要原理:(1)以转录子特定区域的一个9~11bp的寡核苷酸短序列(即标签,tag)特异地代表该转录子。因为一个10bp的寡核苷酸有1 000 000多种(410)排列方式,而目前所知人类基因组也只有大约100 000个不同的转录子,这种10bp标签所代表的复杂性超出了人类以及任何其他哺乳动物细胞中的基因数目,它含有足够的信息代表该转录子。(2)将多个短序列标签串连在一个克隆中,通过测序以连续的方式同时分析多个标签及其代表的转录子,明显提高了分析效率。(3)根据测序结果,计算某一标签出现的次数来确定相应转录子的拷贝数与表达水平。
这一技术的基本流程见图1[8]。该项技术中的两个酶:锚定酶(anchoring enzyme,AE)常采用4个碱基识别位点的限制性内切酶,因为理论上每256个碱基就有1个这种酶的切点,使绝大多数转录子至少被切割1次;标签酶(tagging enzyme,TE)为Ⅱs型限制性内切酶(如BsmFⅠ),其切点距识别位点10~14bp,切割后即产生与连接子相连的9~11bp的短cDNA tag。在连接与扩增步骤(5、6)中,只有tag呈尾-尾相连(ditag)的分子才能得到扩增,(因为PCR的引物是依据连接子的序列设计的),这一步骤除扩增ditag的数目外,对于最后分析中确定tag的方向,ditag之间的界限均具有重要意义。最后所得测序结果用SAGE软件包进行分析,从粗序列中提取tag序列及其拷贝数的信息,每一个tag可通过对比已知核苷酸序列数据库确认其所代表的基因。筛选cDNA文库获得全长。

, 百拇医药
图1 SAGE方法流程图
Velculescu等[7]最早应用SAGE分析了胰腺细胞的基因表达,测定了1 000个tag,通过比较核苷酸数据库,发现95%以上的9bp tag具有很好的代表性,以一些胰腺特异高表达的基因为探针筛选来自同一组织标本的cDNA文库,所获得的克隆数与SAGE分析得出的相对丰度相吻合,表明SAGE能够良好地反映原初组织中的表达状况,而且用tag序列作寡核苷酸探针筛选cDNA文库,也获得了良好的杂交信号。
2 SAGE的应用
2.1 应用SAGE进行转录组分析 所谓转录组(transcriptome)是指一定类型细胞内所有转录的基因及其丰度,它决定着细胞的表型,与基因组不同,转录组是不稳定的,不同种类的细胞具有不同的转录组。1997年,Velculescu等[9]应用SAGE成功地分析了酵母的转录组,这也是迄今为止第1个详尽的真核生物转录组。酵母基因组经序列分析预计有6121个基因[10]。应用SAGE对酵母在3种生长状态下——对数生长期、S期阻滞和G2/M阻滞的表达状况进行分析,测序共获得60 633个tag,其中93%(56 291)代表来自酵母基因组的转录子,对应于4665个表达基因,占酵母基因总数的75%,丰度每个细胞0.3~200个拷贝不等,其余来自线粒体或质粒。Monte Carlo分析表明,该分析检测到每个细胞1个拷贝的转录子的可信度达97%。利用这些转录子及其丰度的信息,绘制的虚拟Rot曲线与RNA-DNA饱和杂交所绘制的Rot曲线非常吻合,某些基因(SUP44/RPS4)的定量杂交分析结果也与SAGE所得出的丰度极为相似[11]。这些都表明SAGE分析能够真实、全面地反映酵母的基因表达 状况。此外,还发现了160多个在以前序列分析时未能预测出的开放读框(ORF),其中包括一些高丰度的基因,这对于酵母基因组是很好的补充。依据表达基因及其丰度与它们在染色体上的位置绘制的各染色体表达分布图,为分析染色体不同部位的转录活性以及基因的表达调控提供了重要而直观的信息,这也是迄今为止唯一以cDNA序列及丰度为基础的染色体的表达分布图。
, 百拇医药
2.2 SAGE在分析正常组织与癌组织基因表达差异中的应用 Zhang等[12]以结肠癌为例,用SAGE对正常与肿瘤细胞间的基因表达差异进行了分析。测序来自正常结肠粘膜、原发结肠癌、结肠癌细胞系、原发胰腺癌、胰腺癌细胞系共300 000多个tag,代表约50 000个基因,丰度自1~5000个拷贝/细胞不等。通过比较发现正常结肠细胞与肿瘤细胞及不同结肠癌细胞系之间,大多数基因表达水平相似,只有约500个基因在肿瘤细胞中呈10倍以上的明显改变,其中300个以上为已知,大多数在以往的文献中未见与肿瘤相关的表达改变的报道。通过对比胰腺癌、结肠癌与正常结肠粘膜,发现除胰腺癌特异表达的基因外,有约100多个基因表达水平在两种肿瘤中均呈上调,二者有47%的共性。这一研究结果除提供了从低丰度到高丰度基因在肿瘤细胞中的表达变化的信息外,还为解释每一个差异表达基因的意义提供了整体的框架,以便更好地了解它们的相对重要性,为的肿瘤发生机理与诊断治疗的应用性进一步研究奠定了良好的基础。
2.3 SAGE在研究由抑癌基因p53介导的细胞周期调控与凋亡中的应用 肿瘤抑制基因p53是人类肿瘤中最常发生突变的基因,它可以在DNA受损的细胞中引起细胞周期阻滞或凋亡[13,14],它主要是通过直接或间接调节其他一系列基因的转录来发挥作用。最近Madden等[15]应用SAGE对一个受Ha-ras和温度敏感p53转化的鼠胚胎成纤维细胞(REF)突变体模型,进行了诱导p53基因表达前后其他基因表达改变的分析。共检测了60 000个转录子,代表15 000个不同的基因,其中除8个在以前研究中已确定的P53蛋白诱导表达的基因外,还首次发现另外24个受P53蛋白调控的基因,利用这些tag作为探针分离到了一些与P53蛋白介导的细胞周期调控有关的新基因。Polyak等[16]通过分析100 000多个转录子,发现了14个由P53蛋白诱导表达的与凋亡相关的基因,并提出了P53所介导的凋亡的可能机理。Hermeking 等[17]对P53在G2/M阻滞作用的研究中,分析了电离辐射引起的G2/M阻滞前后共约50 000个转录子,它们来自约20 000个不同的基因,从中发现了与G2/M控制机理有关的基因14-3-3σ,并推测了在真核生物进化中保守和可能的G2/M监测点机理。SAGE技术在上述P53介导的基因表达改变的研究中的应用,显示其不仅可以从整体上分析细胞的基因表达状况,而且在了解与某些特殊生命现象相关的表达基因中也具有很大的潜能。
, 百拇医药
除上述应用外,SAGE还可用于对多个类型或多个发育阶段细胞基因表达模式的解剖分析,早期诊断的生物标志物及治疗靶点的发现,不同疗效患者基因表达差异的分析,药物的发现与筛选等多个领域,SAGE作为一种能够提供生物学和临床研究中最基本生物信息的技术,具有广阔的应用前景。
*通信联系人
参考文献
1 Collins FS,Patrinos A,Jordan E,et al.New goals for the US human genome project:1998-2003.Science,1998,282(5389)∶682-689.
2 Diatchenko L,Lau YC,Campbell AP,et al.Suppression subtractive hybridization:a method for generating differentially regulated or tissue-specific cDNA probes and libraries.Proc Natl Acad Sci U S A ,1996,93(12)∶6025-6030.
, 百拇医药
3 Liang P,pardee AB.Differential display of eukaryotic messenger RNA by means of the polymerase chain reaction.Science,1992,257(5072)∶967-971.
4 Bertiolo DJ,Schlichter UH,Adams MJ,et al.An analysis of differential display shows a strong bias towards high copy number mRNAs.Nucleic Acids Res,1995,23(21)∶4520-4523.
5 Adams MD,Kelley JM,Gocayne JD,et al.Complementary DNA sequencing:expres-sed sequence tags and human genome project.Science,1991,252(5013)∶1651-1656.
, 百拇医药
6 WilleyJC,Crawford EL,Jackson CM,et al.Expression measurement of many genes simultaneously by quantitative RT-PCR using standardized mixtures of competitive templates.Am J Resir Cell Mol Biol,1998,19(1)∶6-17.
7 Velculescu VE,Zhang L,Vogelstein B,et al.Serial analysis of gene expression.Science,1995,270(5235)∶484-487.
8 Bertelsen AH,Velculescu VE.High-throughput gene expression analysis using SAGE.Elsevier trends J,1998,3(4)∶152-159.
, 百拇医药
9 Velculescu VE,Zhang L,Zhou W,et al.Characterization of the yeast trans-criptome.Cell,1997,88(2)∶243-251.
10 Goffeau A,Barrell BG,Bussey H,et al.Life with 6000 genes.Science,1996,274(5287)∶546-567.
11 Lyer V,Struhl K.Absolute mRNA levels and transcriptional initiation rates in saccharomyces cerevisiae.Proc Natl Acad Sci U S A,1996,93(11)∶5208-5212.
12 Zhang L,Zhou W,Velculescu VE,et al.Gene expression profiles in normal and cancer cells.Science,1997,276(5316)∶1268-1272.
, http://www.100md.com
13 Kastan MB.Participation of P53 protein in the cellular response to DNA damage.Cancer Res,1991,51(23 pt 1)∶6304-6311.
14 Shaw P,Bovey R,Tardy S,et al.Introduction of apoptosis by wild-type p53 in a human colon tumor-derived cell line.Proc Natl Acad Sci U S A,1992,89(10)∶4495-4499.
15 Madden SL,Gallella EA,Zhu J,et al.SAGE transcript profiles for p53-dependent growth regulation.Oncogene,1997,15(9)∶1079-1085.
16 Polyak K,Xia Y,Zweier JL,et al.A model for p53-induced apoatosis.Nature,1997,389(6648)∶300-305.
17 Hermeking H,Lengauer C,Polyak K,et al.14-3-3 is a p53-regulated inhibitor of G2/M progression.Mol Cell,1997,1(1)∶3-11.
(收稿:1999-02-20 修回:1999-06-23), 百拇医药(周津 王秀琴 刘芝华 吴旻*)