传染性法氏囊病病毒中国强毒株A节段cDNA基因的克隆和序列分析
作者:胡子信 张曼夫
单位:中国农业大学生物化学与分子生物学系,北京 100094
关键词:传染性法氏囊病病毒A节段cDNA;反转录聚合酶联反应(RT-PCR);分子克隆;序列分析
病毒学报990406 摘要:以来自哈尔滨传染性法氏囊病病毒(IBDV)强毒株(Harbin毒株,H)的基因组RNA为模板,用反转录聚合酶链反应(RT-PCR)的方法得到了其A节段的全长cDNA片段,分5'端(1 659bp)和3'端(1 444bp)上下两段分别克隆到pGEMB®-T载体上,测定了其核苷酸顺序,在长为3 101 bp中含有两个阅读框ORF A1和ORF A2,分别编码1 012个氨基酸的前体蛋白(VP2-4-3)和145个氨基酸的VP5,ORF A1和ORF A2有部分的重叠。将核苷酸序列及推测出的氨基酸序列与已报道的IBDV血清Ⅰ型和Ⅱ型毒株的相应序列进行了比较,结果表明:H毒株与其它血清Ⅰ型毒株之间,在核苷酸水平上存在25bp-267bp的差异;在氨基酸水平上存在17~40个氨基酸的差异。在VP2-4-3内比较显示,H毒株与P2、Cu-1之间氨基酸的差异最小为1.7%,H毒株与UK661之间氨基酸的差异最大为3.9%。变异主要发生在VP2的可变区(206-350位氨基酸),在H毒株所特有的12个氨基酸当中,该区就占5个,代表1.76%的变异。VP4、VP3和VP5区各有两个特有氨基酸的替代,分别代表0.74%、0.69%和1.38%的变异。序列分析显示,H毒株在抗原性和毒力上均存在着变异,然而,它既不同于欧洲、日本和以色列分离的超强毒,也不同于美国的变异株。与其亲缘关系最近的是欧洲的较早期的分离株,但是在可变区,尤其是在两个亲水区又有明显的突变。
, http://www.100md.com
中图分类号:S858.31;Q343.1 文献标识码:A 文章编号:1000-8721(1999)04-0330-09
CLONING AND SEQUENCE ANALYSIS OF SEGMENT A cDNA OF A VERY VIRULENT INFECTIOUS BURSAL DISEASE VIRUS
HU Zi-xin, ZHANG Man-fu
(Department of Molecular Biology and Biochemistry, China Agricultural University, Beijing 100094, China)
Abstract: By RT-PCR, the nucleotide sequences of the Harbin vvIBDV segment A, two ORFS for VP5 and precursor polyprotein were determined. The results showed that the cloned 3101bp segment A gene contains two overlapping open reading frames (ORFA1 and ORFA2). The large ORF of 3036bp encodes a polyprotein (VP2-4-3), whereas the small ORF is 435bp coding VP5. The deduced amino acid sequence of the Harbin strain was compared with those of other serotype 1 and 2 strains. The results showed that the sequence of the Chinese isolate, the Harbin strain, has variations for virulence and antigenecity. There are 25 to 267 nucleotide differences among segment A of Harbin strain and the other serotype I strains of IBDV. The polyprotein encoded by Harbin strain showed 17 to 40 amino acid differences from those of the other serotype I strains, with 12 substitutions unique to Harbin strain. The minimum amino acid divergence within polyprotein (1.7%) was observed among Harbin strain and P2, Cu-1 of serotype 1. The maximum amino acid difference was 3.9% observed between Harbin strain and Uk661. Within the 454 residues of VP2 of Harbin strain, there are 5 amino acid substitutions, representing 1.76% variation at the VP2 variable region between residues 206 to 350. Within the 267 residues of VP4, there are only 2 amino acid substitutions, representing 0.74% variation. Within the 289 residues of VP3, there are also 2 amino acid substitutions, representing 0.69% variation. Within the 145 residues of VP5, there are 2 amino acid substitutions, representing 1.38% variation. Harbin strain is neither similar to the vv strains from Europe, Japan and Israel nor to the variant strains from the United States and China Guanzhou. It is most closely related to the strains isolated earlier from Europe, however has distinct amino acid mutations in variable region, especially in two hydrophilic domains.
, 百拇医药
Key words: Infectious bursal disease virus; Segment A; Reverse transcription-polymerase chain reaction; Sequence and phylogenetic analysis
鸡传染性法氏囊病(Infectious bursal disease,IBD)是幼鸡的一种高度免疫抑制性疾病,在世界范围内引起了严重的经验损失。IBDV属于双链双节段RNA病毒科,基因组由A和B两个节段组成,包括5'端非编码区、编码区和3'端非编码区。A节段(3.3~3.4kb)包括两个完整的阅读框架ORFA1和ORFA2,分别编码VP2-4-3和VP5;B节段(2.8~2.9kb)编码VP1。VP1是病毒RNA聚合酶,它与病毒RNA复制有关。VP2和VP3是病毒的主要结构蛋白,共同构成病毒衣壳,是主要的宿主保护性抗原。VP2是血清型特异性抗原,VP3为群特异性抗原。抗原变异主要发生在VP2的可变区,VP4在病毒蛋白成熟过程中起重要作用,VP5功能目前尚不清楚[1,2]。
, http://www.100md.com
近年来在欧洲和亚洲由于超强毒(vvIBDV)的出现,该病毒已经成为包括中国在内的世界性引起养鸡业经济损失的最重要病原。然而,vvIBDV高致病性的分子基础及其起源尚不完全清楚。本研究旨在对中国近年分离到的强毒IBDV-H毒株进行分子结构分析。通过反转录聚合酶链反应(RT-PCR)扩增A节段的cDNA基因,在克隆和序列分析的基础上,与已报导的Ⅰ型毒株、变异株及Ⅱ型毒株的A节段序列进行比较,构建系统发育树,进而从分子结构上来确定IBDV-H毒株的抗原和毒力的基础,寻找vvIBDV的起源;通过分析IBDV-H毒株和其他Ⅰ型毒株、变异株及Ⅱ型毒株的相互关系,更好地理解病毒的传播方式。不同国家和地区IBDV毒株之间差异的鉴定,可以帮助我们建立准确的诊断方法,正确地使用疫苗,也为研制开发新的有效的疫苗打下基础。
材料与方法
1 IBDV-H毒株 由中国农业科学院哈尔滨兽医研究所陈冠春等于1992年从哈尔滨地区某鸡场的病鸡法氏囊中分离得到,是近年来在中国大陆分离的典型强毒之一。故选该毒株经4周龄SPF雏鸡繁殖作为实验材料。
, 百拇医药
2 病毒纯化和病毒基因组dsRNA的提取 以IBDV-H毒株接种4周龄SPF雏鸡,接种后96小时收取法氏囊,以超速离心纯化病毒后,用酚-氯仿抽提提取病毒RNA[1]。
3 RT-PCR引物设计 参见文献[2]。
4 病毒基因组dsRNA的反转录和聚合酶链反应(PCR) 以病毒基因组dsRNA为模板,以P1为引物,按Gibco BRL superscript preamplification system提供的方法,合成了全长A节段cDNA,以此cDNA为模板,利用PCR反应,按Gibco BRL Enlongase polymerase提供的方法,放大扩增得到有部分重叠的上下两段PCR产物,其长度分别为1 659bp(37~1 696)和1 444bp(1 689~3 133)。
5 cDNA克隆和序列分析 将上下两段PCR产物分别进行纯化,再克隆到pGEMB®-T载体(Promega Co.)上,选取经酶切及PCR鉴定后的阳性克隆进行序列测定。采用渐进法(prime walking),即5'端和3'端分别先用通用引物进行反应,而后依据两端测出序列,另外设计中间段测序引物,再进行测序反应,共计7段结果拼接出A节段全长cDNA序列。为保证测序结果的准确,全部测序反应重复两次。测序时模板的制备采用PEG法,利用ABI377自动测序仪。
, 百拇医药
6 序列编辑和分析 利用DNAsis(HITACHI SOFTWARE.ENGINEERING CO.LTD,1991)、GeneDoc(Nicholas,K.B.,Nicholas H.B.Jr.,and Deerfield,D.W.Ⅱ.1997)和DNASTAR(DNASTAR,INC.Madison,Wisconsin.Fourth Edition,October 1997,12)等软件,对所测定的IBDV-A节段的核苷酸序列及推测出的氨基酸序列进行编辑,并与已报道的IBDV血清Ⅰ型和Ⅱ毒株的相应序列进行同源分析。比较时所引用参考序列及其Genbank接受号包括:Ⅰ型,Harbin AF092171、UK661 X92760、OKYM D49706、52/70 D00869、Cu-1X16107、P2 X84034、STC D00499、GLS M71346、GZ29112 AF051837、002-73 X03993;Ⅱ型,OHM66722、23/82 Z21971[3-9]。
结果
, http://www.100md.com
1 IBDV-H毒株A节段的序列分析
用RT-PCR方法得到了IBDV-H毒株A节段的全长cDNA,分5'端(1 659bp)和3'端(1 444bp)上下两段分别克隆到pGEMBR-T载体上,测定了其核苷酸顺序。为了保证结果的正确性,所有测序反应重复两次。使用DNAsis和DNASTAR软件分析显示,在长为3 101bp核苷酸序列中,含有两个阅读框ORF1和ORF2,分别编码1 012个氨基酸的前体蛋白(VP2-4-3)和145个氨基酸的VP5,ORFA1和ORFA2有部分的重叠。整个序列既没有缺失也没有插入。此核苷酸序列已被GenBank收录,接受号是AF092171。
图1列出了12个毒株的ORF A1氨基酸序列的比较,包括超强毒UK661、OKYM和Harbin,经典Ⅰ型毒株52/70、Cu-1、P2、STC和002-73,变异株GLS和GZ29112,以及血清Ⅱ型毒株23/82和OH。与其它血清Ⅰ型毒株相比,H毒株在核苷酸水平上有25~267个核苷酸的差异;在氨基酸水平上,有17~40个氨基酸的差异,其中有12个氨基酸为H毒株所特有,它们分别位于19(P)、129(L)、178(G)、213(G)、216(L)、267(G)、290(S)、300(G)、606(R)、611(H)、917(L)和985(M);其中8个是在VP2;5个在可变区;2个(606和611)在VP4;2个(917和985)在VP3。还有6个氨基酸,即79(S)、279(N)、316(R)、330(R)、541(V)和981(L),虽然不是H毒株本身所特有,但是也只能在个别毒株才见到。
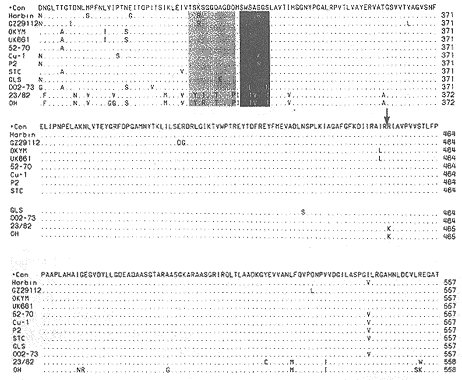

, http://www.100md.com
图1 IBDV-H毒株及参考毒株的VP2-4-3氨基酸序列的比较
氨基酸一致用点表示,列出的是不一致氨基酸;两个亲水区用阴影表示,黑阴影代表七肽区,两个箭头分别表示VP2和VP4,VP4和VP3之间的裂解点。
Figure 1 Alignment of the deduced amino acid sequence of the segment A precursor polyprotein(VP2-VP4-VP3) of the China vv Harbin isolate to the corresponding sequences of published IBDV strains
The alignment is shown as a PRETTYPLOT, in which non-identical residues to a consensus are shown and the identical residues are indicated by dots. Proposed processing sites and two hydrophilic regions are marked with shadows. The heptapeptide is indicated by black shadows(*Con:Consensus).
, http://www.100md.com
就VP2-4-3而言,在所比较的Ⅰ型毒株之间,H毒株与Cu-1和P2差异最小,为1.7%;H毒株与UK661差异最大,为3.9%。在VP2区的454个氨基酸残基中就有5个氨基酸的变异,变异率为1.76%,并且主要发生在可变区206~350之间(D-G213、F-L216、D-G267、M-S290和E-G300);在VP4区的267个氨基酸残基中有2个氨基酸的替代(H-R606和Y-H611),变异率为0.74%;在VP3区的289个氨基酸残基中也有2个氨基酸的替代(S-L917和K-M985),变异率为0.69%。
从VP5来看,与其它6个毒株相比,145个氨基酸中有2个(K-E14、L-P140)发生变异,其变异率为1.38%。而这两个替代均发生在ORF A1(VP2-4-3)和ORF A2(VP5)重叠部分,其中第14位K-E的突变只发生在VP5,在VP2-4-3并没有突变;而第140位L-P的突变不仅是VP5,而且VP2-4-3也同时发生了突变(图2)。
, http://www.100md.com
图2 IBDV-H毒株及参考毒株的VP5氨基酸序列的比较
氨基酸一致用点表示,列出的是不一致的氨基酸。
Figure 2 Multiple sequence alignments of deduced amino acid sequence of VP5 encoded by small open reading frame of segment A of IBDV strains: Harbin, P2, Cu-1, UK661, STC, 52/70 and 23/82
Differences from the consensus sequence are shown and dots indicate the same residues as consensus.
2 IBDV-H毒株与其它毒株的亲缘关系分析
, http://www.100md.com
将IBDV-H毒株的氨基酸序列与已报道的IBDV血清Ⅰ型、变异株和Ⅱ型毒株的相应序列进行了同源分析,结果是:从整个ORF A1(VP2-4-3)来看,H毒株和P2、Cu-1同源性最高,其亲缘关系最近(图3a),而从每个单一蛋白VP2、VP4和VP3,包括ORF A2(VP5)同源比较所得出的系统树,其结果均是一致的(图3b、c、d、e)。
图3 IBDV各毒株的亲缘关系图谱
Figure 3 Phylogenetic tree of strains of IBDV
讨论
通过反转录得到了IBDV-H毒株A节段的全长cDNA片段,用PCR分5'端(1659bp)和3'端(1444 bp)上下两段扩增得到PCR产物,分别克隆到pGEMB®-T载体上,测定了其核苷酸顺序。在长为3 101个bp中含有两个阅读框ORF A1和ORF A2,分别编码1 012个氨基酸的前体蛋白(VP2-4-3)和145个氨基酸的VP5,ORF A1和ORF A2有部分的重叠。将核苷酸序列及推测出的氨基酸序列与已报道的IBDV血清Ⅰ型、变异株和Ⅱ型毒株的相应序列进行了比较,结果表明,中国分离的强毒株H毒株,其决定毒力和抗原性的序列均有变异,但它既不同于欧洲[3,10]、日本和以色列的超强毒[4,9],也不同于美国和中国广州的变异株[11,12]。其亲缘关系是和欧洲较早时期分离的毒株最近,但在可变区,特别是在两个亲水区呈现了明显的突变。
, 百拇医药
据有关文献报道,超强毒IBDV(vvIBDV)如欧洲的UK661、日本的OKYM等,222位的A、256位的Ⅰ和294位的Ⅰ是保守的[3,12-14],国内陈红英等人[15](1998)对IBDV AH-2、HD96、B2/28、JS-18和D11-2株的VP2高变区的序列分析显示,这三个位置的氨基酸也是保守的,然而H-毒株却与此不同。
从世界各地分离的毒株的序列比较可以看出,不同毒株之间序列的变异主要发生在VP2区,尤其是206~350位氨基酸残基之间变异最大[11,16,17]。VP2是主要的保护性抗原[10]。VP3和VP4相对保守,不仅在同一血清型之间,甚至在两个血清型之间也很相似[5]。本研究发现的H毒株12个特有的氨基酸中,8个在VP2,其中有5个在可变区,2个位于第一个亲水区内(212-224),1个位于第二个亲水区内(314-324)。第一个亲水区与构象依赖性抗原表位的构象稳定有关,第二个亲水区为中和抗体的结合位点[13,18]。因此,在这两个区域如有变异很可能诱导抗原性的变化。经典Ⅰ型毒株中这两个亲水区的氨基酸序列是完全保守的,与经典Ⅰ型毒株相比,欧洲的UK661、日本的OKYM、中国的GZ29112株包括变异株GLS也只有1个氨基酸的替代,而H毒株有2个氨基酸的突变。在亲水区内氨基酸的替代也许与其毒力的增强有关。同时这些氨基酸的变化可能引起了抗原性的改变,造成具有较高水平母源抗体鸡的发病。大多数变异株在两个亲水区的确都发生了氨基酸的变化,但Miss株是个例外,在两个亲水区,Miss株与STC、52/70和PBG98等经典株具有完全相同的氨基酸序列[11]。因此可以说在其它位置的氨基酸的变化通过诱导VP2构象的改变进而导致抗原的变异[19]。
, 百拇医药
第二个亲水区之后的七肽区多年来被认为是IBDV毒力强弱的一个重要标志。强毒株具有相当保守的富含丝氨酸(S)的SWSASGS序列,弱毒株具有较少的丝氨酸,不具致病力的血清Ⅱ型OH和23/82在七肽区内有3个氨基酸发生变异[1,8,18,20,21]。但从我们的分析结果看,大部分经典株和国外的超强毒七肽区是保守的,血清Ⅰ型002-73株也有3个氨基酸发生变异,而中国的强毒H株使七肽区的SWSASGS序列变成了SWSARGS,这就意味着富含丝氨酸的七肽区内丝氨酸的突变可能不是毒力降低的唯一决定因素。
虽然中国强毒Harbin株的起源还不能定论,但从序列同源性比较的系统树显示是与欧洲的较早时期分离的毒株最接近。也许是恢复突变,也不能排除基因重组,要想进一步了解出现新的高致病性的IBDV的机制,还需做更详细的分子流行病学的研究。
基金项目:国家自然科学基金重大项目(39893290);北京市自然科学基金(5952004);国家教委博士点基金。
, http://www.100md.com
作者简介:胡子信(1963-),男(蒙古),副教授,博士研究生,主要从事动物病毒,免疫和分子生物学方面的教学和研究。
参考文献
1 Azad A A, Barret S A, Fahey K J. The characterization and molecular cloning of the double-stranded RNA genome of an Australian infectious bursal disease virus[J]. Virology, 1985, 143:35-44.
2 胡子信,张曼夫.传染性法氏囊病病毒A节段编码序列cDNA基因的克隆和序列分析[J].中国预防兽医学报,1999,3
3 Brown M D, Skinner M A. Coding sequences of both genome segments of a European ‘very virulent’isolate [J]. Virus Res, 1996, 40:1-15.
, 百拇医药
4 Yamaguchi T, Ogawa M, Inoshima Y, et al. Sequence and phylogenetic analysis of highly virulent infectious bursal disease virus[J]. Arch Virol, 1997, 142:154-172.
5 Bayliss C D, Spies U, Shaw K, et al. A comparison of the sequences of segment A of four infectious bursal disease virus strains and identification of a variable region in VP2 [J]. J Gen Virol, 1990, 71:1303-1312.
6 Dybing J K. Sequence analysis of IBDV of serotype 2 and expression of the VP5 cDNA [R]. M. Sc, University of Prince Edward Island, Charlottesville 1992.
, 百拇医药
7 Kibenge F S, Jackwood D J, Mercado C C. Nucleotide sequence analysis of genome segment A of infectious bursal disease virus[J]. J Gen Virol, 1990, 71: 569-577.
8 Heppell J, Berthiaume L, Corbin F, et al. Comparison of amino acid sequences deduced from a cDNA fragment obtained from infectious pancreatic necrosis virus (IPNV) strains of different serotypes[J]. Virology, 1993, 195:840-84.
9 Pitcovski J, Goldberg D, Levi B Z, et al. Coding region of segment A sequence of a very virulent isolate of IBDV-Comparison with isolates from different countries and virulence[J]. Avian Dis, 1998, 42:497-506.
, 百拇医药
10 Eterradossi N, Rivallan G, Toquin D, et al. Limited antigenic variation among recent infectious bursal disease virus isolates from France[J]. Arch Virol, 1997, 142: 2079-2087.
11 Dormitorio T V, Giambrone J J, Duck L W. Sequence conparison of the variable VP2 region of eight infectious bursal disease virus isolates[J]. Avian Dis, 1997, 41: 36-44.
12 Cao Y C, Yeung W S, Law M, et al. Molecular characterization of seven Chinese isolates of infectious bursal disease virus: classical, very virulent, and variant strains[J]. Avian Dis, 1998, 42: 340-351.
, 百拇医药
13 Jackwood D J, Sommer S E. Genetic heterogeneity in the VP2 gene of infectious bursal disease viruses detected in commercially reared chickens[J]. Avian Dis, 1998, 42: 321-339.
14 Yamaguchi T, Ogawa M, Inoshima Y, et al. Identification of sequence changes responsible for the attenuation of highly virulent infectious bursal disease virus[J]. Virology, 1996, 223: 219-223.
15 Chen H Y, Zhou Q, Zhang M. F. Sequence analysis of the VP2 hypervariable region of nine infectious bursal disease virus isolates from Mainland China[J]. Avian Dis 1998, 42: 762-769.
, 百拇医药
16 Brown M D, Green P, Skinner M A. VP2 sequences of recent European ‘very virulent’isolates of infectious bursal disease virus are closely related to each other but are distinct from those of ‘classical’strains[J]. J Gen Virol 1994, 75: 675-680.
17 Lana D P, Beisel C E, Silva R F. Genetic mechanisms of antigenic variation in infectious bursal disease virus: Analysis of a naturally occurring variant virus[J]. Virus Genes, 1992, 6(3): 247-259.
, 百拇医药
18 Heine H-G, Haritou M, Failla P. Sequence analysis and expression of the host-protective immunogen VP2 of a variant strain of infectious bursal disease virus, which can circumvent vaccination with standard type 1 strains[J]. J Gen Virol, 1991, 72: 1835-1843.
19 Van Den Berg TP, Gonze M, Morales D, et al. Acute infectious bursal disease in poultry:immunological and molecular basis of antigenicity of a highly virulent strain[J]. Avian Pathol, 1996, 25: 751-768.
, 百拇医药
20 Jackwood D J, Jackwood R J, Sommer SE. Identification and comparison of point mutations associated in classic and variant infectious bursal disease viruses[J]. Virus Res, 1997, 49: 131-137.
21 Vakharia V N, He J K, Ahamed B. Molecular basis of antigenic variation in infectious bursal disease virus[J]. Virus Res, 1994, 31: 265-273.
收稿日期:1998-12-09;修回日期:1999-03-26, 百拇医药
单位:中国农业大学生物化学与分子生物学系,北京 100094
关键词:传染性法氏囊病病毒A节段cDNA;反转录聚合酶联反应(RT-PCR);分子克隆;序列分析
病毒学报990406 摘要:以来自哈尔滨传染性法氏囊病病毒(IBDV)强毒株(Harbin毒株,H)的基因组RNA为模板,用反转录聚合酶链反应(RT-PCR)的方法得到了其A节段的全长cDNA片段,分5'端(1 659bp)和3'端(1 444bp)上下两段分别克隆到pGEMB®-T载体上,测定了其核苷酸顺序,在长为3 101 bp中含有两个阅读框ORF A1和ORF A2,分别编码1 012个氨基酸的前体蛋白(VP2-4-3)和145个氨基酸的VP5,ORF A1和ORF A2有部分的重叠。将核苷酸序列及推测出的氨基酸序列与已报道的IBDV血清Ⅰ型和Ⅱ型毒株的相应序列进行了比较,结果表明:H毒株与其它血清Ⅰ型毒株之间,在核苷酸水平上存在25bp-267bp的差异;在氨基酸水平上存在17~40个氨基酸的差异。在VP2-4-3内比较显示,H毒株与P2、Cu-1之间氨基酸的差异最小为1.7%,H毒株与UK661之间氨基酸的差异最大为3.9%。变异主要发生在VP2的可变区(206-350位氨基酸),在H毒株所特有的12个氨基酸当中,该区就占5个,代表1.76%的变异。VP4、VP3和VP5区各有两个特有氨基酸的替代,分别代表0.74%、0.69%和1.38%的变异。序列分析显示,H毒株在抗原性和毒力上均存在着变异,然而,它既不同于欧洲、日本和以色列分离的超强毒,也不同于美国的变异株。与其亲缘关系最近的是欧洲的较早期的分离株,但是在可变区,尤其是在两个亲水区又有明显的突变。
, http://www.100md.com
中图分类号:S858.31;Q343.1 文献标识码:A 文章编号:1000-8721(1999)04-0330-09
CLONING AND SEQUENCE ANALYSIS OF SEGMENT A cDNA OF A VERY VIRULENT INFECTIOUS BURSAL DISEASE VIRUS
HU Zi-xin, ZHANG Man-fu
(Department of Molecular Biology and Biochemistry, China Agricultural University, Beijing 100094, China)
Abstract: By RT-PCR, the nucleotide sequences of the Harbin vvIBDV segment A, two ORFS for VP5 and precursor polyprotein were determined. The results showed that the cloned 3101bp segment A gene contains two overlapping open reading frames (ORFA1 and ORFA2). The large ORF of 3036bp encodes a polyprotein (VP2-4-3), whereas the small ORF is 435bp coding VP5. The deduced amino acid sequence of the Harbin strain was compared with those of other serotype 1 and 2 strains. The results showed that the sequence of the Chinese isolate, the Harbin strain, has variations for virulence and antigenecity. There are 25 to 267 nucleotide differences among segment A of Harbin strain and the other serotype I strains of IBDV. The polyprotein encoded by Harbin strain showed 17 to 40 amino acid differences from those of the other serotype I strains, with 12 substitutions unique to Harbin strain. The minimum amino acid divergence within polyprotein (1.7%) was observed among Harbin strain and P2, Cu-1 of serotype 1. The maximum amino acid difference was 3.9% observed between Harbin strain and Uk661. Within the 454 residues of VP2 of Harbin strain, there are 5 amino acid substitutions, representing 1.76% variation at the VP2 variable region between residues 206 to 350. Within the 267 residues of VP4, there are only 2 amino acid substitutions, representing 0.74% variation. Within the 289 residues of VP3, there are also 2 amino acid substitutions, representing 0.69% variation. Within the 145 residues of VP5, there are 2 amino acid substitutions, representing 1.38% variation. Harbin strain is neither similar to the vv strains from Europe, Japan and Israel nor to the variant strains from the United States and China Guanzhou. It is most closely related to the strains isolated earlier from Europe, however has distinct amino acid mutations in variable region, especially in two hydrophilic domains.
, 百拇医药
Key words: Infectious bursal disease virus; Segment A; Reverse transcription-polymerase chain reaction; Sequence and phylogenetic analysis
鸡传染性法氏囊病(Infectious bursal disease,IBD)是幼鸡的一种高度免疫抑制性疾病,在世界范围内引起了严重的经验损失。IBDV属于双链双节段RNA病毒科,基因组由A和B两个节段组成,包括5'端非编码区、编码区和3'端非编码区。A节段(3.3~3.4kb)包括两个完整的阅读框架ORFA1和ORFA2,分别编码VP2-4-3和VP5;B节段(2.8~2.9kb)编码VP1。VP1是病毒RNA聚合酶,它与病毒RNA复制有关。VP2和VP3是病毒的主要结构蛋白,共同构成病毒衣壳,是主要的宿主保护性抗原。VP2是血清型特异性抗原,VP3为群特异性抗原。抗原变异主要发生在VP2的可变区,VP4在病毒蛋白成熟过程中起重要作用,VP5功能目前尚不清楚[1,2]。
, http://www.100md.com
近年来在欧洲和亚洲由于超强毒(vvIBDV)的出现,该病毒已经成为包括中国在内的世界性引起养鸡业经济损失的最重要病原。然而,vvIBDV高致病性的分子基础及其起源尚不完全清楚。本研究旨在对中国近年分离到的强毒IBDV-H毒株进行分子结构分析。通过反转录聚合酶链反应(RT-PCR)扩增A节段的cDNA基因,在克隆和序列分析的基础上,与已报导的Ⅰ型毒株、变异株及Ⅱ型毒株的A节段序列进行比较,构建系统发育树,进而从分子结构上来确定IBDV-H毒株的抗原和毒力的基础,寻找vvIBDV的起源;通过分析IBDV-H毒株和其他Ⅰ型毒株、变异株及Ⅱ型毒株的相互关系,更好地理解病毒的传播方式。不同国家和地区IBDV毒株之间差异的鉴定,可以帮助我们建立准确的诊断方法,正确地使用疫苗,也为研制开发新的有效的疫苗打下基础。
材料与方法
1 IBDV-H毒株 由中国农业科学院哈尔滨兽医研究所陈冠春等于1992年从哈尔滨地区某鸡场的病鸡法氏囊中分离得到,是近年来在中国大陆分离的典型强毒之一。故选该毒株经4周龄SPF雏鸡繁殖作为实验材料。
, 百拇医药
2 病毒纯化和病毒基因组dsRNA的提取 以IBDV-H毒株接种4周龄SPF雏鸡,接种后96小时收取法氏囊,以超速离心纯化病毒后,用酚-氯仿抽提提取病毒RNA[1]。
3 RT-PCR引物设计 参见文献[2]。
4 病毒基因组dsRNA的反转录和聚合酶链反应(PCR) 以病毒基因组dsRNA为模板,以P1为引物,按Gibco BRL superscript preamplification system提供的方法,合成了全长A节段cDNA,以此cDNA为模板,利用PCR反应,按Gibco BRL Enlongase polymerase提供的方法,放大扩增得到有部分重叠的上下两段PCR产物,其长度分别为1 659bp(37~1 696)和1 444bp(1 689~3 133)。
5 cDNA克隆和序列分析 将上下两段PCR产物分别进行纯化,再克隆到pGEMB®-T载体(Promega Co.)上,选取经酶切及PCR鉴定后的阳性克隆进行序列测定。采用渐进法(prime walking),即5'端和3'端分别先用通用引物进行反应,而后依据两端测出序列,另外设计中间段测序引物,再进行测序反应,共计7段结果拼接出A节段全长cDNA序列。为保证测序结果的准确,全部测序反应重复两次。测序时模板的制备采用PEG法,利用ABI377自动测序仪。
, 百拇医药
6 序列编辑和分析 利用DNAsis(HITACHI SOFTWARE.ENGINEERING CO.LTD,1991)、GeneDoc(Nicholas,K.B.,Nicholas H.B.Jr.,and Deerfield,D.W.Ⅱ.1997)和DNASTAR(DNASTAR,INC.Madison,Wisconsin.Fourth Edition,October 1997,12)等软件,对所测定的IBDV-A节段的核苷酸序列及推测出的氨基酸序列进行编辑,并与已报道的IBDV血清Ⅰ型和Ⅱ毒株的相应序列进行同源分析。比较时所引用参考序列及其Genbank接受号包括:Ⅰ型,Harbin AF092171、UK661 X92760、OKYM D49706、52/70 D00869、Cu-1X16107、P2 X84034、STC D00499、GLS M71346、GZ29112 AF051837、002-73 X03993;Ⅱ型,OHM66722、23/82 Z21971[3-9]。
结果
, http://www.100md.com
1 IBDV-H毒株A节段的序列分析
用RT-PCR方法得到了IBDV-H毒株A节段的全长cDNA,分5'端(1 659bp)和3'端(1 444bp)上下两段分别克隆到pGEMBR-T载体上,测定了其核苷酸顺序。为了保证结果的正确性,所有测序反应重复两次。使用DNAsis和DNASTAR软件分析显示,在长为3 101bp核苷酸序列中,含有两个阅读框ORF1和ORF2,分别编码1 012个氨基酸的前体蛋白(VP2-4-3)和145个氨基酸的VP5,ORFA1和ORFA2有部分的重叠。整个序列既没有缺失也没有插入。此核苷酸序列已被GenBank收录,接受号是AF092171。
图1列出了12个毒株的ORF A1氨基酸序列的比较,包括超强毒UK661、OKYM和Harbin,经典Ⅰ型毒株52/70、Cu-1、P2、STC和002-73,变异株GLS和GZ29112,以及血清Ⅱ型毒株23/82和OH。与其它血清Ⅰ型毒株相比,H毒株在核苷酸水平上有25~267个核苷酸的差异;在氨基酸水平上,有17~40个氨基酸的差异,其中有12个氨基酸为H毒株所特有,它们分别位于19(P)、129(L)、178(G)、213(G)、216(L)、267(G)、290(S)、300(G)、606(R)、611(H)、917(L)和985(M);其中8个是在VP2;5个在可变区;2个(606和611)在VP4;2个(917和985)在VP3。还有6个氨基酸,即79(S)、279(N)、316(R)、330(R)、541(V)和981(L),虽然不是H毒株本身所特有,但是也只能在个别毒株才见到。



, http://www.100md.com
图1 IBDV-H毒株及参考毒株的VP2-4-3氨基酸序列的比较
氨基酸一致用点表示,列出的是不一致氨基酸;两个亲水区用阴影表示,黑阴影代表七肽区,两个箭头分别表示VP2和VP4,VP4和VP3之间的裂解点。
Figure 1 Alignment of the deduced amino acid sequence of the segment A precursor polyprotein(VP2-VP4-VP3) of the China vv Harbin isolate to the corresponding sequences of published IBDV strains
The alignment is shown as a PRETTYPLOT, in which non-identical residues to a consensus are shown and the identical residues are indicated by dots. Proposed processing sites and two hydrophilic regions are marked with shadows. The heptapeptide is indicated by black shadows(*Con:Consensus).
, http://www.100md.com
就VP2-4-3而言,在所比较的Ⅰ型毒株之间,H毒株与Cu-1和P2差异最小,为1.7%;H毒株与UK661差异最大,为3.9%。在VP2区的454个氨基酸残基中就有5个氨基酸的变异,变异率为1.76%,并且主要发生在可变区206~350之间(D-G213、F-L216、D-G267、M-S290和E-G300);在VP4区的267个氨基酸残基中有2个氨基酸的替代(H-R606和Y-H611),变异率为0.74%;在VP3区的289个氨基酸残基中也有2个氨基酸的替代(S-L917和K-M985),变异率为0.69%。
从VP5来看,与其它6个毒株相比,145个氨基酸中有2个(K-E14、L-P140)发生变异,其变异率为1.38%。而这两个替代均发生在ORF A1(VP2-4-3)和ORF A2(VP5)重叠部分,其中第14位K-E的突变只发生在VP5,在VP2-4-3并没有突变;而第140位L-P的突变不仅是VP5,而且VP2-4-3也同时发生了突变(图2)。

, http://www.100md.com
图2 IBDV-H毒株及参考毒株的VP5氨基酸序列的比较
氨基酸一致用点表示,列出的是不一致的氨基酸。
Figure 2 Multiple sequence alignments of deduced amino acid sequence of VP5 encoded by small open reading frame of segment A of IBDV strains: Harbin, P2, Cu-1, UK661, STC, 52/70 and 23/82
Differences from the consensus sequence are shown and dots indicate the same residues as consensus.
2 IBDV-H毒株与其它毒株的亲缘关系分析
, http://www.100md.com
将IBDV-H毒株的氨基酸序列与已报道的IBDV血清Ⅰ型、变异株和Ⅱ型毒株的相应序列进行了同源分析,结果是:从整个ORF A1(VP2-4-3)来看,H毒株和P2、Cu-1同源性最高,其亲缘关系最近(图3a),而从每个单一蛋白VP2、VP4和VP3,包括ORF A2(VP5)同源比较所得出的系统树,其结果均是一致的(图3b、c、d、e)。

图3 IBDV各毒株的亲缘关系图谱
Figure 3 Phylogenetic tree of strains of IBDV
讨论
通过反转录得到了IBDV-H毒株A节段的全长cDNA片段,用PCR分5'端(1659bp)和3'端(1444 bp)上下两段扩增得到PCR产物,分别克隆到pGEMB®-T载体上,测定了其核苷酸顺序。在长为3 101个bp中含有两个阅读框ORF A1和ORF A2,分别编码1 012个氨基酸的前体蛋白(VP2-4-3)和145个氨基酸的VP5,ORF A1和ORF A2有部分的重叠。将核苷酸序列及推测出的氨基酸序列与已报道的IBDV血清Ⅰ型、变异株和Ⅱ型毒株的相应序列进行了比较,结果表明,中国分离的强毒株H毒株,其决定毒力和抗原性的序列均有变异,但它既不同于欧洲[3,10]、日本和以色列的超强毒[4,9],也不同于美国和中国广州的变异株[11,12]。其亲缘关系是和欧洲较早时期分离的毒株最近,但在可变区,特别是在两个亲水区呈现了明显的突变。
, 百拇医药
据有关文献报道,超强毒IBDV(vvIBDV)如欧洲的UK661、日本的OKYM等,222位的A、256位的Ⅰ和294位的Ⅰ是保守的[3,12-14],国内陈红英等人[15](1998)对IBDV AH-2、HD96、B2/28、JS-18和D11-2株的VP2高变区的序列分析显示,这三个位置的氨基酸也是保守的,然而H-毒株却与此不同。
从世界各地分离的毒株的序列比较可以看出,不同毒株之间序列的变异主要发生在VP2区,尤其是206~350位氨基酸残基之间变异最大[11,16,17]。VP2是主要的保护性抗原[10]。VP3和VP4相对保守,不仅在同一血清型之间,甚至在两个血清型之间也很相似[5]。本研究发现的H毒株12个特有的氨基酸中,8个在VP2,其中有5个在可变区,2个位于第一个亲水区内(212-224),1个位于第二个亲水区内(314-324)。第一个亲水区与构象依赖性抗原表位的构象稳定有关,第二个亲水区为中和抗体的结合位点[13,18]。因此,在这两个区域如有变异很可能诱导抗原性的变化。经典Ⅰ型毒株中这两个亲水区的氨基酸序列是完全保守的,与经典Ⅰ型毒株相比,欧洲的UK661、日本的OKYM、中国的GZ29112株包括变异株GLS也只有1个氨基酸的替代,而H毒株有2个氨基酸的突变。在亲水区内氨基酸的替代也许与其毒力的增强有关。同时这些氨基酸的变化可能引起了抗原性的改变,造成具有较高水平母源抗体鸡的发病。大多数变异株在两个亲水区的确都发生了氨基酸的变化,但Miss株是个例外,在两个亲水区,Miss株与STC、52/70和PBG98等经典株具有完全相同的氨基酸序列[11]。因此可以说在其它位置的氨基酸的变化通过诱导VP2构象的改变进而导致抗原的变异[19]。
, 百拇医药
第二个亲水区之后的七肽区多年来被认为是IBDV毒力强弱的一个重要标志。强毒株具有相当保守的富含丝氨酸(S)的SWSASGS序列,弱毒株具有较少的丝氨酸,不具致病力的血清Ⅱ型OH和23/82在七肽区内有3个氨基酸发生变异[1,8,18,20,21]。但从我们的分析结果看,大部分经典株和国外的超强毒七肽区是保守的,血清Ⅰ型002-73株也有3个氨基酸发生变异,而中国的强毒H株使七肽区的SWSASGS序列变成了SWSARGS,这就意味着富含丝氨酸的七肽区内丝氨酸的突变可能不是毒力降低的唯一决定因素。
虽然中国强毒Harbin株的起源还不能定论,但从序列同源性比较的系统树显示是与欧洲的较早时期分离的毒株最接近。也许是恢复突变,也不能排除基因重组,要想进一步了解出现新的高致病性的IBDV的机制,还需做更详细的分子流行病学的研究。
基金项目:国家自然科学基金重大项目(39893290);北京市自然科学基金(5952004);国家教委博士点基金。
, http://www.100md.com
作者简介:胡子信(1963-),男(蒙古),副教授,博士研究生,主要从事动物病毒,免疫和分子生物学方面的教学和研究。
参考文献
1 Azad A A, Barret S A, Fahey K J. The characterization and molecular cloning of the double-stranded RNA genome of an Australian infectious bursal disease virus[J]. Virology, 1985, 143:35-44.
2 胡子信,张曼夫.传染性法氏囊病病毒A节段编码序列cDNA基因的克隆和序列分析[J].中国预防兽医学报,1999,3
3 Brown M D, Skinner M A. Coding sequences of both genome segments of a European ‘very virulent’isolate [J]. Virus Res, 1996, 40:1-15.
, 百拇医药
4 Yamaguchi T, Ogawa M, Inoshima Y, et al. Sequence and phylogenetic analysis of highly virulent infectious bursal disease virus[J]. Arch Virol, 1997, 142:154-172.
5 Bayliss C D, Spies U, Shaw K, et al. A comparison of the sequences of segment A of four infectious bursal disease virus strains and identification of a variable region in VP2 [J]. J Gen Virol, 1990, 71:1303-1312.
6 Dybing J K. Sequence analysis of IBDV of serotype 2 and expression of the VP5 cDNA [R]. M. Sc, University of Prince Edward Island, Charlottesville 1992.
, 百拇医药
7 Kibenge F S, Jackwood D J, Mercado C C. Nucleotide sequence analysis of genome segment A of infectious bursal disease virus[J]. J Gen Virol, 1990, 71: 569-577.
8 Heppell J, Berthiaume L, Corbin F, et al. Comparison of amino acid sequences deduced from a cDNA fragment obtained from infectious pancreatic necrosis virus (IPNV) strains of different serotypes[J]. Virology, 1993, 195:840-84.
9 Pitcovski J, Goldberg D, Levi B Z, et al. Coding region of segment A sequence of a very virulent isolate of IBDV-Comparison with isolates from different countries and virulence[J]. Avian Dis, 1998, 42:497-506.
, 百拇医药
10 Eterradossi N, Rivallan G, Toquin D, et al. Limited antigenic variation among recent infectious bursal disease virus isolates from France[J]. Arch Virol, 1997, 142: 2079-2087.
11 Dormitorio T V, Giambrone J J, Duck L W. Sequence conparison of the variable VP2 region of eight infectious bursal disease virus isolates[J]. Avian Dis, 1997, 41: 36-44.
12 Cao Y C, Yeung W S, Law M, et al. Molecular characterization of seven Chinese isolates of infectious bursal disease virus: classical, very virulent, and variant strains[J]. Avian Dis, 1998, 42: 340-351.
, 百拇医药
13 Jackwood D J, Sommer S E. Genetic heterogeneity in the VP2 gene of infectious bursal disease viruses detected in commercially reared chickens[J]. Avian Dis, 1998, 42: 321-339.
14 Yamaguchi T, Ogawa M, Inoshima Y, et al. Identification of sequence changes responsible for the attenuation of highly virulent infectious bursal disease virus[J]. Virology, 1996, 223: 219-223.
15 Chen H Y, Zhou Q, Zhang M. F. Sequence analysis of the VP2 hypervariable region of nine infectious bursal disease virus isolates from Mainland China[J]. Avian Dis 1998, 42: 762-769.
, 百拇医药
16 Brown M D, Green P, Skinner M A. VP2 sequences of recent European ‘very virulent’isolates of infectious bursal disease virus are closely related to each other but are distinct from those of ‘classical’strains[J]. J Gen Virol 1994, 75: 675-680.
17 Lana D P, Beisel C E, Silva R F. Genetic mechanisms of antigenic variation in infectious bursal disease virus: Analysis of a naturally occurring variant virus[J]. Virus Genes, 1992, 6(3): 247-259.
, 百拇医药
18 Heine H-G, Haritou M, Failla P. Sequence analysis and expression of the host-protective immunogen VP2 of a variant strain of infectious bursal disease virus, which can circumvent vaccination with standard type 1 strains[J]. J Gen Virol, 1991, 72: 1835-1843.
19 Van Den Berg TP, Gonze M, Morales D, et al. Acute infectious bursal disease in poultry:immunological and molecular basis of antigenicity of a highly virulent strain[J]. Avian Pathol, 1996, 25: 751-768.
, 百拇医药
20 Jackwood D J, Jackwood R J, Sommer SE. Identification and comparison of point mutations associated in classic and variant infectious bursal disease viruses[J]. Virus Res, 1997, 49: 131-137.
21 Vakharia V N, He J K, Ahamed B. Molecular basis of antigenic variation in infectious bursal disease virus[J]. Virus Res, 1994, 31: 265-273.
收稿日期:1998-12-09;修回日期:1999-03-26, 百拇医药