引起产科新生儿腹泻暴发的轮状病毒株VP4 VP7编码基因与其毒力关系的探讨
作者:肖玮 钱渊 张又
单位:首都儿科研究所北京市感染与免疫中心实验室,北京基因诊断基础性研究实验室,北京 100020
关键词:轮状病毒;毒力;基因型;血清型;VP4;VP7
病毒学报990404 摘要:克隆并测定了引起产科新生儿腹泻暴发的P2[6]、G4型轮状病毒(BN株)VP4的VP8片段和VP7编码基因的核苷酸序列,并据此推导出其氨基酸序列。与相应标准株和地方株(包括有毒株和无毒株)比较的结果表明,所测VP8序列与相同型别(P2[6])的标准株M37(无毒株)和ST3(无毒株)、地方株N16(无毒株)和VE7156(有毒株)之间的同源性为92.8%~98.6%,胰酶作用位点各毒株间相同;位于aa49、aa50、aa52、aa53、aa78处的氨基酸在有毒株与无毒株间(包括BN株)不同,但分别保守。VP7基因与同型(G4)标准株ST3(A亚型/无毒株)和VA70(B亚型/有毒株)、意大利地方株PV5249(A亚型/有毒株)和北京地方株CR117(有毒株)、同型猪有毒株Gott之间的同源性为91.4%~97.8%,其中与A亚型的同源性为95.5%~96.3%,而与B亚型的同源性为91.4%,提示VP7为G4A亚型,位于aa38、aa78、aa145、aa238位点的氨基酸在有毒株与无毒株之间不同,但分别保守。分析了虽为P2[6]型却反常地引起新生儿腹泻暴发毒株(BN)的VP8与VP7基因的变异情况,并对轮状病毒毒力与VP4、VP7基因变异的相互关系进行了讨论,为慎重确定轮状病毒疫苗候选毒株提供理论依据。
, http://www.100md.com
中图分类号:R373.9;Q78 文献标识码:A 文章编号:1000-8721(1999)04-0314-09
A PRELIMINARY STUDY ON RELATIONSHIP BETWEEN THE VIRAL VIRULENCE AND VIRAL GENES ENCODING VP4 VP7 PROTEINS OF P2 [6] G4 ROTAVIRUSE THAT CAUSED A NEONATAL DIARRHEA OUTBREAK
XIAO Wei, QIAN Yuan, ZHANG You
(Beijing Municipal Laboratory of Infection and Immunity, Beijing Gene Diagnosis Center of Capital Institute of Pediatrics, Beijing 100020, China)
, 百拇医药
Abstract: The genes encoding VP8 (specific fragment from VP4 gene) and VP7 of BN rotavirus strain (serotype and genotype P2 [6] G4), which caused a neonatal diarrhea outbreak in Beijing, were cloned and sequenced, and their deduced amino acid sequences were analyzed. Amino acid homology of VP8 of BN strain (P2 [6] ) to corresponding type P2 [6] reference strains M37 (asymptomatic)、ST3 (asymptomatic) and field strains N16 (asymptomatic)、VE7156(symptomatic) ranged from 92.8-98.6%, all of these P2 [6] rotavirus strains (asymptomatic and symptomatic) had identical sequences in the trypsin cleavage sites. Amino acid homology of VP7 (G4) to corresponding serotype G4 reference strains ST3 (G4A/asymptomatic)、VA70 (G4B/symptomatic) and field strains PV5249 (G4A/symptomatic)、CR117 (symptomatic) and Gottfried strain of Porcine (G4/symptomatic) ranged from 91.4-97.8%, in which the homology to G4 A subtype rotaviruses (95.5-96.3%) was higher than that to G4 B subtype rotaviruses (91.4%), suggesting VP7 of BN strain belongs to A subtype for G4 serotype. The P2 [6] rotavirus used to be believed to associate with asymptomatic infections, but BN strain rotavirus belonging to P2 [6] type caused an diarrhea outbreak in neonates. The variation of the genes encoding VP8、VP7 and the correlation between variation and viral virulence were discussed.
, http://www.100md.com
Key words: Rotavirus; Virulence; Genotype; Serotype; VP4; VP7
A组轮状病毒是婴幼儿重症腹泻的最重要病原,其两个主要外壳结构蛋白VP4与VP7分别形成独立的分型系统P血清型和G血清型。VP4为蛋白酶(proteinase)敏感蛋白,因此称为P血清型,同时,VP4又依基因序列不同而分型,称为P[]基因型。而VP7为糖蛋白(glycoprotein),因此称为G血清型。迄今,已知有13个P血清型、20个P[]基因型和14个G血清型。其中属P2血清型的毒株又为P[6]基因型,通常从无症状感染的新生儿的粪便中分离[1],一般认为是自然减毒株或无毒株,其VP7既可以是G1,也可以是G2、G3或G4,代表株为M37。同时,VP4和VP7还分别诱导产生中和抗体,从而成为疫苗研制的重要成分[2],因此有人建议将P2[6]型无毒株作为自然减毒的最佳的多价疫苗候选毒株[2,3]。
, 百拇医药
然而,最近我们发现一起轮状病毒(BN株)引起的北京一医院产科母婴同室新生儿腹泻暴发,经斑点杂交及巢式PCR证实,其VP4和VP7为P2G4型[4],并初步判定其VP4为P[6]基因型。这显然与上述相饽。Hoshino和Kirkwood报道,VP4、VP7各自独立地在轮状病毒的毒力方面起着重要作用[5,6]。VP4在胰酶作用下可裂解为VP8和VP5两个片段,其中VP8含有与毒力有关的胰酶作用区[7],并且抗原分析确定,VP8片段包含了VP4主要抗原位点,且负责VP4血清型特异中和反应[8,9]。为了解通常认为无毒而此次引起新生儿腹泻暴发的P2型毒株(BN)的主要结构蛋白VP4和VP7的基因,更进一步地认识轮状病毒的毒力相关基因,以期对我国轮状病毒的流行和毒力变异作深入的了解,为轮状病毒感染的防治提供帮助,我们克隆并测定了此有毒新生儿株(BN株)的VP8和VP7编码基因,并推导出氨基酸序列,分别与已发表的有毒的和无毒的相同型别的标准株、地方株序列进行比较。
, http://www.100md.com
材料与方法
1 病毒 BN毒株是1997年11月在北京某医院产科母婴同室的新生儿腹泻暴发患者的粪便中经用10%聚丙烯酰胺凝胶电泳检测到的,所有检测的标本均呈现完全一致的A组轮状病毒核酸图形;而同期采集的无腹泻症状新生儿的粪便标本中未检测到轮状病毒核酸;同期收集的门诊腹泻患儿粪便中的轮状病毒核酸图型与新生儿标本中的不一致,呈现多样的核酸图形。进一步用斑点杂交和巢式PCR鉴定证明,所有检测到的BN株均是:VP4为P2型,VP7为G4型。而门诊病人标本中的轮状病毒其VP4均为P1型,VP7为1型或2型[4]。用于比较的P2[6]型毒株有从无症状新生儿粪便中分离到的无毒株:标准株M37、ST3,北京地方株N16[10];引起新生儿腹泻的有毒株:美国地方株VE7156[11]。G4型毒株:标准株ST3(4A亚型/无毒株)[12]、VA70(4B亚型/有毒株)[12],北京地方株CR117(有毒株)[13],意大利地方株PV5249(4A亚型/有毒株)[12],引起猪腹泻的有毒株Gottfried(Gott)[12]。
, http://www.100md.com
2 病毒RNA的提取 400μl 10%粪便经1%SDS 56℃作用30分钟,加等体积的酚-氯仿抽提后用羟基磷灰石(HA)纯化病毒dsRNA[10]。
3 逆转录聚合酶链反应(RT-PCR) 模板为上述纯化的病毒dsRNA。参照Gentsoh[14]设计引物P1(con3)(nt1-17):5′-GGCTATAAAATGGCTTC-3′,P2(con2)(nt887-868):5′-ATTTCGGACCATTTATAACC-3′用于扩增VP8片段;P3(END9)(-):5′-GGTCACATCATACAATTCTAATCTAAG-3′(1 062~1 036)和P4(BEG9)(+):5′-GGCTTTAAAAGAGAGAATTTCCGTCTGG-3′(1~28)用于扩增VP7全基因[13]。RT-PCR参照文献[15]进行。
4 克隆 用Millipore柱纯化PCR产物,末端用T4DNA多聚酶补齐后插入质粒pTZ18R的Smal I处,具体方法见文献[10]。
, http://www.100md.com
5 测序 用T7 DNA测序试剂盒,按说明书用末端终止法分别对VP4和VP7两个基因的两个阳性克隆同时进行测序。所用引物为pTZ18R上的-40、Reverse及根据VP4基因上的相对保守区设计的引物P5(nt206~223):5′-GATGGTCCTTATCAAC-3′、P6(nt658~639):5′-CATTACACTTAGATTCTTG-3′和根据VP7基因设计的引物P7(nt552~531):5′-CCATTGGATTACACAACCATTC-3′、P8(nt819~801):5′-GCTACGTTTTCTCTTGGTCC-3′。用DNASIS和PROSIS软件进行核苷酸及氨基酸序列分析。
6 主要工具酶和试剂 实验用各种限制性内切酶、Taq DNA聚合酶、T4DNA多聚酶、T4DNA连接酶、CIP等均购自GIBCO BRL公司。引物由上海生工生物工程有限公司合成。T7DNA测序试剂盒购于上海 Promega公司。[α-32P]dATP购自福瑞公司。
, 百拇医药
结果
按T7 DNA测序试剂盒说明,用末端终止法分别对上述阳性克隆的双链cDNA测序。同时推导出氨基酸序列,分别与相应的标准株、地方株及Gott株的氨基酸序列进行比较,结果见图1、图2和表1。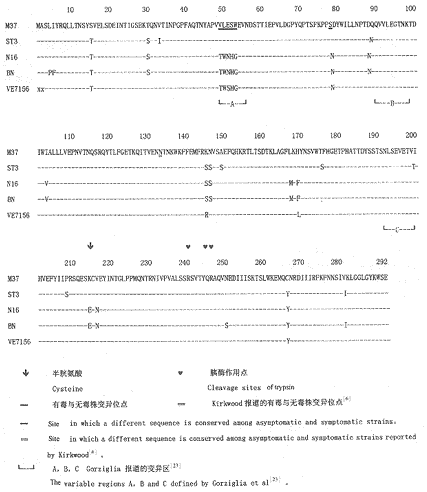
图1 A组轮状病毒P2[6]型地方株BN与同型有毒株与无毒标准株及地方株VP4血清型特异片段VP8的氨基酸序列比较
Figrure 1 Comparision of the amino acid sequences of VP8 (specific fragment from VP4 gene) among P2 [6] rotavirus BN field strain and previously published P2 strains recovered from asymptomatic and symptomatic infections
, http://www.100md.com
图2 A组轮状病毒G4地方株BN与同型(包括A、B亚型)有毒与无毒标准株和地方株VP7氨基酸序列比较
Figure 2 Comparison of the amino acid sequences of VP7 among G4 rotavirus BN field strain and previously published G4 (including A and B subtypes) strains recovered from asymptomatic and symptomatic infections VR1-VR9 variable regions of amino acid sequence was defined by Clark[18]. Potential N-glycosylation site is shown in the box.
表1 A组轮状病毒P2[6]G4型地方株BN与同型有毒、无毒的标准株和地方株VP8、VP7氨基酸序列同源性(%)
, http://www.100md.com
Table 1 Percentage of amino acid identity of VP8, VP7 among P2 [6] G4 field strain BN and previously published P2 [6] G4 strains recovered from asymptomatic or symptomatic infections. RV strain
M37
ST3
N16
VE7156
ST3
VA70
PV5249
, 百拇医药
CR117
Gott
Genotype
P[6]
P[6]
P[6]
P[6]
Serotype
P2
P2
P2
P2
G4A
, 百拇医药
G4B
G4A
G4
G4
Virulence
-
-
-
+
-
+
+
+
+
, 百拇医药
VP8
92.8
93.5
98.6
95.2
VP7
95.5
91.4
96.3
97.8
93.9
表中所列的参考株序列VP4的GenBank Accession Number为:M37/L20877,ST3/L33895,VE7157/L25265;VP7的GenBank Accession Number为:ST3/M37364,VA70/M37369,PV5249/M86490,Gott/X06386。
, 百拇医药
Available GenBank Accession Numbers for the sequences in this Table are: for VP4:M37(L20877),ST3(L33895),VE7157(L25265);for VP7:ST3(M37364),VA70(M37369),PV5249(M86490),Gott(X06386).
BN株的VP8片段为887个碱基,起始密码子位于第10~12个碱基处,与标准株和地方株一致,为VP4读码框架的起点。表1显示BN株的VP8片段与P2[6]型标准株M37、ST3[12]和同一地区(北京)的P2[6]型无毒地方株N16[10]及有毒地方株VE7156[11]相应片段氨基酸的同源性较高,为92.8%~98.6%,与钱渊等的结论[4],与Gorziglia关于两个轮状病毒之间VP4氨基酸同源性为89%或89%以上则认为是同一基因型别的结论[16]相符,判定BN株确为P2[6]型。图1为BN株与同型4个毒株的VP8氨基酸序列的比较。结果表明,与蛋白二硫键有关的半胱氨酸和与VP4构型有关的脯氨酸,除ST3于aa208处由S代替P(P→S)外,在5个毒株中完全一致;在胰酶作用区5个毒株一致:在aa240、aa246处均各有一精氨酸构成胰酶作用位点,以及aa245处的潜在胰酶作用位点消失(K→Q)和aa242处V替代I(I→V),aa243处T替代Q(Q→T);但是在胰酶作用区侧翼第aa250处,BN发生N→S变异,因而与其它毒株不同。另外,在有毒株与无毒株之间位于aa49、aa50、aa52、aa53、aa78处的氨基酸不同,但分别保守;在aa49、aa50、aa52、aa53、aa78位点,有毒株(包括N16)为T、W、H、G、N,无毒株(除N16外)为V、L、S、W、S。其中BN株与有毒株(VE7156)的碱基一致。但值得一提的是,N16为无毒株,却在这5个氨基酸序列位点与有毒株一致。另外在可变区B和C,BN毒株与其它所比较的4个毒株(包括有毒株和无毒株)完全同序。
, 百拇医药
BN株的VP7基因与相应型别标准株及地方株一致,具有1 062个碱基,有两个翻译起始密码子,分别位于核苷酸序列49~51和136~138处的开放读码框架,第一个起始密码子有一弱的起始序列UXXAUGU,第二个起始密码子有一强的起始序列AXXAUGG[17],翻译终止密码子位于核苷酸序列1 027~1 029bp处;都在aa67~71位置存在N糖基化位点,以及氨基端存在两个疏水区(H1、H2)为VP7蛋白的信号肽序列[13]。
表1显示与G4A亚型标准株ST3、B亚型VA70、同一地区G4型的有毒株CR117[13]、G4 A亚型有毒地方株PV5249[12]、猪的有毒株Gott之间具有较高的同源性,为91.4%~97.8%。其中与ST3和PV5249的同源性为95.5%~96.3%,高于与VA70的同源性(91.4%)。同时,如图2所示,与ST3之间有15个氨基酸不同,而与VA70之间有28个氨基酸不同,提示BN株为A亚型。对这6个同为G4型的有毒和无毒株进行比较后发现,位于aa38、aa78、aa145、aa238的氨基酸,有毒株与无毒株不相同,但分别保守,在BN株和有毒株、Gott株分别为苏氨酸(T)、丝氨酸(S)、丙氨酸(A)、天冬氨酸(D),而在无毒株ST3则为丙氨酸(A)、缬氨酸(V)、缬氨酸(V)、天冬酰胺(N)。另外,无毒株ST3在aa238~240处的NIT构成一个潜在的糖基化位点,而在BN株和有毒株则由于D、V代替了aa238~239处的N、I从而使此处的糖基化位点消失。
, http://www.100md.com
讨论
轮状病毒不但是全世界范围内最重要的婴幼儿重症腹泻的病原,也是发展中国家婴幼儿腹泻死亡的主要原因之一,因此,对于轮状病毒的防治显得尤其重要。找出与轮状病毒毒力有关的基因位点,对于制定防治策略是至关重要的。一般认为新生儿的轮状病毒感染是无症状的[2]。而本文所提到的BN株却引起新生儿腹泻的暴发,这一点通过对同一时期门诊的有腹泻或无腹泻病人粪便进行聚丙烯酰胺凝胶电泳图形分析及其VP4及VP7的基因分型已得到证实[4]。这是否与其基因的变异有关?是哪一个基因片段与毒力的改变有关?这些问题引起了我们极大的兴趣。轮状病毒的主要外壳结构蛋白VP4和VP7在诱导产生中和抗体方面起了主要作用,因此本研究工作首先对BN株的VP4、VP7的氨基酸序列进行了初步的分析。已知VP7的氨基酸序列存在9个高变区(VR1~VR9),这些高变区在相同血清型轮状病毒间保守,而在不同型间高变[18]。认为这些高变区涉及中和抗原位点的形成。 Green[19]等报道,如果两个毒株在VP7氨基酸序列的同源性大于85%,则可判定为同一血清型。Gerna在用单克隆抗体对G4型的毒株进行抗原分析时发现,G4型毒株还可分为两个亚型[20]。Green等进一步对不同亚型G4毒株的VP7基因进行了氨基酸序列的分析,找出了亚型分型的分子基础[12]。我们在将BN株与同型(G4型)标准株A亚型ST3、B亚型VA70的VP7氨基酸序列作比较时发现,其同源性分别为95.5%和91.4%,与A亚型的同源性高于与B亚型(见表2),因此判定BN为G4型,并提示属A亚型的可能性大于B亚型。
, http://www.100md.com
我们进一步分析了BN株VP7基因的氨基酸序列,BN与ST3有15个氨基酸不同(见图2)。Green在文献[12]所报道的同为A亚型的意大利地方株PV5249与ST3的氨基酸序列比较表明,有7个氨基酸不同,其中5个氨基酸变异与BN株的变异一致。BN与VA70有28个氨基酸不同,其中20个(71.4%)位于高可变区,在这20个氨基酸中有8个位于VR2-VR3区域,有9个位于VR5、VR7、VR8。而Green也报道亚型之间变异的氨基酸座落于VR2、VR3、VR5、VR7、VR8[12]。Dyall-Smith报道,VR5、VR7、VR8分别构成VP7的3个抗原位点A、B、C[21]。更为有意义的是,Green在对VP7 G4血清亚型分析的报道中指出,A、B亚型之间有15个氨基酸不同(图2中粗线标示),但在各亚型内却分别保守[12]。而图2显示,BN的这15个氨基酸位点均与ST3完全相同,也就是说BN株在这15个氨基酸位点的变化与Green所报道的A亚型的变化完全一致。所以这些序列的分析结果也支持BN为A亚型。当然A、B亚型的最终判断还依赖于亚型特异的中和性单克隆抗体。
, 百拇医药
将BN株的VP7氨基酸序列与同为G4的有毒和无毒的5个轮状病毒毒株的VP7序列的比较中发现,有4个氨基酸位点(aa38A→T,aa78V→S,aa146V→A,aa238N→D)(无毒株→有毒株)在有毒株和无毒株之间不同,但在有毒株或无毒株内却各自保守(见图2中黑三角标示)。同时与猪的有毒株Gott进行比较,结果BN株也与Gott的这4个氨基酸(除Gott的aa38为Y外)的变异一致。aa238位点上N→D的变异导致有毒株在此位点上的糖基化位点的消失。Kirkwood[6]认为,此位点存在于与无毒株感染有关的抗原F区,如果此区发生变异则可以导致其抗原性的改变,进而可能改变其感染性。另外,在aa38位点正好位于H2(aa30~50)疏水区,其A→T的变异可能会影响病毒的复制并诱导T淋巴细胞的细胞毒性[6]。这些序列分析提示,VP7的这些氨基酸序列变异可能与轮状病毒的毒力有关,这与Hoshino[5]有关VP7与病毒毒力关系的报道是一致的。至于新生儿腹泻暴发与VP7 G4型及其亚型的关系,就目前所掌握的资料看,还不十分清楚。是否由于G4型较少见,所以母传抗体水平较低而导致G4型轮状病毒易引起新生儿腹泻暴发。Gerna[22]1990年曾报道,G4B型引起意大利新生儿腹泻暴发。而我们的序列分析结果提示,引起北京地区此次新生儿腹泻暴发的轮状病毒毒株为G4 A亚型。因此,所有这些有关VP7与病毒毒力的关系还有待于更多的研究资料来证实。
, 百拇医药
从以往的文献中可以看到,人们普遍认为VP4与轮状病毒的毒力有关。Gorziglia等[23]于1988年报道,在VP4全序列中,有A、B、C、D、F等5个区域(VP8有A、B、C3个区域)和91个氨基酸(VP8有50个氨基酸)位点分别在轮状病毒各有毒株内或各无毒株内保守,但在有毒株与无毒株之间是不同的,从而认为这5个区域和91个氨基酸位点与病毒的毒力有关。Kirkwood[6]1996年报道,VP4氨基酸序列中aa133、aa303和aa380位点与病毒的毒力有关,其中aa133位于VP8氨基酸序列中。我们将BN株VP4血清型特异片段VP8的氨基酸序列与同型标准株和地方株(包括有毒株和无毒株)的氨基酸序列进行比较(见图1),其结果显示,在Gorziglia提到的aa49~aa53区域和aa78位点的6个氨基酸在有毒(包括BN株)与无毒毒株间的变异并无严格的界限,因为虽然N16从无症状的新生儿分离,但在aa49~aa53其变化与有毒株一致,而且图1又显示,在B、C两区所列5个毒株包括有毒株和无毒株均完全一致而保守。另外,在上述VP8的50个氨基酸中,除aa49、aa50、aa52、aa53和aa78外,其余45个氨基酸也似与毒力无关。同时Santos(1994)[11]对VP4全基因进行分析的结果,也否定了Gorziglia(1988)的VP4全序列中存在与轮状病毒毒力有关的氨基酸位点的报道。aa133(图中以双线标示)在本文所列毒株的序列中,有毒株与无毒株也未见有明显的差异。进一步将被认为与病毒毒力有关的胰酶作用区在这5个毒株中进行比较(见图1),发现所列有毒与无毒的毒株在胰酶作用区完全一致而保守,即在aa240、aa246位点各有一个精氨酸构成胰酶作用位点,aa245处的赖氨酸由谷氨酰胺代替,从而使此处的潜在胰酶作用位点消失。据文献报道,这些变化均属于P2[6]型无毒株的变化[2]。作为北京地区P2[6]型无毒株的N16,却和同一地区引起新生儿腹泻暴发流行的有毒株BN株及其它地区的有毒株的氨基酸变化一致。因此,这些毒株在胰酶区氨基酸序列的相同性只能说明这5个毒株均为同一个型别,即P2[6]型,但不能解释与毒力的关系。
, 百拇医药
综上所述,尽管有一些报道表明,VP4或VP8序列中的某个区域或某个氨基酸位点与轮状病毒的毒力有关[23],但又有一些报道不支持这些观点[11]。从我们对BN株的序列分析结果来看,氨基酸序列在同一型别(P2[6])的有毒株与无毒株之间没有明显差异,这可能与所用于比较的毒株例数较少,毒株来源有限有关。而VP4或VP8在轮状病毒的毒力方面是否确切有作用,还有待更多资料和更深入的研究。但就目前对BN株的研究表明,至少可以确定P2[6]型的轮状病毒既可以是无毒株而引起新生儿无症状感染,也可以是致病毒株引起新生儿腹泻暴发。因此在选用这类毒株作疫苗时应格外慎重。Santos[11]认为,无毒的P2[6]型是一种新生儿的自然减毒株,这一减毒过程一方面涉及病毒本身,另一方面也涉及宿主各方面因素,包括抗病毒抗体、胰酶抑制因子、母乳中的某些细胞成分、非免疫球蛋白的作用、单一的肠道菌群等,以及新生儿未发育成熟的消化道。但同一毒株在一个短时期内引起众多新生儿腹泻的暴发,单从宿主因素不好解释,而毒株某一基因片段发生变异导致毒力改变的可能性更大一些。最近有文献报道,某些非结构蛋白与轮状病毒毒力有关,我们对这一毒株其它基因的研究还在进行中。
, 百拇医药
基金项目:卫生部科研基金(1385.312);北京市自然科学基金(91-G-117)。
作者简介:肖玮(1961-),女,湖南,助理研究员,学士,研究方向为医学病毒学,现在首都儿科研究所病毒室。
参考文献
1 Gorziglia M, Nishikawa K, Hoshino Y, et al. Similarity of the outer capsid protein VP4 of the Gottfried strain of porcine rotavirus to that of asymptomatic human rotavirus strains [J] . J Virol, 1990, 64: 414-418
2 Kapikian A Z, Chanock R M. Rotaviruses [M] . In: Fields Virology. Fields B N, et al. ed. 3rd ed, New York: Lippincottraven Publishers, Philadelphia, 1996, 1669-1671
, 百拇医药
3 袁丽娟,钱渊.A组轮状病毒分子生物学研究进展[J].国外医学病毒学分册,1992,6:193-195
4 钱渊,张又,肖玮,等.P2、G4型轮状病毒引起产科新生儿腹泻暴发流行[J].中华儿科杂志,1999,37:283-286.
5 Hoshino Y, Siaf L J, Kang S Y, et al. Identification of Group A rotavirus genes associated with virulence of a porcine rotavirus and host range restriction of a human rotavirus in the gnotobiotic piglet model [J] . Virology, 1995, 209: 274-280
6 Kirkwood C D, Coulson B S, Bishop R F. G3P2 rotaviruses causing diarrhoeal disease in neonates differ in VP4, VP7 and NSP4 sequence from G3P2 strains causing asymptomatic neonatal infection [J] . Arch Virol, 1996, 141: 1661-1676
, 百拇医药
7 Gorziglia M, Hoshino Y, Buckler-white A, et al. Conservation of amino acid sequence of VP8 and cleavage region of 84-kDa outer capsid protein among rotaviruses recovered from asymptomatic neonatal infection [J] . Proc Natl Acad Sci USA, 1986, 83: 7039-7043
8 Estes M K, Cohen J. Rotavirus gene structure and function[J]. Microbiol Rev, 1989, 53: 410-449
9 Larralde G, Li B, Kapikian A Z, et al. Serotype-specific epitope (s) present on the VP8 subunit of rotavirus VP4 [J] . J Virol, 1991, 65: 3213-3218
, 百拇医药
10 肖玮,钱渊.人A组轮状病毒北京地方株VP4血清型特异片段编码基因的序列分析[J].微生物学报,1998,38(3):197-203
11 Santos N, Gouvea V, de Carmo Timenetsky M, et al. Comparative analysis of VP8* sequences from rotaviruses possessing M37-like VP4 from children with and without diarrhea [J] . J Gen Virol, 1994, 75: 1775-1780
12 Green K Y, Sarasini A, Qian Y, et al. Genetic variation in rotavirus serotype 4 subtypes [J] . Virology, 1992, 188: 362-368
, 百拇医药
13 李国华,钱渊,熊朝晖,等.北京地区G1-G4型人轮状病毒地方株VP7编码基因的序列分析[J].病毒学报,1998,14(2):126-132
14 Gentsch J R, Glass R I, Woods P, et al. Identification of group A rotavirus gene 4 type by polymerase chain reaction [J]. J Chin Microbiol, 1992, 30: 1365-1373
15 熊朝晖,钱渊,袁丽娟,等.应用多聚酶链扩增技术鉴别混合感染轮状病毒的血清型别[J].中华微生物学和免疫学杂志,1995,15:105-108
16 Gorziglia M, Larralde G, Kapikian A Z, et al. Antigenic relationships among human rotaviruses as determined by outer capsid VP4 [J] . Proc Natl Acd Sci USA, 1990, 87: 7155-7159
, 百拇医药
17 Kozak M. Point mutations close to the AUG initiation codon affect the efficiency of translation of rat preproinsulin in vivo[J] . Nature, 1984, 308: 241-246
18 Green K Y, Hosshio Y, Ikegami N. Sequence analysis of the gene encoding the serotype-specific glycoprotein (VP7) of two new human rotavirus serotypes [J] . Virology, 1989, 168: 429-433
19 Green K Y, Sears J F, Taniguchi K, et al. Prediction of human rotavirus serotype by nucleotide sequence analysis of the VP7 protein gene [J] . J Virol, 1988, 62: 1819-1823
, 百拇医药
20 Gerna G, Sarasini A, Di Matteo A, et al. Identification of two subtypes of serotype 4 human rotavirus by using VP7-specific neutralizing monoclonal antibodies [J] . J Clin Microbiol, 1988, 26: 1388-1392
21 Dyall-smith M L, Lazdins I, Tregear G W, et al. Location of the major antigenic sites involved in rotavirus serotype-specific neutralization [J] . Proc Natl Acad Sci USA, 1986, 83: 3465-3468
22 Gerna G, Forster J, Parea M, et al. Nosocomial outbreak of neonatal gastroenteritis caused by a new serotype 4, subtype 4B human rotavirus [J] . J Med Virol, 1990, 31: 175-182
23 Gorziglia M, Green K, Nishikawa K, et al. Sequence of the fourth gene of human rotavirus recovered from asymptomatic or symptomatic infections [J] . J Virol, 1988, 62: 2978-2984
收稿日期:1998-10-09;修回日期:1999-04-08, 百拇医药
单位:首都儿科研究所北京市感染与免疫中心实验室,北京基因诊断基础性研究实验室,北京 100020
关键词:轮状病毒;毒力;基因型;血清型;VP4;VP7
病毒学报990404 摘要:克隆并测定了引起产科新生儿腹泻暴发的P2[6]、G4型轮状病毒(BN株)VP4的VP8片段和VP7编码基因的核苷酸序列,并据此推导出其氨基酸序列。与相应标准株和地方株(包括有毒株和无毒株)比较的结果表明,所测VP8序列与相同型别(P2[6])的标准株M37(无毒株)和ST3(无毒株)、地方株N16(无毒株)和VE7156(有毒株)之间的同源性为92.8%~98.6%,胰酶作用位点各毒株间相同;位于aa49、aa50、aa52、aa53、aa78处的氨基酸在有毒株与无毒株间(包括BN株)不同,但分别保守。VP7基因与同型(G4)标准株ST3(A亚型/无毒株)和VA70(B亚型/有毒株)、意大利地方株PV5249(A亚型/有毒株)和北京地方株CR117(有毒株)、同型猪有毒株Gott之间的同源性为91.4%~97.8%,其中与A亚型的同源性为95.5%~96.3%,而与B亚型的同源性为91.4%,提示VP7为G4A亚型,位于aa38、aa78、aa145、aa238位点的氨基酸在有毒株与无毒株之间不同,但分别保守。分析了虽为P2[6]型却反常地引起新生儿腹泻暴发毒株(BN)的VP8与VP7基因的变异情况,并对轮状病毒毒力与VP4、VP7基因变异的相互关系进行了讨论,为慎重确定轮状病毒疫苗候选毒株提供理论依据。
, http://www.100md.com
中图分类号:R373.9;Q78 文献标识码:A 文章编号:1000-8721(1999)04-0314-09
A PRELIMINARY STUDY ON RELATIONSHIP BETWEEN THE VIRAL VIRULENCE AND VIRAL GENES ENCODING VP4 VP7 PROTEINS OF P2 [6] G4 ROTAVIRUSE THAT CAUSED A NEONATAL DIARRHEA OUTBREAK
XIAO Wei, QIAN Yuan, ZHANG You
(Beijing Municipal Laboratory of Infection and Immunity, Beijing Gene Diagnosis Center of Capital Institute of Pediatrics, Beijing 100020, China)
, 百拇医药
Abstract: The genes encoding VP8 (specific fragment from VP4 gene) and VP7 of BN rotavirus strain (serotype and genotype P2 [6] G4), which caused a neonatal diarrhea outbreak in Beijing, were cloned and sequenced, and their deduced amino acid sequences were analyzed. Amino acid homology of VP8 of BN strain (P2 [6] ) to corresponding type P2 [6] reference strains M37 (asymptomatic)、ST3 (asymptomatic) and field strains N16 (asymptomatic)、VE7156(symptomatic) ranged from 92.8-98.6%, all of these P2 [6] rotavirus strains (asymptomatic and symptomatic) had identical sequences in the trypsin cleavage sites. Amino acid homology of VP7 (G4) to corresponding serotype G4 reference strains ST3 (G4A/asymptomatic)、VA70 (G4B/symptomatic) and field strains PV5249 (G4A/symptomatic)、CR117 (symptomatic) and Gottfried strain of Porcine (G4/symptomatic) ranged from 91.4-97.8%, in which the homology to G4 A subtype rotaviruses (95.5-96.3%) was higher than that to G4 B subtype rotaviruses (91.4%), suggesting VP7 of BN strain belongs to A subtype for G4 serotype. The P2 [6] rotavirus used to be believed to associate with asymptomatic infections, but BN strain rotavirus belonging to P2 [6] type caused an diarrhea outbreak in neonates. The variation of the genes encoding VP8、VP7 and the correlation between variation and viral virulence were discussed.
, http://www.100md.com
Key words: Rotavirus; Virulence; Genotype; Serotype; VP4; VP7
A组轮状病毒是婴幼儿重症腹泻的最重要病原,其两个主要外壳结构蛋白VP4与VP7分别形成独立的分型系统P血清型和G血清型。VP4为蛋白酶(proteinase)敏感蛋白,因此称为P血清型,同时,VP4又依基因序列不同而分型,称为P[]基因型。而VP7为糖蛋白(glycoprotein),因此称为G血清型。迄今,已知有13个P血清型、20个P[]基因型和14个G血清型。其中属P2血清型的毒株又为P[6]基因型,通常从无症状感染的新生儿的粪便中分离[1],一般认为是自然减毒株或无毒株,其VP7既可以是G1,也可以是G2、G3或G4,代表株为M37。同时,VP4和VP7还分别诱导产生中和抗体,从而成为疫苗研制的重要成分[2],因此有人建议将P2[6]型无毒株作为自然减毒的最佳的多价疫苗候选毒株[2,3]。
, 百拇医药
然而,最近我们发现一起轮状病毒(BN株)引起的北京一医院产科母婴同室新生儿腹泻暴发,经斑点杂交及巢式PCR证实,其VP4和VP7为P2G4型[4],并初步判定其VP4为P[6]基因型。这显然与上述相饽。Hoshino和Kirkwood报道,VP4、VP7各自独立地在轮状病毒的毒力方面起着重要作用[5,6]。VP4在胰酶作用下可裂解为VP8和VP5两个片段,其中VP8含有与毒力有关的胰酶作用区[7],并且抗原分析确定,VP8片段包含了VP4主要抗原位点,且负责VP4血清型特异中和反应[8,9]。为了解通常认为无毒而此次引起新生儿腹泻暴发的P2型毒株(BN)的主要结构蛋白VP4和VP7的基因,更进一步地认识轮状病毒的毒力相关基因,以期对我国轮状病毒的流行和毒力变异作深入的了解,为轮状病毒感染的防治提供帮助,我们克隆并测定了此有毒新生儿株(BN株)的VP8和VP7编码基因,并推导出氨基酸序列,分别与已发表的有毒的和无毒的相同型别的标准株、地方株序列进行比较。
, http://www.100md.com
材料与方法
1 病毒 BN毒株是1997年11月在北京某医院产科母婴同室的新生儿腹泻暴发患者的粪便中经用10%聚丙烯酰胺凝胶电泳检测到的,所有检测的标本均呈现完全一致的A组轮状病毒核酸图形;而同期采集的无腹泻症状新生儿的粪便标本中未检测到轮状病毒核酸;同期收集的门诊腹泻患儿粪便中的轮状病毒核酸图型与新生儿标本中的不一致,呈现多样的核酸图形。进一步用斑点杂交和巢式PCR鉴定证明,所有检测到的BN株均是:VP4为P2型,VP7为G4型。而门诊病人标本中的轮状病毒其VP4均为P1型,VP7为1型或2型[4]。用于比较的P2[6]型毒株有从无症状新生儿粪便中分离到的无毒株:标准株M37、ST3,北京地方株N16[10];引起新生儿腹泻的有毒株:美国地方株VE7156[11]。G4型毒株:标准株ST3(4A亚型/无毒株)[12]、VA70(4B亚型/有毒株)[12],北京地方株CR117(有毒株)[13],意大利地方株PV5249(4A亚型/有毒株)[12],引起猪腹泻的有毒株Gottfried(Gott)[12]。
, http://www.100md.com
2 病毒RNA的提取 400μl 10%粪便经1%SDS 56℃作用30分钟,加等体积的酚-氯仿抽提后用羟基磷灰石(HA)纯化病毒dsRNA[10]。
3 逆转录聚合酶链反应(RT-PCR) 模板为上述纯化的病毒dsRNA。参照Gentsoh[14]设计引物P1(con3)(nt1-17):5′-GGCTATAAAATGGCTTC-3′,P2(con2)(nt887-868):5′-ATTTCGGACCATTTATAACC-3′用于扩增VP8片段;P3(END9)(-):5′-GGTCACATCATACAATTCTAATCTAAG-3′(1 062~1 036)和P4(BEG9)(+):5′-GGCTTTAAAAGAGAGAATTTCCGTCTGG-3′(1~28)用于扩增VP7全基因[13]。RT-PCR参照文献[15]进行。
4 克隆 用Millipore柱纯化PCR产物,末端用T4DNA多聚酶补齐后插入质粒pTZ18R的Smal I处,具体方法见文献[10]。
, http://www.100md.com
5 测序 用T7 DNA测序试剂盒,按说明书用末端终止法分别对VP4和VP7两个基因的两个阳性克隆同时进行测序。所用引物为pTZ18R上的-40、Reverse及根据VP4基因上的相对保守区设计的引物P5(nt206~223):5′-GATGGTCCTTATCAAC-3′、P6(nt658~639):5′-CATTACACTTAGATTCTTG-3′和根据VP7基因设计的引物P7(nt552~531):5′-CCATTGGATTACACAACCATTC-3′、P8(nt819~801):5′-GCTACGTTTTCTCTTGGTCC-3′。用DNASIS和PROSIS软件进行核苷酸及氨基酸序列分析。
6 主要工具酶和试剂 实验用各种限制性内切酶、Taq DNA聚合酶、T4DNA多聚酶、T4DNA连接酶、CIP等均购自GIBCO BRL公司。引物由上海生工生物工程有限公司合成。T7DNA测序试剂盒购于上海 Promega公司。[α-32P]dATP购自福瑞公司。
, 百拇医药
结果
按T7 DNA测序试剂盒说明,用末端终止法分别对上述阳性克隆的双链cDNA测序。同时推导出氨基酸序列,分别与相应的标准株、地方株及Gott株的氨基酸序列进行比较,结果见图1、图2和表1。

图1 A组轮状病毒P2[6]型地方株BN与同型有毒株与无毒标准株及地方株VP4血清型特异片段VP8的氨基酸序列比较
Figrure 1 Comparision of the amino acid sequences of VP8 (specific fragment from VP4 gene) among P2 [6] rotavirus BN field strain and previously published P2 strains recovered from asymptomatic and symptomatic infections

, http://www.100md.com
图2 A组轮状病毒G4地方株BN与同型(包括A、B亚型)有毒与无毒标准株和地方株VP7氨基酸序列比较
Figure 2 Comparison of the amino acid sequences of VP7 among G4 rotavirus BN field strain and previously published G4 (including A and B subtypes) strains recovered from asymptomatic and symptomatic infections VR1-VR9 variable regions of amino acid sequence was defined by Clark[18]. Potential N-glycosylation site is shown in the box.
表1 A组轮状病毒P2[6]G4型地方株BN与同型有毒、无毒的标准株和地方株VP8、VP7氨基酸序列同源性(%)
, http://www.100md.com
Table 1 Percentage of amino acid identity of VP8, VP7 among P2 [6] G4 field strain BN and previously published P2 [6] G4 strains recovered from asymptomatic or symptomatic infections. RV strain
M37
ST3
N16
VE7156
ST3
VA70
PV5249
, 百拇医药
CR117
Gott
Genotype
P[6]
P[6]
P[6]
P[6]
Serotype
P2
P2
P2
P2
G4A
, 百拇医药
G4B
G4A
G4
G4
Virulence
-
-
-
+
-
+
+
+
+
, 百拇医药
VP8
92.8
93.5
98.6
95.2
VP7
95.5
91.4
96.3
97.8
93.9
表中所列的参考株序列VP4的GenBank Accession Number为:M37/L20877,ST3/L33895,VE7157/L25265;VP7的GenBank Accession Number为:ST3/M37364,VA70/M37369,PV5249/M86490,Gott/X06386。
, 百拇医药
Available GenBank Accession Numbers for the sequences in this Table are: for VP4:M37(L20877),ST3(L33895),VE7157(L25265);for VP7:ST3(M37364),VA70(M37369),PV5249(M86490),Gott(X06386).
BN株的VP8片段为887个碱基,起始密码子位于第10~12个碱基处,与标准株和地方株一致,为VP4读码框架的起点。表1显示BN株的VP8片段与P2[6]型标准株M37、ST3[12]和同一地区(北京)的P2[6]型无毒地方株N16[10]及有毒地方株VE7156[11]相应片段氨基酸的同源性较高,为92.8%~98.6%,与钱渊等的结论[4],与Gorziglia关于两个轮状病毒之间VP4氨基酸同源性为89%或89%以上则认为是同一基因型别的结论[16]相符,判定BN株确为P2[6]型。图1为BN株与同型4个毒株的VP8氨基酸序列的比较。结果表明,与蛋白二硫键有关的半胱氨酸和与VP4构型有关的脯氨酸,除ST3于aa208处由S代替P(P→S)外,在5个毒株中完全一致;在胰酶作用区5个毒株一致:在aa240、aa246处均各有一精氨酸构成胰酶作用位点,以及aa245处的潜在胰酶作用位点消失(K→Q)和aa242处V替代I(I→V),aa243处T替代Q(Q→T);但是在胰酶作用区侧翼第aa250处,BN发生N→S变异,因而与其它毒株不同。另外,在有毒株与无毒株之间位于aa49、aa50、aa52、aa53、aa78处的氨基酸不同,但分别保守;在aa49、aa50、aa52、aa53、aa78位点,有毒株(包括N16)为T、W、H、G、N,无毒株(除N16外)为V、L、S、W、S。其中BN株与有毒株(VE7156)的碱基一致。但值得一提的是,N16为无毒株,却在这5个氨基酸序列位点与有毒株一致。另外在可变区B和C,BN毒株与其它所比较的4个毒株(包括有毒株和无毒株)完全同序。
, 百拇医药
BN株的VP7基因与相应型别标准株及地方株一致,具有1 062个碱基,有两个翻译起始密码子,分别位于核苷酸序列49~51和136~138处的开放读码框架,第一个起始密码子有一弱的起始序列UXXAUGU,第二个起始密码子有一强的起始序列AXXAUGG[17],翻译终止密码子位于核苷酸序列1 027~1 029bp处;都在aa67~71位置存在N糖基化位点,以及氨基端存在两个疏水区(H1、H2)为VP7蛋白的信号肽序列[13]。
表1显示与G4A亚型标准株ST3、B亚型VA70、同一地区G4型的有毒株CR117[13]、G4 A亚型有毒地方株PV5249[12]、猪的有毒株Gott之间具有较高的同源性,为91.4%~97.8%。其中与ST3和PV5249的同源性为95.5%~96.3%,高于与VA70的同源性(91.4%)。同时,如图2所示,与ST3之间有15个氨基酸不同,而与VA70之间有28个氨基酸不同,提示BN株为A亚型。对这6个同为G4型的有毒和无毒株进行比较后发现,位于aa38、aa78、aa145、aa238的氨基酸,有毒株与无毒株不相同,但分别保守,在BN株和有毒株、Gott株分别为苏氨酸(T)、丝氨酸(S)、丙氨酸(A)、天冬氨酸(D),而在无毒株ST3则为丙氨酸(A)、缬氨酸(V)、缬氨酸(V)、天冬酰胺(N)。另外,无毒株ST3在aa238~240处的NIT构成一个潜在的糖基化位点,而在BN株和有毒株则由于D、V代替了aa238~239处的N、I从而使此处的糖基化位点消失。
, http://www.100md.com
讨论
轮状病毒不但是全世界范围内最重要的婴幼儿重症腹泻的病原,也是发展中国家婴幼儿腹泻死亡的主要原因之一,因此,对于轮状病毒的防治显得尤其重要。找出与轮状病毒毒力有关的基因位点,对于制定防治策略是至关重要的。一般认为新生儿的轮状病毒感染是无症状的[2]。而本文所提到的BN株却引起新生儿腹泻的暴发,这一点通过对同一时期门诊的有腹泻或无腹泻病人粪便进行聚丙烯酰胺凝胶电泳图形分析及其VP4及VP7的基因分型已得到证实[4]。这是否与其基因的变异有关?是哪一个基因片段与毒力的改变有关?这些问题引起了我们极大的兴趣。轮状病毒的主要外壳结构蛋白VP4和VP7在诱导产生中和抗体方面起了主要作用,因此本研究工作首先对BN株的VP4、VP7的氨基酸序列进行了初步的分析。已知VP7的氨基酸序列存在9个高变区(VR1~VR9),这些高变区在相同血清型轮状病毒间保守,而在不同型间高变[18]。认为这些高变区涉及中和抗原位点的形成。 Green[19]等报道,如果两个毒株在VP7氨基酸序列的同源性大于85%,则可判定为同一血清型。Gerna在用单克隆抗体对G4型的毒株进行抗原分析时发现,G4型毒株还可分为两个亚型[20]。Green等进一步对不同亚型G4毒株的VP7基因进行了氨基酸序列的分析,找出了亚型分型的分子基础[12]。我们在将BN株与同型(G4型)标准株A亚型ST3、B亚型VA70的VP7氨基酸序列作比较时发现,其同源性分别为95.5%和91.4%,与A亚型的同源性高于与B亚型(见表2),因此判定BN为G4型,并提示属A亚型的可能性大于B亚型。
, http://www.100md.com
我们进一步分析了BN株VP7基因的氨基酸序列,BN与ST3有15个氨基酸不同(见图2)。Green在文献[12]所报道的同为A亚型的意大利地方株PV5249与ST3的氨基酸序列比较表明,有7个氨基酸不同,其中5个氨基酸变异与BN株的变异一致。BN与VA70有28个氨基酸不同,其中20个(71.4%)位于高可变区,在这20个氨基酸中有8个位于VR2-VR3区域,有9个位于VR5、VR7、VR8。而Green也报道亚型之间变异的氨基酸座落于VR2、VR3、VR5、VR7、VR8[12]。Dyall-Smith报道,VR5、VR7、VR8分别构成VP7的3个抗原位点A、B、C[21]。更为有意义的是,Green在对VP7 G4血清亚型分析的报道中指出,A、B亚型之间有15个氨基酸不同(图2中粗线标示),但在各亚型内却分别保守[12]。而图2显示,BN的这15个氨基酸位点均与ST3完全相同,也就是说BN株在这15个氨基酸位点的变化与Green所报道的A亚型的变化完全一致。所以这些序列的分析结果也支持BN为A亚型。当然A、B亚型的最终判断还依赖于亚型特异的中和性单克隆抗体。
, 百拇医药
将BN株的VP7氨基酸序列与同为G4的有毒和无毒的5个轮状病毒毒株的VP7序列的比较中发现,有4个氨基酸位点(aa38A→T,aa78V→S,aa146V→A,aa238N→D)(无毒株→有毒株)在有毒株和无毒株之间不同,但在有毒株或无毒株内却各自保守(见图2中黑三角标示)。同时与猪的有毒株Gott进行比较,结果BN株也与Gott的这4个氨基酸(除Gott的aa38为Y外)的变异一致。aa238位点上N→D的变异导致有毒株在此位点上的糖基化位点的消失。Kirkwood[6]认为,此位点存在于与无毒株感染有关的抗原F区,如果此区发生变异则可以导致其抗原性的改变,进而可能改变其感染性。另外,在aa38位点正好位于H2(aa30~50)疏水区,其A→T的变异可能会影响病毒的复制并诱导T淋巴细胞的细胞毒性[6]。这些序列分析提示,VP7的这些氨基酸序列变异可能与轮状病毒的毒力有关,这与Hoshino[5]有关VP7与病毒毒力关系的报道是一致的。至于新生儿腹泻暴发与VP7 G4型及其亚型的关系,就目前所掌握的资料看,还不十分清楚。是否由于G4型较少见,所以母传抗体水平较低而导致G4型轮状病毒易引起新生儿腹泻暴发。Gerna[22]1990年曾报道,G4B型引起意大利新生儿腹泻暴发。而我们的序列分析结果提示,引起北京地区此次新生儿腹泻暴发的轮状病毒毒株为G4 A亚型。因此,所有这些有关VP7与病毒毒力的关系还有待于更多的研究资料来证实。
, 百拇医药
从以往的文献中可以看到,人们普遍认为VP4与轮状病毒的毒力有关。Gorziglia等[23]于1988年报道,在VP4全序列中,有A、B、C、D、F等5个区域(VP8有A、B、C3个区域)和91个氨基酸(VP8有50个氨基酸)位点分别在轮状病毒各有毒株内或各无毒株内保守,但在有毒株与无毒株之间是不同的,从而认为这5个区域和91个氨基酸位点与病毒的毒力有关。Kirkwood[6]1996年报道,VP4氨基酸序列中aa133、aa303和aa380位点与病毒的毒力有关,其中aa133位于VP8氨基酸序列中。我们将BN株VP4血清型特异片段VP8的氨基酸序列与同型标准株和地方株(包括有毒株和无毒株)的氨基酸序列进行比较(见图1),其结果显示,在Gorziglia提到的aa49~aa53区域和aa78位点的6个氨基酸在有毒(包括BN株)与无毒毒株间的变异并无严格的界限,因为虽然N16从无症状的新生儿分离,但在aa49~aa53其变化与有毒株一致,而且图1又显示,在B、C两区所列5个毒株包括有毒株和无毒株均完全一致而保守。另外,在上述VP8的50个氨基酸中,除aa49、aa50、aa52、aa53和aa78外,其余45个氨基酸也似与毒力无关。同时Santos(1994)[11]对VP4全基因进行分析的结果,也否定了Gorziglia(1988)的VP4全序列中存在与轮状病毒毒力有关的氨基酸位点的报道。aa133(图中以双线标示)在本文所列毒株的序列中,有毒株与无毒株也未见有明显的差异。进一步将被认为与病毒毒力有关的胰酶作用区在这5个毒株中进行比较(见图1),发现所列有毒与无毒的毒株在胰酶作用区完全一致而保守,即在aa240、aa246位点各有一个精氨酸构成胰酶作用位点,aa245处的赖氨酸由谷氨酰胺代替,从而使此处的潜在胰酶作用位点消失。据文献报道,这些变化均属于P2[6]型无毒株的变化[2]。作为北京地区P2[6]型无毒株的N16,却和同一地区引起新生儿腹泻暴发流行的有毒株BN株及其它地区的有毒株的氨基酸变化一致。因此,这些毒株在胰酶区氨基酸序列的相同性只能说明这5个毒株均为同一个型别,即P2[6]型,但不能解释与毒力的关系。
, 百拇医药
综上所述,尽管有一些报道表明,VP4或VP8序列中的某个区域或某个氨基酸位点与轮状病毒的毒力有关[23],但又有一些报道不支持这些观点[11]。从我们对BN株的序列分析结果来看,氨基酸序列在同一型别(P2[6])的有毒株与无毒株之间没有明显差异,这可能与所用于比较的毒株例数较少,毒株来源有限有关。而VP4或VP8在轮状病毒的毒力方面是否确切有作用,还有待更多资料和更深入的研究。但就目前对BN株的研究表明,至少可以确定P2[6]型的轮状病毒既可以是无毒株而引起新生儿无症状感染,也可以是致病毒株引起新生儿腹泻暴发。因此在选用这类毒株作疫苗时应格外慎重。Santos[11]认为,无毒的P2[6]型是一种新生儿的自然减毒株,这一减毒过程一方面涉及病毒本身,另一方面也涉及宿主各方面因素,包括抗病毒抗体、胰酶抑制因子、母乳中的某些细胞成分、非免疫球蛋白的作用、单一的肠道菌群等,以及新生儿未发育成熟的消化道。但同一毒株在一个短时期内引起众多新生儿腹泻的暴发,单从宿主因素不好解释,而毒株某一基因片段发生变异导致毒力改变的可能性更大一些。最近有文献报道,某些非结构蛋白与轮状病毒毒力有关,我们对这一毒株其它基因的研究还在进行中。
, 百拇医药
基金项目:卫生部科研基金(1385.312);北京市自然科学基金(91-G-117)。
作者简介:肖玮(1961-),女,湖南,助理研究员,学士,研究方向为医学病毒学,现在首都儿科研究所病毒室。
参考文献
1 Gorziglia M, Nishikawa K, Hoshino Y, et al. Similarity of the outer capsid protein VP4 of the Gottfried strain of porcine rotavirus to that of asymptomatic human rotavirus strains [J] . J Virol, 1990, 64: 414-418
2 Kapikian A Z, Chanock R M. Rotaviruses [M] . In: Fields Virology. Fields B N, et al. ed. 3rd ed, New York: Lippincottraven Publishers, Philadelphia, 1996, 1669-1671
, 百拇医药
3 袁丽娟,钱渊.A组轮状病毒分子生物学研究进展[J].国外医学病毒学分册,1992,6:193-195
4 钱渊,张又,肖玮,等.P2、G4型轮状病毒引起产科新生儿腹泻暴发流行[J].中华儿科杂志,1999,37:283-286.
5 Hoshino Y, Siaf L J, Kang S Y, et al. Identification of Group A rotavirus genes associated with virulence of a porcine rotavirus and host range restriction of a human rotavirus in the gnotobiotic piglet model [J] . Virology, 1995, 209: 274-280
6 Kirkwood C D, Coulson B S, Bishop R F. G3P2 rotaviruses causing diarrhoeal disease in neonates differ in VP4, VP7 and NSP4 sequence from G3P2 strains causing asymptomatic neonatal infection [J] . Arch Virol, 1996, 141: 1661-1676
, 百拇医药
7 Gorziglia M, Hoshino Y, Buckler-white A, et al. Conservation of amino acid sequence of VP8 and cleavage region of 84-kDa outer capsid protein among rotaviruses recovered from asymptomatic neonatal infection [J] . Proc Natl Acad Sci USA, 1986, 83: 7039-7043
8 Estes M K, Cohen J. Rotavirus gene structure and function[J]. Microbiol Rev, 1989, 53: 410-449
9 Larralde G, Li B, Kapikian A Z, et al. Serotype-specific epitope (s) present on the VP8 subunit of rotavirus VP4 [J] . J Virol, 1991, 65: 3213-3218
, 百拇医药
10 肖玮,钱渊.人A组轮状病毒北京地方株VP4血清型特异片段编码基因的序列分析[J].微生物学报,1998,38(3):197-203
11 Santos N, Gouvea V, de Carmo Timenetsky M, et al. Comparative analysis of VP8* sequences from rotaviruses possessing M37-like VP4 from children with and without diarrhea [J] . J Gen Virol, 1994, 75: 1775-1780
12 Green K Y, Sarasini A, Qian Y, et al. Genetic variation in rotavirus serotype 4 subtypes [J] . Virology, 1992, 188: 362-368
, 百拇医药
13 李国华,钱渊,熊朝晖,等.北京地区G1-G4型人轮状病毒地方株VP7编码基因的序列分析[J].病毒学报,1998,14(2):126-132
14 Gentsch J R, Glass R I, Woods P, et al. Identification of group A rotavirus gene 4 type by polymerase chain reaction [J]. J Chin Microbiol, 1992, 30: 1365-1373
15 熊朝晖,钱渊,袁丽娟,等.应用多聚酶链扩增技术鉴别混合感染轮状病毒的血清型别[J].中华微生物学和免疫学杂志,1995,15:105-108
16 Gorziglia M, Larralde G, Kapikian A Z, et al. Antigenic relationships among human rotaviruses as determined by outer capsid VP4 [J] . Proc Natl Acd Sci USA, 1990, 87: 7155-7159
, 百拇医药
17 Kozak M. Point mutations close to the AUG initiation codon affect the efficiency of translation of rat preproinsulin in vivo[J] . Nature, 1984, 308: 241-246
18 Green K Y, Hosshio Y, Ikegami N. Sequence analysis of the gene encoding the serotype-specific glycoprotein (VP7) of two new human rotavirus serotypes [J] . Virology, 1989, 168: 429-433
19 Green K Y, Sears J F, Taniguchi K, et al. Prediction of human rotavirus serotype by nucleotide sequence analysis of the VP7 protein gene [J] . J Virol, 1988, 62: 1819-1823
, 百拇医药
20 Gerna G, Sarasini A, Di Matteo A, et al. Identification of two subtypes of serotype 4 human rotavirus by using VP7-specific neutralizing monoclonal antibodies [J] . J Clin Microbiol, 1988, 26: 1388-1392
21 Dyall-smith M L, Lazdins I, Tregear G W, et al. Location of the major antigenic sites involved in rotavirus serotype-specific neutralization [J] . Proc Natl Acad Sci USA, 1986, 83: 3465-3468
22 Gerna G, Forster J, Parea M, et al. Nosocomial outbreak of neonatal gastroenteritis caused by a new serotype 4, subtype 4B human rotavirus [J] . J Med Virol, 1990, 31: 175-182
23 Gorziglia M, Green K, Nishikawa K, et al. Sequence of the fourth gene of human rotavirus recovered from asymptomatic or symptomatic infections [J] . J Virol, 1988, 62: 2978-2984
收稿日期:1998-10-09;修回日期:1999-04-08, 百拇医药