庚型肝炎病毒基因组全长cDNA的拼接及克隆朱分禄
作者:朱分禄 戚中田 任 浩 宋燕斌 邵 力△
单位:
关键词:GBV-C/HGV基因组;全长cDNA;拼接;克隆
第二军医大学学报980401 述评 庚型肝炎病毒(HGV)自发现以来,虽然在世界各地均有自血清中用RT-PCR扩增出该病毒序列的报道,但对其是否可致肝炎尚有争论。本文利用自日本患者血清获得的克隆有HGV基因片段的质粒,通过基因片段的扩增及拼接,获得了全长9 373 bp的HGV cDNA。测核苷酸序列及推导的氨基酸顺序与原来的HGV-Iw同源性为99.71%及99.76%。这一全长cDNA如能证实具有一定的功能,将是利用细胞或动物进行功能性研究的主要工具,并可为阐明HGV是否有致病性提供重要的实验依据。
中国微生物学会理事长
卫生部医学分子病毒室主任
, 百拇医药
上海医科大学教授 闻玉梅
摘要 目的: 构建GB病毒C/庚型肝炎病毒(GBV-C/HGV)基因组全长cDNA克隆。方法: 以覆盖GBV-C/HGV分离株HGV-Iw全长基因组且互相重叠的5个基因片段Iw5,Iwq2,Iwh6,Iw3和Iw3为起始材料,采用重叠延伸PCR拼接和连接酶连接的方法,构建GBV-C/HGV基因组全长cDNA克隆。结果: GBV-C/HGV基因组全长cDNA克隆的酶切图谱与预期一致;核苷酸序列分析显示,克隆的GBV-C/HGV基因组全长cDNA的核苷酸及推测的氨基酸序列与原HGV-Iw序列一致。结论: GBV-C/HGV基因组全长cDNA的克隆已经构建成功。该克隆的构建,为深入研究GBV-C/HGV的致病性及致病机制、复制及转录和翻译机制、形态及形态发生等提供了重要的实验材料。
中国图书资料分类法分类号 Q781
Splicing and cloning of the full-length genomic cDNA of GB virus C/hepatitis G virus
, http://www.100md.com
Zhu Fenlu, Qi Zhongtian, Ren Hao, Song Yanbin, Shao Li (Department of Microbiology, Department of Basic Medicine, Second Military Medical University, Shanghai, 200433)
Abstract Objective: To construct GBV-C/HGV full-length genomic cDNA clone. Methods: On the basis of 5 cDNA fragments termed in order from 5 end to 3 end of the genome:Iw5,Iwq2,Iwh6,Iw3 and Iw3 of a GBV-C/HGV isolate HGV-Iw,the GBV-C/HGV full-length genomic cDNA clone was constructed using overlap extension PCR and ligation methods. Results: By digesting with the predicted endonucleases,the physical map of GBV-C/HGV full-length genomic cDNA clone was identical with the expected results. The determination of nucleotide sequence showed that the nucleotide and amino acid sequences of GBV-C/HGV full-length genomic cDNA was the same as original isolate HGV-Iw. Conclusion: GBV-C/HGV full-length genomic cDNA clone has been constructed successfully,and it will play an important role in further studing on the virus pathogenicity and pathogenesis,replication,transcription and translation machanism,virus morphology and morphogenesis.
, http://www.100md.com
Key words GBV-C/HGV genome; full-length cDNA; splicing; cloning
1995年,两个研究小组几乎同时分别获得了一种新型肝炎病毒的全序列,分别命名为GB病毒C(GBV-C)和庚型肝炎病毒(HGV)[1~3]。GBV-C和HGV的核苷酸及氨基酸同源性分别为85%和95%,因此认为它们是同一种病毒的两个不同分离株,称为GBV-C/HGV。GBV-C/HGV属黄病毒科,为单股正链RNA病毒,基因组长约9.4kb,从5至3端依次为5非编码区,结构基因区C,E1,E2,非结构基因区NS2,NS3,NS4,NS5a,NS5b及3非编码区。基因组仅含有一个开放阅读框, 编码一个约2 900个氨基酸的多聚蛋白质前体。
GBV-C/HGV基因组结构的研究进展很快,迄今已发表的GBV-C/HGV全序列有11株之多[2,3,4~8],但均为GBV-C/HGV基因片段的克隆,至今未见GBV-C/HGV基因组全长cDNA单个完整克隆的报道。GBV-C/HGV基因组全长cDNA单个完整克隆的缺乏,阻碍了对该病毒的深入研究。本研究在利用长PCR分段扩增和克隆GBV-C/HGV基因的基础上,采用重叠延伸PCR拼接和连接结合的方法,首次成功地克隆了GBV-C/HGV基因组全长cDNA。GBV-C/HGV基因组全长cDNA的克隆成功,对研究GBV-C/HGV基因组的结构和功能、GBV-C/HGV的致病性及致病机制、复制及转录和翻译机制、形态及形态发生等均具有重要的意义。
, 百拇医药
1 材料和方法
1.1 质粒和菌种 质粒Iw5,Iwq2,Iwh6,Iw3和Iw3是从一日本患者血清中扩增GBV-C/HGV基因片段并克隆至pT7 blue T-vector获得。5个质粒分别含有从5至3端覆盖GBV-C/HGV整个基因组、相邻片段互相重叠的5个GBV-C/HGV基因片段[5](图1)。克隆载体pUC18,pGEM-3Zf(+)购自华美生物工程公司,宿主菌JM109由本室保存。
图 1 GBV-C/HGV基因组结构和cDNA片段的位置
Fig 1 Structure of full length GBV-C/HGV genome and
, http://www.100md.com
the location of the cDNA fragments
nt: nucleotide
1.2 酶及试剂 所有的PCR反应均采用宝灵曼公司的ExpandTM Long Template PCR System,DNA聚合酶Ⅰ大片段(Klenow)、限制性内切酶和连接酶购自Biolab或Promega公司,普通试剂购自华美生物工程公司。
1.3 引物设计与合成 应用12条由Sangon生物工程公司合成的Y1~Y12引物进行GBV-C/HGV基因的扩增和拼接(表1)。在引物Y1和Y10的5端分别加上EcoRⅠ和NotⅠ位点及XhoⅠ和XbaⅠ位点,以便于进行克隆。
1.4 GBV-C/HGV基因片段的扩增、拼接及克隆 以质粒Iw5,Iwq2,Iwh6,Iw3和Iw3为模板,分别扩增GBV-C/HGV 5个同名基因片段。经凝胶电泳纯化,Klenow酶处理后,再进行拼接反应。经扩增和拼接可获得覆盖GBV-C/HGV整个基因组、且相邻片段互相重叠的3个基因片段。将它们分别克隆至pUC18和pGEM-3Zf(+)载体中,得到3个不同的重组质粒,再分别经适宜的酶切,回收GBV-C/HGV基因片段,与酶切后的pGEM-3Zf(+)载体连接,转化大肠杆菌,获得GBV-C/HGV全长cDNA克隆。
, 百拇医药
表 1 扩增及拼接GBV-C/ HGV基因片段的引物
Tab 1 Primers for amplifying and splicing GBV-C/HGV genes Primers
Sequences (5-3)
Location of primers in genome
Polarity
Y 1
GCG AAT TCG CGG CCG CCC CCC CCC CGG CAC TGG GTG CAA GC
1~26
+
, 百拇医药
Y 2
AGA GAG ACA TTG AAG GGC GAC
321~341
-
Y 3
GAC AGG GTT GGT AGG TCG TAA ATC C
109~133
+
Y 4
CCG TCC TTG ATG ATG GAA CTG TC
4 399~4 421
, 百拇医药
-
Y 5
TAT GGG CAT GGC ATA CCC CT
4 261~4 280
+
Y 6
GAG GGC CAC GAT GAT GTT AG
8 696~8 715
-
Y 7
TTC TGC TCC ACT TGG CTC GCT GAG T
, http://www.100md.com
8 416~8 440
+
Y 8
CTG GAT GCC ACA ACA GGC CCC T
8 881~8 902
-
Y 9
AGG GGC CTG TTG TGG CAT CCA G
8 881~8 902
+
Y10
GCT CTA GAC TCG AGA ATA AAA CCC GGC CTT TGG GCC G
, http://www.100md.com
9 353~9 375
-
Y11
TTG TTC GAT CAT ATG GGG TCC TTC TCG C
2 977~3 004
+
Y12
GAC CGG AGT CCC TTC AAG TAT CTC CTG G
7 737~7 764
-
1.5 GBV-C/HGV全长cDNA克隆的核苷酸序列测定 在初步的酶切鉴定正确之后,将GBV-C/HGV全长cDNA克隆分别从5和3端用Dye Terminator Cycles Sequencing方法进行核苷酸序列测定。利用PC/Gene软件进行序列比较分析。
, 百拇医药
2 结 果
2.1 GBV-C/HGV基因片段的扩增及拼接 以质粒Iw5,Iwq2,Iwh6,Iw3和Iw3为模板,分别对应引物Y1/Y2,Y3/Y4,Y5/Y6,Y7/Y8和Y9/Y10,扩增获得GBV-C/HGV 5个同名基因片段。推测基因片段的长度分别为:Iw5,358 bp;Iwq2,4 313 bp;Iwh6,4 455 bp;Iw3,487 bp;Iw3,509 bp。将PCR产物行1%琼脂糖凝胶电泳,所有基因片段均与预期的大小一致(图2中的第2,4,6,9,10泳带)。
GBV-C/HGV 5端基因片段的拼接以Iw5和Iwq2基因片段为模板,Y1和Y4为引物,其中Iw5基因片段的3端与Iwq2基因片段的5端重叠。经过重叠延伸PCR(SOEing and PCR)反应(PCR条件:94℃预变性2 min,然后94℃ 30s,60℃ 1 min,68℃ 6 min 35个循环,反应结束前延伸10 min),获得了长约4 437 bp的Iw5-q2基因片段(图2中的第3泳带)。依据同样的原理,以Iwq2和Iwh6基因片段为模板,Y11和Y12为引物进行拼接扩增,获得了长约4 788 bp的Iwq2-h6基因片段(图2中的第5泳带)。 3端基因片段的拼接是先以Iw3和Iw3基因片段为模板, Y7和Y10为引物, 拼接得到了长约974 bp的Iw3-3基因片段(图2中的第8泳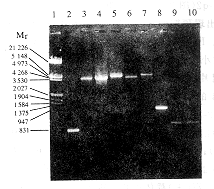
, 百拇医药
图 2 GBV-C/HGV cDNA片段的凝胶电泳分析
Fig 2 Gel electrophoresis analysis of
GBV-C/HGV cDNA fragments
1:λDNA/EcoRⅠ+ HindⅢ;2:Iw5;3:Iw5-q2;4:Iwq2;5:Iwq2-h6;
6:Iwh6;7:Iwh6-3; 8:Iw3-3;9:Iw3;10:Iw3
图 3 重组质粒 pUC-5-q2的构建
Fig 3 Construction of recombinant plasmid pUC-5-q2
, 百拇医药
带);再以Iw3-3片段与Iwh6基因片段为模板,Y5和Y10为引物进行拼接扩增,获得了长约5 129 bp的Iwh6-3基因片段(图2中的第7泳带)。
2.2 GBV-C/HGV基因片段及全长cDNA的克隆 将拼接扩增获得的GBV-C/HGV基因片段分别克隆至pUC18和pGEM-3Zf(+)载体。基因片段经凝胶电泳纯化后,Iw5-q2经EcoRⅠ+BamHⅠ酶切,回收约3 301 bp的基因片段,与同样经EcoRⅠ+BamHⅠ酶切的pUC18载体连接,获得重组质粒pUC-5-q2(图3)。该重组质粒经EcoRⅠ+BamHⅠ酶切,出现预期约3 301 bp的基因片段(图4中第2泳带)。Iwq2-h6基因片段经BamHⅠ+SalⅠ酶切,出现大小分别为308 bp, 1 181 bp和3 298 bp的3个基因片段,回收约3 298 bp的基因片段,克隆至pUC18的BamHⅠ和SalⅠ位点,得到重组质粒pUC-q2-h6。BamHⅠ+SalⅠ酶切鉴定发现该重组质粒切出预期约3 298 bp的基因片段(图4中的第4泳带)。Iwh6-3基因片段经SalⅠ和XbaⅠ酶切后回收约2 806 bp的基因片段, 与经过同种酶处理的pGEM-3Zf(+)载体连接、 转化, 转化子质粒经SalⅠ和XbaⅠ双酶切, 出现约2 806 bp的预期基因片段, 证实为重组质粒pGEM-h6-3(图4中的第6泳带)。
, 百拇医药
在完成3个基因片段的分段克隆后,将pUC-5-q2用EcoRⅠ和BamHⅠ酶切,回收约3 301 bp的基因片段;pUC-q2-h6经BamHⅠ和SalⅠ酶切,回
图 4 重组质粒 pUC-5-q2,pUC-q2-h6和
pGEM-h6-3的酶切电泳分析
Fig 4 Endonuclease digestion and gel electrophoresis
analysis of the recombinant plasmids pUC-5-q2,pUC-q2-h6 and pGEM-h6-3
1:λDNA/EcoRⅠ+ HindⅢ;2:pUC-5-q2/EcoRⅠ+ BamHⅠ;
, http://www.100md.com
3:pUC18/ EcoRⅠ+ BamHⅠ;4:pUC-q2-h6/BamHⅠ+ SalⅠ;
5:pUC18/ BamHⅠ+ SalⅠ; 6:pGEM-h6-3/SalⅠ+XbaⅠ;
7:pGEM-3Zf(+)/ SalⅠ+XbaⅠ
图 5 重组质粒 pHGVqz的构建
Fig 5 Construction of recombinant plasmid pHGVqz
收约3 298 bp的基因片段;pGEM-h6-3经SalⅠ和XbaⅠ酶切,回收约2 806 bp的基因片段。将pGEM-3Zf载体经EcoRⅠ和XbaⅠ酶切后回收,在同一连接系统中同时加入酶切后的pGEM-3Zf基因片段及3个GBV-C/HGV基因片段,经T4 DNA连接酶连接,转化大肠杆菌JM109,蓝白菌落筛选后挑取白色菌落进行酶切鉴定,得到GBV-C/HGV全长cDNA克隆pHGVqz(图5)。
, http://www.100md.com
2.3 GBV-C/HGV基因片段和全长cDNA克隆的酶切鉴定分析 根据HGV-Iw全序列的酶切位点分析,选取不同的内切酶分别对PCR扩增获得的5个GBV-C/HGV cDNA基因片段进行酶切鉴定,酶切电泳酶谱与预期结果完全一致(图略)。GBV-C/HGV基因组全长 cDNA克隆的鉴定,选用EcoRⅠ,NotⅠ,XbaⅠ,BamHⅠ,SalⅠ,PstⅠ和HindⅢ等内切酶进行鉴定。预期酶切结果:EcoRⅠ酶切,重组质粒被切为5 767 bp和6 836 bp的两个基因片段,在1%的琼脂糖凝胶电泳时,出现一条带;HindⅢ酶切,出现约为2 260 bp和10 343 bp的两条带;PstⅠ酶切出现约为2 577 bp, 3 671 bp和6 405 bp的3条带;SalⅠ酶切应出现约为2 821 bp和9 782 bp的两条带。双酶切结果:BamHⅠ+XbaⅠ,重组质粒被切为6 119 bp和6 484 bp的两个基因片段,在1%琼脂糖凝胶电泳时呈现一条带;EcoRⅠ+XbaⅠ酶切出现约为2 568 bp, 3 199 bp和6 836 bp的3条带;NotⅠ+XbaⅠ酶切出现约为9 404 bp
, http://www.100md.com
图 6 GBV-C/HGV基因组全长cDNA 克隆
的酶切图谱分析
Fig 6 Physical mapping analysis of GBV-C/HGV
full-length genomic cDNA clone digested by
the predicted endonucleases
1:λDNA/EcoRⅠ+ HindⅢ;2:pHGVqz/EcoRⅠ;3:pHGVqz/
HindⅢ; 4:pHGVqz/PstⅠ;5:pHGVqz/SalⅠ;6:pHGVqz/
BamHⅠ+ XbaⅠ; 7:pHGVqz/EcoRⅠ+XbaⅠ;8:pHGVqz/
, 百拇医药
NotⅠ+XbaⅠ; 9:pGEM-3Zf(+)/ EcoRⅠ+XbaⅠ
的GBV-C/HGV全长cDNA基因片段和约3 199 bp的pGEM-3Zf载体基因片段两条带。GBV-C/HGV基因组全长cDNA克隆的酶切图谱见图6。
2.4 GBV-C/HGV全长cDNA克隆的核苷酸序列测定 核苷酸序列测定结果显示,克隆的GBV-C/HGV的基因组全长cDNA长9 373 bp,推测编码氨基酸2 873个(序列略)。将克隆的GBV-C/HGV cDNA核苷酸序列及推测氨基酸序列与GBV-C[2],HGV[3],HGV1[3],HGVC964[4]和HGV-Iw[5]的相应序列进行了同源性比较(表2)。
表 2 GBV-C/HGV cDNA核苷酸及氨基酸序列同源性比较
, http://www.100md.com
Tab 2 Nucleotide and amino acid identity of GBV-C/HGV cDNA Compared
sequences
Accession No.
Nucleotide
identity (%)
Amino acid
identity (%)
GBV-C
U36380
86.90
, 百拇医药
96.03
HGV
U44402
93.27
98.33
HGV1
U45966
90.84
97.98
HGVC964
U75356
88.98
95.20
, http://www.100md.com
HGV-Iw
D87255
99.71
99.76
同源性比较结果证实我们已成功地获得了GBV-C/HGV的基因组全长cDNA克隆。
3 讨 论
3.1 HGV-Iw基因片段的拼接 迄今虽已报道了11株GBV-C/HGV全序列, 但均不是GBV-C/HGV基因组全长cDNA单个完整克隆。 本研究中作为实验基础的5个重组质粒克隆的GBV-C/HGV 5个同名基因片段, 从5至3端相邻片段互相重叠且覆盖长9 375 bp的HGV-Iw基因组[5], 其中Iwq2和Iwh6片段是应用长PCR方法获得的, 长度分别为4 313 bp和4 455 bp, 使我们避免了从数个几百个碱基的小基因片段起始的繁杂过程。
, 百拇医药
由重叠延伸PCR反应进行基因拼接是以PCR方法为基础, 先进行重叠延伸反应产生重组DNA分子, 再经PCR进行指数扩增的一种构建新的DNA 分子的技术方法[9]。 该方法具有无需限制性内切酶和连接酶、不管拼接位点在核苷酸序列的什么位置均可准确拼接的优点。 因该方法是以PCR反应为基础的, 所以PCR反应中由DNA聚合酶产生的错误掺入的缺陷在进行基因拼接时同样存在。 本实验中我们选用ExpendTM Long Template PCR System, 其DNA聚合酶混合物中的Pwo DNA 聚合酶具有校正阅读功能, 从而显著降低了扩增产物的错误频率。 此外, 由重叠延伸PCR拼接基因片段, 其预拼接的基因长度有一定限制, 较短的基因片段易于拼接和扩增。 本研究中拼接基因的长度约1~5 kb, 拼接结果良好。 但在初期实验直接经重叠延伸PCR拼接全长cDNA, 虽然也获得了预期全长cDNA约9.4 kb的基因片段, 但其扩增效率较差, 致使无法直接从约9.4kb的基因片段完成全长cDNA的克隆。
, 百拇医药
3.2 GBV-C/HGV全长cDNA基因的克隆策略 如上所述, 由于经直接拼接无法获取足量的用于克隆的全长cDNA基因, 因此改为采用先分段克隆GBV-C/HGV基因片段, 再克隆全长cDNA基因的方法。
在完成5端Iw5-q2和3端h6-3基因片段的拼接之后, 又设计引物, 拼接扩增了Iwq2-h6基因片段。 在设计引物Y11和Y12时, 既要考虑使Iwq2-h6基因片段5端与Iw5-q2基因片段的3端重叠基因部分包括BamHⅠ位点在内, 3端与Iwh6-3基因片段重叠部分包括SalⅠ位点;又要考虑在Iwq2-h6基因片段经BamHⅠ+SalⅠ酶切之后, 其长约3 298 bp的片段能顺利回收, 便于操作。 在分段克隆GBV-C/HGV基因片段时, 用pUC18载体克隆Iw-q2和Iw-q2-h6基因片段, 用pGEM-3Zf载体克隆Iwh6-3基因片段, 主要是从待克隆的基因片段与载体的大小相比便于进行基因操作考虑。 在完成GBV-C/HGV基因片段的分段克隆后, 将3个重组质粒分别用不同的酶切, 回收GBV-C/HGV基因片段及酶切后的载体片段, 经T4 DNA连接酶连接, 转化大肠杆菌JM109, 最后获得了GBV-C/HGV基因组全长cDNA克隆。
, http://www.100md.com
3.3 构建GBV-C/HGV基因组全长cDNA克隆的意义 对于一种新病毒, 最令人关注的是病毒的致病性。 GBV-C/HGV是否导致人类肝炎, 至今尚无明确定论。 初期研究结果显示,GBV-C/HGV与输血后肝炎及其他急、慢性肝病有关[1,3]。 近期的研究表明, GBV-C/HGV不能引起肝炎或导致的肝炎症状轻微, 对共感染的HBV, HCV所引起的肝病症状没有影响[4,10,11]。 但多数研究者同时也发现, GBV-C/HGV可经血传播, 在人群中有较高的感染率, 可引起长期持续的感染。 因此, 迫切需要对GBV-C/HGV进行广泛而深入的研究。 但GBV-C/HGV基因组全长cDNA克隆的缺乏, 阻碍了研究的进行。 这次GBV-C/HGV基因组全长cDNA的克隆成功, 为研究GBV-C/HGV的致病性及致病机制, GBV-C/HGV的复制、转录和翻译机制, GBV-C/HGV的形态及形态发生, GBV-C/HGV和HCV的比较研究等许多方面提供了重要的实验材料。
, 百拇医药 致谢: 在本研究工作中, 本校长征医院缪晓辉副教授给予了许多有益的启示和真诚的帮助;长海医院何建文博士对实验设计提出了宝贵的建议, 在此一并表示深深的谢意!
参 考 文 献
1 Simons JN, Leary TP, Dawson GJ, et al. Isolation of novel virus-like sequences associated with human hepatitis. Nat Med, 1995, 1(6):564
2 Leary TP, Muerhoff SM, Simons JN, et al. Sequence and genomic organization of GBV-C:a novel member of the Flaviviridae associated with human non-A-E hepatitis. J Med Virol, 1996, 48(1):60
, http://www.100md.com
3 Linnen J, Wages J Jr, Zhang-Keck ZY, et al. Molecular cloning and disease association of hepatitis G virus:a transfusion-transmissible agent. Science, 1996, 271(5248):505
4 周育森, 陈 微, 赵秋敏, 等. 中国人庚型肝炎病毒全基因cDNA克隆及序列测定.军事医学科学院院刊, 1996, 20(4):249
5 Shao L, Shinzawa H, Ishikawa K, et al. Sequence of hepatitis G virus genome isolated from a Japanese patient with non-A-E hepatitis:amplification and cloning by long reverse transcription-PCR. Biochem Biophys Res Commun, 1996, 228(3):785
, http://www.100md.com
6 Erker JC, Simons JN, Moerhoff AS, et al. Molecular cloning and characterization of a GB virus C isolate from a patient with non-A-E hepatitis. J Gen Virol, 1996, 77(Pt:11):2713
7 Okamoto H, Nakao H, Inoue T, et al. The entire nucleotide sequence of two GB virus C/hepatitis G virus isolates of distinct genotypes from Japan. J Gen Virol, 1997, 78(Pt:4): 737
8 Nakao H, Okamoto H, Fukuda M, et al. Mutation rate of GB virus C/hepatitis G virus over the entire genome and in subgenomic regions. Virology, 1997, 233(1):43
, http://www.100md.com
9 Horton RM, Hunt HD, Ho SN, et al. Engineering hybrid genes without the use of restriction enzymes:gene splicing by overlap extension. Gene , 1989, 77(1):61
10 Alter HJ. The cloning and clinical implications of HGV and HBGV-C. N Engl J Med, 1997, 334(23):1536
11 Miyakawa Y, Mayumi M. Hepatitis G virus:a true hepatitis virus or an accidental tourist? N Engl J Med, 1997, 336(11):795
(1998-06-08收稿, 1998-07-13修回), 百拇医药
单位:
关键词:GBV-C/HGV基因组;全长cDNA;拼接;克隆
第二军医大学学报980401 述评 庚型肝炎病毒(HGV)自发现以来,虽然在世界各地均有自血清中用RT-PCR扩增出该病毒序列的报道,但对其是否可致肝炎尚有争论。本文利用自日本患者血清获得的克隆有HGV基因片段的质粒,通过基因片段的扩增及拼接,获得了全长9 373 bp的HGV cDNA。测核苷酸序列及推导的氨基酸顺序与原来的HGV-Iw同源性为99.71%及99.76%。这一全长cDNA如能证实具有一定的功能,将是利用细胞或动物进行功能性研究的主要工具,并可为阐明HGV是否有致病性提供重要的实验依据。
中国微生物学会理事长
卫生部医学分子病毒室主任
, 百拇医药
上海医科大学教授 闻玉梅
摘要 目的: 构建GB病毒C/庚型肝炎病毒(GBV-C/HGV)基因组全长cDNA克隆。方法: 以覆盖GBV-C/HGV分离株HGV-Iw全长基因组且互相重叠的5个基因片段Iw5,Iwq2,Iwh6,Iw3和Iw3为起始材料,采用重叠延伸PCR拼接和连接酶连接的方法,构建GBV-C/HGV基因组全长cDNA克隆。结果: GBV-C/HGV基因组全长cDNA克隆的酶切图谱与预期一致;核苷酸序列分析显示,克隆的GBV-C/HGV基因组全长cDNA的核苷酸及推测的氨基酸序列与原HGV-Iw序列一致。结论: GBV-C/HGV基因组全长cDNA的克隆已经构建成功。该克隆的构建,为深入研究GBV-C/HGV的致病性及致病机制、复制及转录和翻译机制、形态及形态发生等提供了重要的实验材料。
中国图书资料分类法分类号 Q781
Splicing and cloning of the full-length genomic cDNA of GB virus C/hepatitis G virus
, http://www.100md.com
Zhu Fenlu, Qi Zhongtian, Ren Hao, Song Yanbin, Shao Li (Department of Microbiology, Department of Basic Medicine, Second Military Medical University, Shanghai, 200433)
Abstract Objective: To construct GBV-C/HGV full-length genomic cDNA clone. Methods: On the basis of 5 cDNA fragments termed in order from 5 end to 3 end of the genome:Iw5,Iwq2,Iwh6,Iw3 and Iw3 of a GBV-C/HGV isolate HGV-Iw,the GBV-C/HGV full-length genomic cDNA clone was constructed using overlap extension PCR and ligation methods. Results: By digesting with the predicted endonucleases,the physical map of GBV-C/HGV full-length genomic cDNA clone was identical with the expected results. The determination of nucleotide sequence showed that the nucleotide and amino acid sequences of GBV-C/HGV full-length genomic cDNA was the same as original isolate HGV-Iw. Conclusion: GBV-C/HGV full-length genomic cDNA clone has been constructed successfully,and it will play an important role in further studing on the virus pathogenicity and pathogenesis,replication,transcription and translation machanism,virus morphology and morphogenesis.
, http://www.100md.com
Key words GBV-C/HGV genome; full-length cDNA; splicing; cloning
1995年,两个研究小组几乎同时分别获得了一种新型肝炎病毒的全序列,分别命名为GB病毒C(GBV-C)和庚型肝炎病毒(HGV)[1~3]。GBV-C和HGV的核苷酸及氨基酸同源性分别为85%和95%,因此认为它们是同一种病毒的两个不同分离株,称为GBV-C/HGV。GBV-C/HGV属黄病毒科,为单股正链RNA病毒,基因组长约9.4kb,从5至3端依次为5非编码区,结构基因区C,E1,E2,非结构基因区NS2,NS3,NS4,NS5a,NS5b及3非编码区。基因组仅含有一个开放阅读框, 编码一个约2 900个氨基酸的多聚蛋白质前体。
GBV-C/HGV基因组结构的研究进展很快,迄今已发表的GBV-C/HGV全序列有11株之多[2,3,4~8],但均为GBV-C/HGV基因片段的克隆,至今未见GBV-C/HGV基因组全长cDNA单个完整克隆的报道。GBV-C/HGV基因组全长cDNA单个完整克隆的缺乏,阻碍了对该病毒的深入研究。本研究在利用长PCR分段扩增和克隆GBV-C/HGV基因的基础上,采用重叠延伸PCR拼接和连接结合的方法,首次成功地克隆了GBV-C/HGV基因组全长cDNA。GBV-C/HGV基因组全长cDNA的克隆成功,对研究GBV-C/HGV基因组的结构和功能、GBV-C/HGV的致病性及致病机制、复制及转录和翻译机制、形态及形态发生等均具有重要的意义。
, 百拇医药
1 材料和方法
1.1 质粒和菌种 质粒Iw5,Iwq2,Iwh6,Iw3和Iw3是从一日本患者血清中扩增GBV-C/HGV基因片段并克隆至pT7 blue T-vector获得。5个质粒分别含有从5至3端覆盖GBV-C/HGV整个基因组、相邻片段互相重叠的5个GBV-C/HGV基因片段[5](图1)。克隆载体pUC18,pGEM-3Zf(+)购自华美生物工程公司,宿主菌JM109由本室保存。

图 1 GBV-C/HGV基因组结构和cDNA片段的位置
Fig 1 Structure of full length GBV-C/HGV genome and
, http://www.100md.com
the location of the cDNA fragments
nt: nucleotide
1.2 酶及试剂 所有的PCR反应均采用宝灵曼公司的ExpandTM Long Template PCR System,DNA聚合酶Ⅰ大片段(Klenow)、限制性内切酶和连接酶购自Biolab或Promega公司,普通试剂购自华美生物工程公司。
1.3 引物设计与合成 应用12条由Sangon生物工程公司合成的Y1~Y12引物进行GBV-C/HGV基因的扩增和拼接(表1)。在引物Y1和Y10的5端分别加上EcoRⅠ和NotⅠ位点及XhoⅠ和XbaⅠ位点,以便于进行克隆。
1.4 GBV-C/HGV基因片段的扩增、拼接及克隆 以质粒Iw5,Iwq2,Iwh6,Iw3和Iw3为模板,分别扩增GBV-C/HGV 5个同名基因片段。经凝胶电泳纯化,Klenow酶处理后,再进行拼接反应。经扩增和拼接可获得覆盖GBV-C/HGV整个基因组、且相邻片段互相重叠的3个基因片段。将它们分别克隆至pUC18和pGEM-3Zf(+)载体中,得到3个不同的重组质粒,再分别经适宜的酶切,回收GBV-C/HGV基因片段,与酶切后的pGEM-3Zf(+)载体连接,转化大肠杆菌,获得GBV-C/HGV全长cDNA克隆。
, 百拇医药
表 1 扩增及拼接GBV-C/ HGV基因片段的引物
Tab 1 Primers for amplifying and splicing GBV-C/HGV genes Primers
Sequences (5-3)
Location of primers in genome
Polarity
Y 1
GCG AAT TCG CGG CCG CCC CCC CCC CGG CAC TGG GTG CAA GC
1~26
+
, 百拇医药
Y 2
AGA GAG ACA TTG AAG GGC GAC
321~341
-
Y 3
GAC AGG GTT GGT AGG TCG TAA ATC C
109~133
+
Y 4
CCG TCC TTG ATG ATG GAA CTG TC
4 399~4 421
, 百拇医药
-
Y 5
TAT GGG CAT GGC ATA CCC CT
4 261~4 280
+
Y 6
GAG GGC CAC GAT GAT GTT AG
8 696~8 715
-
Y 7
TTC TGC TCC ACT TGG CTC GCT GAG T
, http://www.100md.com
8 416~8 440
+
Y 8
CTG GAT GCC ACA ACA GGC CCC T
8 881~8 902
-
Y 9
AGG GGC CTG TTG TGG CAT CCA G
8 881~8 902
+
Y10
GCT CTA GAC TCG AGA ATA AAA CCC GGC CTT TGG GCC G
, http://www.100md.com
9 353~9 375
-
Y11
TTG TTC GAT CAT ATG GGG TCC TTC TCG C
2 977~3 004
+
Y12
GAC CGG AGT CCC TTC AAG TAT CTC CTG G
7 737~7 764
-
1.5 GBV-C/HGV全长cDNA克隆的核苷酸序列测定 在初步的酶切鉴定正确之后,将GBV-C/HGV全长cDNA克隆分别从5和3端用Dye Terminator Cycles Sequencing方法进行核苷酸序列测定。利用PC/Gene软件进行序列比较分析。
, 百拇医药
2 结 果
2.1 GBV-C/HGV基因片段的扩增及拼接 以质粒Iw5,Iwq2,Iwh6,Iw3和Iw3为模板,分别对应引物Y1/Y2,Y3/Y4,Y5/Y6,Y7/Y8和Y9/Y10,扩增获得GBV-C/HGV 5个同名基因片段。推测基因片段的长度分别为:Iw5,358 bp;Iwq2,4 313 bp;Iwh6,4 455 bp;Iw3,487 bp;Iw3,509 bp。将PCR产物行1%琼脂糖凝胶电泳,所有基因片段均与预期的大小一致(图2中的第2,4,6,9,10泳带)。
GBV-C/HGV 5端基因片段的拼接以Iw5和Iwq2基因片段为模板,Y1和Y4为引物,其中Iw5基因片段的3端与Iwq2基因片段的5端重叠。经过重叠延伸PCR(SOEing and PCR)反应(PCR条件:94℃预变性2 min,然后94℃ 30s,60℃ 1 min,68℃ 6 min 35个循环,反应结束前延伸10 min),获得了长约4 437 bp的Iw5-q2基因片段(图2中的第3泳带)。依据同样的原理,以Iwq2和Iwh6基因片段为模板,Y11和Y12为引物进行拼接扩增,获得了长约4 788 bp的Iwq2-h6基因片段(图2中的第5泳带)。 3端基因片段的拼接是先以Iw3和Iw3基因片段为模板, Y7和Y10为引物, 拼接得到了长约974 bp的Iw3-3基因片段(图2中的第8泳

, 百拇医药
图 2 GBV-C/HGV cDNA片段的凝胶电泳分析
Fig 2 Gel electrophoresis analysis of
GBV-C/HGV cDNA fragments
1:λDNA/EcoRⅠ+ HindⅢ;2:Iw5;3:Iw5-q2;4:Iwq2;5:Iwq2-h6;
6:Iwh6;7:Iwh6-3; 8:Iw3-3;9:Iw3;10:Iw3

图 3 重组质粒 pUC-5-q2的构建
Fig 3 Construction of recombinant plasmid pUC-5-q2
, 百拇医药
带);再以Iw3-3片段与Iwh6基因片段为模板,Y5和Y10为引物进行拼接扩增,获得了长约5 129 bp的Iwh6-3基因片段(图2中的第7泳带)。
2.2 GBV-C/HGV基因片段及全长cDNA的克隆 将拼接扩增获得的GBV-C/HGV基因片段分别克隆至pUC18和pGEM-3Zf(+)载体。基因片段经凝胶电泳纯化后,Iw5-q2经EcoRⅠ+BamHⅠ酶切,回收约3 301 bp的基因片段,与同样经EcoRⅠ+BamHⅠ酶切的pUC18载体连接,获得重组质粒pUC-5-q2(图3)。该重组质粒经EcoRⅠ+BamHⅠ酶切,出现预期约3 301 bp的基因片段(图4中第2泳带)。Iwq2-h6基因片段经BamHⅠ+SalⅠ酶切,出现大小分别为308 bp, 1 181 bp和3 298 bp的3个基因片段,回收约3 298 bp的基因片段,克隆至pUC18的BamHⅠ和SalⅠ位点,得到重组质粒pUC-q2-h6。BamHⅠ+SalⅠ酶切鉴定发现该重组质粒切出预期约3 298 bp的基因片段(图4中的第4泳带)。Iwh6-3基因片段经SalⅠ和XbaⅠ酶切后回收约2 806 bp的基因片段, 与经过同种酶处理的pGEM-3Zf(+)载体连接、 转化, 转化子质粒经SalⅠ和XbaⅠ双酶切, 出现约2 806 bp的预期基因片段, 证实为重组质粒pGEM-h6-3(图4中的第6泳带)。
, 百拇医药
在完成3个基因片段的分段克隆后,将pUC-5-q2用EcoRⅠ和BamHⅠ酶切,回收约3 301 bp的基因片段;pUC-q2-h6经BamHⅠ和SalⅠ酶切,回

图 4 重组质粒 pUC-5-q2,pUC-q2-h6和
pGEM-h6-3的酶切电泳分析
Fig 4 Endonuclease digestion and gel electrophoresis
analysis of the recombinant plasmids pUC-5-q2,pUC-q2-h6 and pGEM-h6-3
1:λDNA/EcoRⅠ+ HindⅢ;2:pUC-5-q2/EcoRⅠ+ BamHⅠ;
, http://www.100md.com
3:pUC18/ EcoRⅠ+ BamHⅠ;4:pUC-q2-h6/BamHⅠ+ SalⅠ;
5:pUC18/ BamHⅠ+ SalⅠ; 6:pGEM-h6-3/SalⅠ+XbaⅠ;
7:pGEM-3Zf(+)/ SalⅠ+XbaⅠ

图 5 重组质粒 pHGVqz的构建
Fig 5 Construction of recombinant plasmid pHGVqz
收约3 298 bp的基因片段;pGEM-h6-3经SalⅠ和XbaⅠ酶切,回收约2 806 bp的基因片段。将pGEM-3Zf载体经EcoRⅠ和XbaⅠ酶切后回收,在同一连接系统中同时加入酶切后的pGEM-3Zf基因片段及3个GBV-C/HGV基因片段,经T4 DNA连接酶连接,转化大肠杆菌JM109,蓝白菌落筛选后挑取白色菌落进行酶切鉴定,得到GBV-C/HGV全长cDNA克隆pHGVqz(图5)。
, http://www.100md.com
2.3 GBV-C/HGV基因片段和全长cDNA克隆的酶切鉴定分析 根据HGV-Iw全序列的酶切位点分析,选取不同的内切酶分别对PCR扩增获得的5个GBV-C/HGV cDNA基因片段进行酶切鉴定,酶切电泳酶谱与预期结果完全一致(图略)。GBV-C/HGV基因组全长 cDNA克隆的鉴定,选用EcoRⅠ,NotⅠ,XbaⅠ,BamHⅠ,SalⅠ,PstⅠ和HindⅢ等内切酶进行鉴定。预期酶切结果:EcoRⅠ酶切,重组质粒被切为5 767 bp和6 836 bp的两个基因片段,在1%的琼脂糖凝胶电泳时,出现一条带;HindⅢ酶切,出现约为2 260 bp和10 343 bp的两条带;PstⅠ酶切出现约为2 577 bp, 3 671 bp和6 405 bp的3条带;SalⅠ酶切应出现约为2 821 bp和9 782 bp的两条带。双酶切结果:BamHⅠ+XbaⅠ,重组质粒被切为6 119 bp和6 484 bp的两个基因片段,在1%琼脂糖凝胶电泳时呈现一条带;EcoRⅠ+XbaⅠ酶切出现约为2 568 bp, 3 199 bp和6 836 bp的3条带;NotⅠ+XbaⅠ酶切出现约为9 404 bp

, http://www.100md.com
图 6 GBV-C/HGV基因组全长cDNA 克隆
的酶切图谱分析
Fig 6 Physical mapping analysis of GBV-C/HGV
full-length genomic cDNA clone digested by
the predicted endonucleases
1:λDNA/EcoRⅠ+ HindⅢ;2:pHGVqz/EcoRⅠ;3:pHGVqz/
HindⅢ; 4:pHGVqz/PstⅠ;5:pHGVqz/SalⅠ;6:pHGVqz/
BamHⅠ+ XbaⅠ; 7:pHGVqz/EcoRⅠ+XbaⅠ;8:pHGVqz/
, 百拇医药
NotⅠ+XbaⅠ; 9:pGEM-3Zf(+)/ EcoRⅠ+XbaⅠ
的GBV-C/HGV全长cDNA基因片段和约3 199 bp的pGEM-3Zf载体基因片段两条带。GBV-C/HGV基因组全长cDNA克隆的酶切图谱见图6。
2.4 GBV-C/HGV全长cDNA克隆的核苷酸序列测定 核苷酸序列测定结果显示,克隆的GBV-C/HGV的基因组全长cDNA长9 373 bp,推测编码氨基酸2 873个(序列略)。将克隆的GBV-C/HGV cDNA核苷酸序列及推测氨基酸序列与GBV-C[2],HGV[3],HGV1[3],HGVC964[4]和HGV-Iw[5]的相应序列进行了同源性比较(表2)。
表 2 GBV-C/HGV cDNA核苷酸及氨基酸序列同源性比较
, http://www.100md.com
Tab 2 Nucleotide and amino acid identity of GBV-C/HGV cDNA Compared
sequences
Accession No.
Nucleotide
identity (%)
Amino acid
identity (%)
GBV-C
U36380
86.90
, 百拇医药
96.03
HGV
U44402
93.27
98.33
HGV1
U45966
90.84
97.98
HGVC964
U75356
88.98
95.20
, http://www.100md.com
HGV-Iw
D87255
99.71
99.76
同源性比较结果证实我们已成功地获得了GBV-C/HGV的基因组全长cDNA克隆。
3 讨 论
3.1 HGV-Iw基因片段的拼接 迄今虽已报道了11株GBV-C/HGV全序列, 但均不是GBV-C/HGV基因组全长cDNA单个完整克隆。 本研究中作为实验基础的5个重组质粒克隆的GBV-C/HGV 5个同名基因片段, 从5至3端相邻片段互相重叠且覆盖长9 375 bp的HGV-Iw基因组[5], 其中Iwq2和Iwh6片段是应用长PCR方法获得的, 长度分别为4 313 bp和4 455 bp, 使我们避免了从数个几百个碱基的小基因片段起始的繁杂过程。
, 百拇医药
由重叠延伸PCR反应进行基因拼接是以PCR方法为基础, 先进行重叠延伸反应产生重组DNA分子, 再经PCR进行指数扩增的一种构建新的DNA 分子的技术方法[9]。 该方法具有无需限制性内切酶和连接酶、不管拼接位点在核苷酸序列的什么位置均可准确拼接的优点。 因该方法是以PCR反应为基础的, 所以PCR反应中由DNA聚合酶产生的错误掺入的缺陷在进行基因拼接时同样存在。 本实验中我们选用ExpendTM Long Template PCR System, 其DNA聚合酶混合物中的Pwo DNA 聚合酶具有校正阅读功能, 从而显著降低了扩增产物的错误频率。 此外, 由重叠延伸PCR拼接基因片段, 其预拼接的基因长度有一定限制, 较短的基因片段易于拼接和扩增。 本研究中拼接基因的长度约1~5 kb, 拼接结果良好。 但在初期实验直接经重叠延伸PCR拼接全长cDNA, 虽然也获得了预期全长cDNA约9.4 kb的基因片段, 但其扩增效率较差, 致使无法直接从约9.4kb的基因片段完成全长cDNA的克隆。
, 百拇医药
3.2 GBV-C/HGV全长cDNA基因的克隆策略 如上所述, 由于经直接拼接无法获取足量的用于克隆的全长cDNA基因, 因此改为采用先分段克隆GBV-C/HGV基因片段, 再克隆全长cDNA基因的方法。
在完成5端Iw5-q2和3端h6-3基因片段的拼接之后, 又设计引物, 拼接扩增了Iwq2-h6基因片段。 在设计引物Y11和Y12时, 既要考虑使Iwq2-h6基因片段5端与Iw5-q2基因片段的3端重叠基因部分包括BamHⅠ位点在内, 3端与Iwh6-3基因片段重叠部分包括SalⅠ位点;又要考虑在Iwq2-h6基因片段经BamHⅠ+SalⅠ酶切之后, 其长约3 298 bp的片段能顺利回收, 便于操作。 在分段克隆GBV-C/HGV基因片段时, 用pUC18载体克隆Iw-q2和Iw-q2-h6基因片段, 用pGEM-3Zf载体克隆Iwh6-3基因片段, 主要是从待克隆的基因片段与载体的大小相比便于进行基因操作考虑。 在完成GBV-C/HGV基因片段的分段克隆后, 将3个重组质粒分别用不同的酶切, 回收GBV-C/HGV基因片段及酶切后的载体片段, 经T4 DNA连接酶连接, 转化大肠杆菌JM109, 最后获得了GBV-C/HGV基因组全长cDNA克隆。
, http://www.100md.com
3.3 构建GBV-C/HGV基因组全长cDNA克隆的意义 对于一种新病毒, 最令人关注的是病毒的致病性。 GBV-C/HGV是否导致人类肝炎, 至今尚无明确定论。 初期研究结果显示,GBV-C/HGV与输血后肝炎及其他急、慢性肝病有关[1,3]。 近期的研究表明, GBV-C/HGV不能引起肝炎或导致的肝炎症状轻微, 对共感染的HBV, HCV所引起的肝病症状没有影响[4,10,11]。 但多数研究者同时也发现, GBV-C/HGV可经血传播, 在人群中有较高的感染率, 可引起长期持续的感染。 因此, 迫切需要对GBV-C/HGV进行广泛而深入的研究。 但GBV-C/HGV基因组全长cDNA克隆的缺乏, 阻碍了研究的进行。 这次GBV-C/HGV基因组全长cDNA的克隆成功, 为研究GBV-C/HGV的致病性及致病机制, GBV-C/HGV的复制、转录和翻译机制, GBV-C/HGV的形态及形态发生, GBV-C/HGV和HCV的比较研究等许多方面提供了重要的实验材料。
, 百拇医药 致谢: 在本研究工作中, 本校长征医院缪晓辉副教授给予了许多有益的启示和真诚的帮助;长海医院何建文博士对实验设计提出了宝贵的建议, 在此一并表示深深的谢意!
参 考 文 献
1 Simons JN, Leary TP, Dawson GJ, et al. Isolation of novel virus-like sequences associated with human hepatitis. Nat Med, 1995, 1(6):564
2 Leary TP, Muerhoff SM, Simons JN, et al. Sequence and genomic organization of GBV-C:a novel member of the Flaviviridae associated with human non-A-E hepatitis. J Med Virol, 1996, 48(1):60
, http://www.100md.com
3 Linnen J, Wages J Jr, Zhang-Keck ZY, et al. Molecular cloning and disease association of hepatitis G virus:a transfusion-transmissible agent. Science, 1996, 271(5248):505
4 周育森, 陈 微, 赵秋敏, 等. 中国人庚型肝炎病毒全基因cDNA克隆及序列测定.军事医学科学院院刊, 1996, 20(4):249
5 Shao L, Shinzawa H, Ishikawa K, et al. Sequence of hepatitis G virus genome isolated from a Japanese patient with non-A-E hepatitis:amplification and cloning by long reverse transcription-PCR. Biochem Biophys Res Commun, 1996, 228(3):785
, http://www.100md.com
6 Erker JC, Simons JN, Moerhoff AS, et al. Molecular cloning and characterization of a GB virus C isolate from a patient with non-A-E hepatitis. J Gen Virol, 1996, 77(Pt:11):2713
7 Okamoto H, Nakao H, Inoue T, et al. The entire nucleotide sequence of two GB virus C/hepatitis G virus isolates of distinct genotypes from Japan. J Gen Virol, 1997, 78(Pt:4): 737
8 Nakao H, Okamoto H, Fukuda M, et al. Mutation rate of GB virus C/hepatitis G virus over the entire genome and in subgenomic regions. Virology, 1997, 233(1):43
, http://www.100md.com
9 Horton RM, Hunt HD, Ho SN, et al. Engineering hybrid genes without the use of restriction enzymes:gene splicing by overlap extension. Gene , 1989, 77(1):61
10 Alter HJ. The cloning and clinical implications of HGV and HBGV-C. N Engl J Med, 1997, 334(23):1536
11 Miyakawa Y, Mayumi M. Hepatitis G virus:a true hepatitis virus or an accidental tourist? N Engl J Med, 1997, 336(11):795
(1998-06-08收稿, 1998-07-13修回), 百拇医药